L'emulsione con modellazione di particelle consente saggi di goccioline prive di microfluidi
Summary
I saggi di goccioline acqua-in-olio sono utili per la chimica analitica, l’evoluzione enzimatica e l’analisi di singole cellule, ma in genere richiedono microfluidica per formare le goccioline. Qui, descriviamo l’emulsificazione modellata con particelle, un approccio privo di microfluidi per eseguire saggi di goccioline.
Abstract
Le reazioni eseguite in goccioline monodisperse offrono una maggiore precisione e sensibilità rispetto a quelle equivalenti eseguite alla rinfusa. Tuttavia, il requisito della microfluidica di formare goccioline controllate impone una barriera ai non esperti, limitandone l’uso. Qui, descriviamo l’emulsione modellata con particelle, un approccio per generare goccioline monodisperse senza microfluidica. Utilizzando sfere di idrogel di template, incapsulamo campioni in goccioline monodisperse mediante semplice vortice. Dimostriamo l’approccio utilizzandolo per eseguire la PCR digitale senza microfluidi.
Introduction
La microfluidica delle goccioline sfrutta la compartimentazione nelle goccioline di picoliter per aumentare la sensibilità e l’accuratezza dei saggi rispetto alle reazioni di massa e ha numerose applicazioni nello screening chimico, nell’ingegneria proteica e nel sequenziamento di prossima generazione1,2,3. Ad esempio, la reazione a catena digitale della polimerasi a goccia (ddPCR) offre una maggiore precisione rispetto alla reazione a catena quantitativa della polimerasi di massa (qPCR), con applicazioni per la variazione genetica nei tumori, il rilevamento di mutazioni che causano malattie e la diagnostica prenatale4,5,6. Una sfida della microfluidica delle goccioline, tuttavia, è il requisito dei dispositivi microfluidici per partizionare i campioni; mentre la microfluidica offre un eccellente controllo sulle proprietà delle goccioline, richiede competenze specializzate per costruire e operare7,8. Di conseguenza, i metodi basati su goccioline sono in gran parte limitati a laboratori esperti o, in rari casi, applicazioni in cui è disponibile uno strumento commerciale9,10. Per ampliare l’uso dei saggi di goccioline, il requisito di strumentazione microfluidica specializzata è un ostacolo che deve essere superato.
In questo articolo, descriviamo l’emulsificazione delle particelle modellate (PTE), un metodo privo di microfluidi per eseguire reazioni in goccioline monodisperse. Nel PTE, le particelle di template inghiottono il campione in goccioline nell’olio vettore mediante semplice vortice (Figura 1). Mentre il sistema si mescola, la porzione acquosa si frammenta in goccioline di dimensioni ridotte fino a quando le goccioline contengono singole particelle, a quel punto un’ulteriore frammentazione non è possibile perché richiede la rottura delle particelle. Il campione inghiottito circonda le particelle come un guscio nelle goccioline, incapsulando così eventuali cellule disperse, reagenti o porzioni funzionali (Figura 1D). Pertanto, pte non richiede attrezzature o competenze per eseguire reazioni di goccioline oltre un comune vortice. Inoltre, la generazione di goccioline richiede secondi rispetto a minuti o ore con la microfluidica e la quantità prodotta è proporzionale al volume del contenitore, non al tempo di funzionamento del dispositivo, rendendolo estremamente scalabile. Questi vantaggi rendono il PTE ideale per condurre saggi di goccioline in una varietà di circostanze in cui la microfluidica non è pratica. Qui, dimostriamo PTE e lo usiamo per condurre ddPCR.
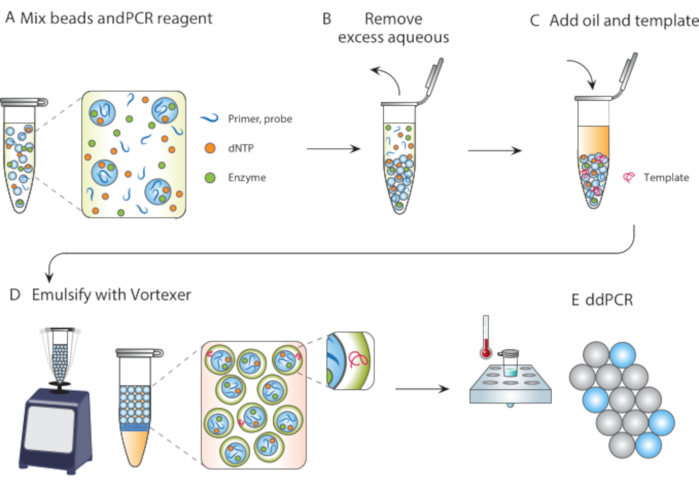
Figura 1. Panoramica del processo di emulsione con modellazione di particelle. (A) Le particelle di template sono mescolate con reagenti. (B) I reagenti in eccesso vengono rimossi dopo la centrifugazione. (C) L’aggiunta di molecole modello avviene prima dell’aggiunta di olio. (D) Il vortice produce goccioline contenenti una singola molecola modello. (E) Il successivo termociclo e l’imaging consentono l’analisi digitale delle goccioline del modello target. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
PTE utilizza particelle per incapsulare campioni in goccioline monodisperse mediante vortice. Oltre alla sua semplicità e accessibilità, PTE offre diversi vantaggi aggiuntivi, tra cui la possibilità di generare istantaneamente grandi volumi di goccioline. Inoltre, il processo può essere condotto in un tubo isolato, ovviando alla necessità di trasferire campioni a dispositivi microfluidici, semplificando il flusso di lavoro complessivo e limitando le opportunità di contaminazione o perdita del campione. Le particelle di template forniscono anche un mezzo con cui progettare il contenuto delle reazioni di goccioline risultanti. Ad esempio, la dimensione delle particelle, la chimica e la bagnabilità possono essere progettate per la biomolecola mirata o la cattura cellulare, mentre le parti funzionali come enzimi, attivi o acidi nucleici possono essere visualizzate sulla particella per facilitare le reazioni, ad esempio per il sequenziamento di singole cellule o la caratterizzazione funzionale. Sebbene l’approccio sia flessibile, vi sono tuttavia importanti vincoli al suo utilizzo. Ad esempio, non è attualmente possibile eseguire aggiunte di goccioline come spesso vengono condotte con la microfluidica, richiedendo che tutti i componenti della reazione siano introdotti prima dell’incapsulamento; ciò richiede che i reagenti siano compatibili e stabili fino a quando le goccioline possono essere generate e, nel caso di combinazioni fastidiose, possono spesso essere affrontate mescolando ed emulsionando rapidamente il campione su ghiaccio. In alternativa, è possibile utilizzare componenti reattivi che possono essere attivati esternamente con luce o calore13. PTE fornisce quindi un metodo flessibile e scalabile per condurre saggi di goccioline accessibili ai non esperti. Questo, unito alla sua innata semplicità e flessibilità, rende PTE ideale per l’esecuzione e lo sviluppo di numerose applicazioni droplet.
Divulgations
The authors have nothing to disclose.
Acknowledgements
Questo lavoro di sviluppo di questo protocollo è stato supportato dal National Institutes of Health (R01-EB019453-02), dall’Office of the Director of National Intelligence, dall’Intelligence Advanced Research Projects Activity attraverso Raytheon BBN Technologies Corp (N66001-18-C-4507), dal Chan-Zuckerberg Biohub Investigator Program, dalla Defense Advanced Research Projects Agency attraverso la Texas A & M University (W911NF1920013) e dai Centers for Disease Control and Prevention attraverso la Johns Hopkins University Applied Laboratorio di Fisica (75D30-11-9C-06818 (CDC3)). Le opinioni e le conclusioni contenute nel presente documento sono quelle degli autori e non devono essere interpretate come necessariamente rappresentative delle politiche ufficiali, espresse o implicite, delle organizzazioni di cui sopra o del governo degli Stati Uniti. Il governo degli Stati Uniti è autorizzato a riprodurre e distribuire ristampe per scopi governativi, nonostante qualsiasi annotazione di copyright in esse contenuta.
Materials
| 0.22 um syringe filter | Milipore Sigma | SLGP033RS | |
| 0.5M EDTA, pH 8.0 | Thermo-Fisher | 15575020 | |
| 0.75 mm biopsy punch | World Precision Instruments | 504529 | |
| 1 mL syringes | BD | 309628 | |
| 1H,1H,2H-Perfluoro-1-Octanol (PFO) | Sigma-Aldrich | 370533 | |
| 1M Tris-HCI, pH 8.0 | Thermo-Fisher | 15568025 | |
| 27 gauge needles | BD | 305109 | |
| 3" silicon wafers, P type, virgin test grade | University Wafers | 447 | |
| 3D-printed centrifuge syringe holder | (custom) | (custom) | |
| Acrylamide solution,40%, for electrophoresis, sterile-filtered | Sigma-Aldrich | A4058-100ML | |
| Ammonium persulfate | Sigma-Aldrich | A3678-25G | |
| Aquapel (fluorinated surface treatment) | Pittsburgh Glass Works | 47100 | |
| Hexane | Sigma-Aldrich | 139386 | |
| FC-40 fluorinated oil | Sigma-Aldrich | F9755 | |
| Isopropanol | Sigma-Aldrich | 109827 | |
| N,N′-Methylenebis(acrylamide) | Sigma-Aldrich | 146072-100G | |
| NaCl | Sigma-Aldrich | S9888 | |
| Novec-7500 Engineering Fluid (HFE oil) | 3M | 98-0212-2928-5 | |
| polyethylene tubing | Scientific Commodities | B31695-PE/2 | |
| fluorosurfactant | Ran Biotechnologies | 008-FluoroSurfactant | |
| PGMEA developer | Sigma-Aldrich | 484431 | |
| Photomasks | CadArt Servcies | (custom) | |
| Platinum Multiplex PCR Master Mix (Taq Master Mix) | Applied Biosystems | 4464263 | |
| Spin coater | Specialty Coating Systems | G3P-8 | |
| Span 80 (sorbitane monooleate) | Sigma-Aldrich | s6760 | |
| SU-8 3025 photoresist | Kayaku | 17030192 | |
| Triton X-100 (octylphenol ethoxylate) | Sigma-Aldrich | t8787 | |
| Tween 20 (polysorbate 20) | Sigma-Aldrich | p2287 | |
| Platinum Multiplex PCR Master Mix (Taq Master Mix) | Applied Biosystems | 4464263 | |
| Yeast FWD | IDT | 5′-GCAGACCAGACCAGAACAAA-3′ | |
| Yeast REV | IDT | 5′-ACACGTATGTATCTAGCCGAATA AC-3 |
|
| Yeast Probe | IDT | 5′-/56-FAM/ATATGTTGT/ZEN/TCACTCGCGCCTGGG/3IABk FQ/-3′ |
|
| EVOS FL AUTO | Life Technologies | ||
| EVOS LED Cube, GFP | Life Technologies | AMEP4651 | |
| SYLGARD 184 KIT 1.1 LB (PDMS base and curing reagents) | Dow Corning | DC4019862 | |
| TEMED | Thermo Fisher | 17919 | |
| Saccharomyces cerevisiae genomic DNA | Milipore | 69240-3 | |
| Expanded plasma cleaner (plasma bonder) | Harrick Plasma | PDC-002 (230V) | |
References
- Mashaghi, S., Abbaspourrad, A., Weitz, D. A., van Oijen, A. M. Droplet microfluidics: A tool for biology, chemistry, and nanotechnology. Trends in Analytical Chemistry. 82, 118-125 (2016).
- Gielen, F., et al. Ultrahigh-throughput-directed enzyme evolution by absorbance-activated droplet sorting (AADS). Proceedings of the National Academy of Sciences of USA. 113 (47), 7383-7389 (2016).
- Mai, S., Murphy, T. W., Lu, C. Microfluidics for genome-wide studies involving next generation sequencing. Biomicrofluidics. 11 (2), 021501 (2017).
- Olmedillas-López, S., García-Arranz, M., García-Olmo, D. Current and emerging Applications of Droplet Digital PCR in Oncology. Molecular Diagnosis and Therapy. 21 (5), 493-510 (2017).
- Tong, Y., Shen, S., Jiang, H., Chen, Z. Application of Digital PCR in Detecting Human Diseases Associated Gene Mutation. Cellular Physiology and Biochemistry. 43 (3), 1718-1730 (2017).
- Yan, Y., et al. Evaluation of droplet digital PCR for non-invasive prenatal diagnosis of phenylketonuria. Analytical and Bioanalytical Chemistry. 411 (27), 7115-7126 (2019).
- Shang, L., Cheng, Y., Zhao, Y. Emerging Droplet Microfluidics. Chemical Reviews. 117 (12), 7964-8040 (2017).
- The, S., Lin, R., Hung, L., Lee, A. P. Droplet Microfluidics. Lab Chip. 8 (2), 198-220 (2008).
- Baker, M. Digital PCR hits its stride. Nature Methods. 9, 541-544 (2012).
- Kolodziejczyk, A. A., Kim, J. K., Svensson, V., Marioni, J. C., Teichmann, S. A. The Technology and Biology of Single-Cell RNA Sequencing. Molecular Cell. 58 (4), 610-620 (2015).
- Hatori, M. N., Kim, S. C., Abate, A. R. Particle-Templated Emulsification for Microfluidics-Free Digital Biology. Analytical Chemistry. 90 (16), 9813-9820 (2018).
- Panda, P., et al. Stop-flow lithography to generate cell-laden microgel particles. Lab Chip. 8 (7), 1056-1061 (2008).
- Yozwiak, C. E., Hirschhorn, T., Stockwell, B. R. Towards a microparticle-based system for pooled assays of small molecules in cellular contexts. ACS Chemical Biology. 13 (3), 761-771 (2018).

