Идентификация связывающих белков малых лигандов с дифференциальным радиальным капиллярным действием лигандного анализа (DRaCALA)
Summary
Дифференциальное радиальное капиллярное действие лигандного анализа (DRaCALA) может быть использовано для идентификации малых лигандсвязывающих белков организма с помощью библиотеки ORFeome.
Abstract
За последнее десятилетие был достигнут огромный прогресс в понимании малых сигнальных молекул в бактериальной физиологии. В частности, целевые белки нескольких вторичных мессенджеров (NSM), полученных из нуклеотидов, были систематически идентифицированы и изучены в модельных организмах. Эти достижения в основном связаны с разработкой нескольких новых методов, включая метод захвата соединений и дифференциальное радиальное капиллярное действие лигандного анализа (DRaCALA), которые использовались для систематической идентификации целевых белков этих малых молекул. В данной работе описывается использование NSM, гуанозин пента- и тетрафосфатов (p)ppGpp, в качестве примера и видеодемонстрации метода DRaCALA. Используя DRaCALA, 9 из 20 известных и 12 новых целевых белков (p)ppGpp были идентифицированы в модельном организме Escherichia coli K-12, демонстрируя силу этого анализа. В принципе, DRaCALA может быть использован для изучения небольших лигандов, которые могут быть помечены радиоактивными изотопами или флуоресцентными красителями. Здесь обсуждаются критические шаги, плюсы и минусы DRaCALA для дальнейшего применения этой техники.
Introduction
Бактерии используют несколько небольших сигнальных молекул для адаптации к постоянно меняющимся средам1,2. Например, аутоиндукруторы, N-ацилгомосериновые лактоны и их модифицированные олигопептиды, опосредовывают межклеточную связь между бактериями для координации поведения популяции, явление, известное как ощущение кворума2. Другой группой малых сигнальных молекул является NSM, включая широко изученный циклический аденозинмонофосфат (цАМФ), циклический ди-АМФ, циклический ди-гуанозинмонофосфат (циклический ди-GMP) и гуанозин пента- и тетрафосфаты (p)ppGpp1. Бактерии производят эти NSM в ответ на множество различных стрессовых состояний. После производства эти молекулы связываются со своими белками-мишенями и регулируют несколько различных физиологических и метаболических путей, чтобы справиться с возникающими стрессами и повысить выживаемость бактерий. Поэтому идентификация белков-мишеней является неизбежной предпосылкой для расшифровки молекулярных функций этих малых молекул.
Последнее десятилетие стало свидетелем бума знаний об этих небольших сигнальных молекулах, в основном благодаря нескольким техническим инновациям, которые раскрыли целевые белки этих малых молекул. К ним относятся метод захвата соединения3,4,5и дифференциальное радиальное капиллярное действие лигандного анализа (DRaCALA)6, которые будут обсуждаться в этой статье.
Изобретенный Винсентом Ли и его коллегами в 2011году 6,DRaCALA использует способность нитроцеллюлозной мембраны дифференциально секвестрировать свободные и связанные с белком лиганды. Молекулы, такие как белки, не могут диффундировать на нитроцеллюлозной мембране, в то время как небольшие лиганды, такие как NSM, способны. Смешивая NSM(например,ppGpp) с тестируемым белком и обличивая их на мембране, можно ожидать двух сценариев(рисунок 1):Если (p)ppGpp связывается с белком, радиомаркированный (p)ppGpp будет удерживаться в центре пятна белком и не будет диффундировать наружу, давая интенсивную маленькую точку(т.е., сильный радиоактивный сигнал) под фосфоритатором. Однако, если (p)ppGpp не связывается с белком, он будет свободно диффундировать наружу, производя большое пятно с однородным фоновым радиоактивным сигналом.
Кроме того, DRaCALA может обнаружить взаимодействие между небольшой молекулой и неочищенным белком в цельном клеточном лизате, если белок присутствует в достаточном количестве. Эта простота позволяет использовать DRaCALA для быстрой идентификации белковых мишеней с помощью библиотеки экспрессии ORFeome. Действительно, целевые белки цАМФ7,циклический ди-АМФ8,циклический ди-GMP9,10и (p)ppGpp11,12,13 были систематически идентифицированы с помощью DRaCALA. В этой видеостатье используется (p)ppGpp в качестве примера для демонстрации и описания критических шагов и соображений при успешном проведении скрининга DRaCALA. Следует отметить, что более подробное описание DRaCALA14 настоятельно рекомендуется прочитать в сочетании с этой статьей перед выполнением DRaCALA.
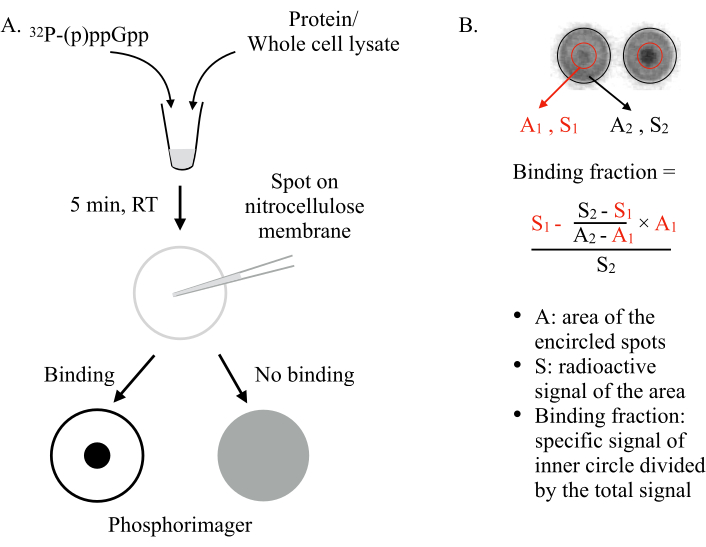
Рисунок 1:Принцип DRaCALA. (A) Схема анализа DRaCALA. Подробности см. в тексте. (B)Количественная оценка и расчет фракции связывания. Подробности см. в тексте. Вкратце, пятна DRaCALA будут проанализированы путем рисования двух кругов, которые ограничивают все пятно и внутреннюю темную точку(т. Е.Сохраненный (p)ppGpp из-за связывания тестируемого белка). Специфическим связывающим сигналом является радиоактивный сигнал внутреннего круга (S1) после вычитания неспецифического фонового сигнала (вычисляется по A1 × ((S2-S1)/(A2-A1))). Фракция связывания представляет специфический сигнал связывания, деленный на общий радиоактивный сигнал (S2). Сокращения: DRaCALA = дифференциальное радиальное капиллярное действие лигандного анализа; (p)ppGpp = гуанозин пента- и тетрафосфаты; RT = комнатная температура. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
Одним из важнейших шагов в выполнении скрининга DRaCALA является получение хороших целоклеточных лизатов. Во-первых, испытуемые белки должны вырабатываться в больших количествах и в растворимых формах. Во-вторых, лизис клеток должен быть полным, а вязкость лизата должна быть минимальной. …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Работа поддерживается грантом проекта NNF (NNF19OC0058331) для YEZ и исследовательской и инновационной программой Европейского Союза Horizon 2020 в рамках грантового соглашения Марии Склодовской-Кюри (Nº 801199) для MLS.

Materials
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID – Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen – Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 – Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID – Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution – 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
References
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry–a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ‘,5 ‘-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).

