लिगांड परख (DRaCALA) के अंतर रेडियल केशिलरी एक्शन के साथ छोटे लिगांड के बाध्यकारी प्रोटीन की पहचान करना
Summary
लिगांड परख (DRaCALA) के अंतर रेडियल केशिलरी एक्शन का उपयोग ओआरफेम लाइब्रेरी का उपयोग करके किसी जीव के छोटे लिगांड बाध्यकारी प्रोटीन की पहचान करने के लिए किया जा सकता है।
Abstract
पिछले दशक में बैक्टीरियल फिजियोलॉजी में छोटे सिग्नलिंग अणुओं की समझ में जबरदस्त प्रगति देखी गई है । विशेष रूप से, कई न्यूक्लियोटाइड-व्युत्पन्न माध्यमिक दूतों (एनएसएम) के लक्षित प्रोटीन की व्यवस्थित रूप से पहचान की गई है और मॉडल जीवों में अध्ययन किया गया है। ये उपलब्धियां मुख्य रूप से कैप्चर कंपाउंड तकनीक और लिगांड परख (DRaCALA) की अंतर रेडियल केशिका कार्रवाई सहित कई नई तकनीकों के विकास के कारण हैं, जिनका उपयोग इन छोटे अणुओं के लक्षित प्रोटीन की व्यवस्थित रूप से पहचान करने के लिए किया जाता था। यह पत्र DRaCALA तकनीक के उदाहरण और वीडियो प्रदर्शन के रूप में एनएसएम, ग्वानोसाइन पेंटा-और टेट्राफॉस्फेट (पी) पीपीजीपीपी के उपयोग का वर्णन करता है। DRaCALA का उपयोग करना, 20 ज्ञात में से 9 ज्ञात और (पी) पीपीजीपीपी के 12 नए लक्ष्य प्रोटीन मॉडल जीव, एस्चेरिचिया कोलाई K-12 में पहचाने गए थे, जो इस परख की शक्ति का प्रदर्शन करते हैं। सिद्धांत रूप में, DRaCALA का उपयोग छोटे लिगामेंट्स का अध्ययन करने के लिए किया जा सकता है जिन्हें रेडियोधर्मी आइसोटोप या फ्लोरोसेंट रंगों द्वारा लेबल किया जा सकता है। इस तकनीक के आगे आवेदन के लिए DRaCALA के महत्वपूर्ण कदम, पेशेवरों और विपक्ष पर चर्चा की जाती है।
Introduction
बैक्टीरिया लगातार बदलते वातावरण के अनुकूल होने के लिए कई छोटे संकेत अणुओं का उपयोग करते हैं1,2. उदाहरण के लिए, ऑटोइंडर्स, एन-एकिलहोमोसेरीन लैक्टोन और उनके संशोधित ओलिगोपेप्टाइड्स, जनसंख्या व्यवहार को समन्वित करने के लिए बैक्टीरिया के बीच अंतरकोशिकीय संचार में मध्यस्थता करते हैं, एक घटना जिसे कोरम संवेदन2के रूप में जाना जाता है। छोटे सिग्नलिंग अणुओं का एक अन्य समूह एनएसएम है, जिसमें व्यापक रूप से अध्ययन किया गया साइक्लिक एडेनोसाइन मोनोफोस्फेट (सीएएमपी), चक्रीय डी-एएमपी, साइक्लिक डी-ग्वानोसाइन मोनोफॉस्फेट (चक्रीय डी-जीएमपी), और ग्वानोसाइन पेंटा-और टेट्रा फॉस्फेट (पी) पीपीजीपी1शामिल हैं। बैक्टीरिया विभिन्न तनाव की स्थिति की एक प्रतिक्रिया के रूप में इन NSMs का उत्पादन। एक बार उत्पादित होने के बाद, ये अणु अपने लक्षित प्रोटीन से बांधते हैं और सामने आए तनावों से निपटने और बैक्टीरियल अस्तित्व को बढ़ाने के लिए कई अलग-अलग शारीरिक और मेटाबोलिक रास्तों को विनियमित करते हैं। इसलिए, इन छोटे अणुओं के आणविक कार्यों को समझने के लिए लक्षित प्रोटीन की पहचान एक अपरिहार्य शर्त है ।
पिछले दशक में इन छोटे संकेत अणुओं के ज्ञान का बूम देखा गया है, मुख्य रूप से कई तकनीकी नवाचारों के कारण है कि इन छोटे अणुओं के लक्ष्य प्रोटीन का अनावरण किया । इनमें कैप्चर कंपाउंड तकनीक3,4,5,और लिगांड परख (DRaCALA) 6 की अंतर रेडियल केशिका कार्रवाईशामिल है जो इस पेपर में चर्चा की जानी है।
20116में विंसेंट ली और सह-कार्यकर्ताओं द्वारा आविष्कार किया गया, DRaCALA एक नाइट्रोसेल्यूलोस झिल्ली की क्षमता को अलग-अलग तनहा मुक्त और प्रोटीन-बाध्य लिगांड के लिए तैनात करता है। प्रोटीन जैसे अणु नाइट्रोसेल्यूलोज झिल्ली पर फैला नहीं सकते, जबकि एनएसएम जैसे छोटे लिगामेंट्स कर सकते हैं । एनएसएम(जैसे,पीपीजीपी) को प्रोटीन के साथ मिलाकर, दो परिदृश्यों की उम्मीद की जा सकती है(चित्र 1): यदि(पी) पीपीजीपी प्रोटीन से बांधता है, तो रेडियोलेबल (पी) पीपीजीपी को प्रोटीन द्वारा स्थान के केंद्र में रखा जाएगा और बाहरी रूप से फैलाना नहीं होगा, जिससे एक तीव्र छोटी डॉट(यानी., एक फॉस्फोरमगर के नीचे मजबूत रेडियोधर्मी संकेत) । हालांकि, अगर (पी) ppGpp प्रोटीन के लिए बाध्य नहीं करता है, यह स्वतंत्र रूप से जावक फैलाना करने के लिए एक समान पृष्ठभूमि रेडियोधर्मी संकेत के साथ एक बड़े स्थान का उत्पादन होगा ।
इसके अलावा, DRaCALA एक छोटे से अणु और एक पूरी कोशिका में एक अशुद्ध प्रोटीन के बीच बातचीत का पता लगा सकते है अगर प्रोटीन एक पर्याप्त मात्रा में मौजूद है । यह सादगी एक ORFeome अभिव्यक्ति पुस्तकालय का उपयोग करके तेजी से प्रोटीन लक्ष्यों की पहचान करने में DRaCALA के उपयोग की अनुमति देता है । दरअसल, DRACALA का उपयोग करके सीएएएमपी7,चक्रीय डी-एएमपी8,चक्रीय दी-जीएमपी9,10,और (पी) पीपीजीपी11,12,13 के लक्षित प्रोटीन को व्यवस्थित रूप से पहचाना गया है। यह वीडियो लेख एक सफल DRaCALA स्क्रीनिंग प्रदर्शन में महत्वपूर्ण कदमों और विचारों को प्रदर्शित करने और वर्णन करने के लिए एक उदाहरण के रूप में (पी) पीपीजीपीपी का उपयोग करता है। ध्यान दें, DRaCALA14 का अधिक गहन विवरण DRaCALA प्रदर्शन करने से पहले इस लेख के साथ संयोजन में पढ़ने की अत्यधिक सिफारिश की जाती है।
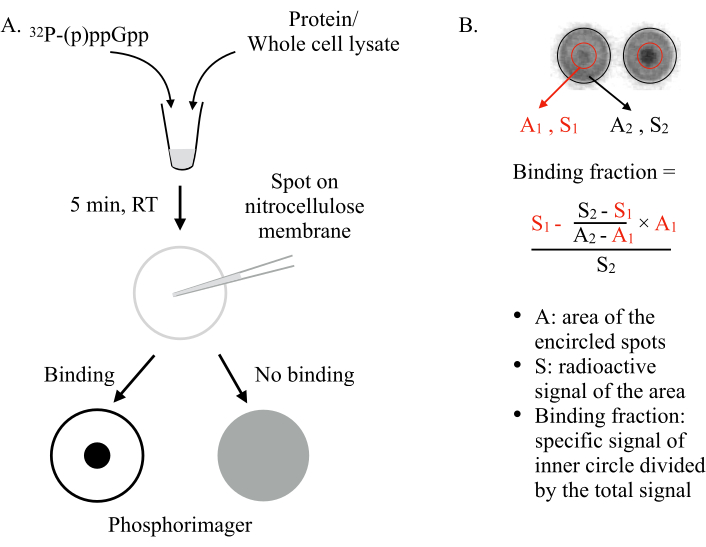
चित्रा 1:DRaCALA का सिद्धांत। (A)DRaCALA परख की योजनाबद्ध । विवरण के लिए पाठ देखें। (ख)बाध्यकारी अंश की मात्राकरण और गणना । विवरण के लिए पाठ देखें। संक्षेप में, DRaCALA स्पॉट दो हलकों कि पूरे स्थान और भीतरी अंधेरे डॉट(यानी, बनाए रखा (पी) परीक्षण प्रोटीन के बाध्यकारी के कारण ppGpp परिमाक्रय ड्राइंग द्वारा विश्लेषण किया जाएगा । विशिष्ट बाध्यकारी संकेत गैर-विशिष्ट पृष्ठभूमि संकेत को घटाने के बाद इनर सर्कल (S1) का रेडियोधर्मी संकेत है (एक1 × ((एस 2-एस1)/(ए2-ए1))))द्वारा गणना की जाती है। बाध्यकारी अंश कुल रेडियोधर्मी संकेत (S2) द्वारा विभाजित विशिष्ट बाध्यकारी संकेत है। संक्षिप्त नाम: DRaCALA = लिगांड परख की अंतर रेडियल केशिका कार्रवाई; (p)ppGpp = गुआनोसिन पेंटा- और टेट्राफस्फेट; आर टी = कमरे का तापमान। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
Protocol
Representative Results
Discussion
DRaCALA स्क्रीनिंग प्रदर्शन में महत्वपूर्ण चरणों में से एक अच्छा पूरे सेल lysates प्राप्त करने के लिए है । सबसे पहले, परीक्षण प्रोटीन बड़ी मात्रा में और घुलनशील रूपों में उत्पादित किया जाना चाहिए। दूसरा, कोशिक?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
इस काम को एनएनएफ प्रोजेक्ट ग्रांट (NNF19OC0058331) द्वारा वाईईजेड को समर्थन दिया जाता है, और यूरोपीय संघ के क्षितिज 2020 अनुसंधान और नवाचार कार्यक्रम मैरी स्कालोडोवस्का-क्यूरी अनुदान समझौते (एनओडी 801199) के तहत एमएलएस को समर्थन दिया जाता है।

Materials
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID – Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen – Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 – Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID – Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution – 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
References
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry–a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ‘,5 ‘-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).

