Neutron Spin Echo Spectroscopie als een unieke sonde voor lipidemembraandynamiek en membraan-eiwitinteracties
Summary
Dit artikel beschrijft de protocollen voor monstervoorbereiding, gegevensreductie en gegevensanalyse in neutronenspinecho (NSE) studies van lipidemembranen. Verstandige deuteriumetikettering van lipiden maakt toegang tot verschillende membraandynamiek op mesoscopische lengte- en tijdschalen mogelijk, waarover vitale biologische processen plaatsvinden.
Abstract
Lipide bilayers vormen de belangrijkste matrix van celmembranen en zijn het primaire platform voor nutriëntenuitwisseling, eiwit-membraan interacties, en virale ontluikende, onder andere vitale cellulaire processen. Voor een efficiënte biologische activiteit moeten celmembranen stijf genoeg zijn om de integriteit van de cel en zijn compartimenten te behouden, maar toch vloeibaar genoeg om membraancomponenten, zoals eiwitten en functionele domeinen, te laten diffuus en interactief te zijn. Deze delicate balans van elastische en vloeibare membraaneigenschappen, en hun impact op de biologische functie, vereist een beter begrip van de collectieve membraandynamiek over mesoscopische lengte- en tijdschalen van belangrijke biologische processen, zoals membraanvervormingen en eiwitbindende gebeurtenissen. Een van de technieken die dit dynamisch bereik effectief kan onderzoeken, is neutronenspinecho (NSE) spectroscopie. In combinatie met deuteriumetikettering kan NSE worden gebruikt om direct toegang te krijgen tot buig- en dikteschommelingen, evenals mesoscopische dynamiek van bepaalde membraankenmerken. Dit document geeft een korte beschrijving van de NSE-techniek en schetst de procedures voor het uitvoeren van NSE-experimenten op liposomale membranen, inclusief details van monstervoorbereidings- en deuteratieschema’s, samen met instructies voor het verzamelen en verminderen van gegevens. Het artikel introduceert ook gegevensanalysemethoden die worden gebruikt om belangrijke membraanparameters te extraheren, zoals de buigstijfheidsmodulus, gebiedsdrukmodulus en viscositeit in het vlak. Om het biologische belang van NSE-studies te illustreren, worden enkele voorbeelden van membraanverschijnselen besproken die door NSE zijn onderzocht, namelijk het effect van additieven op de stijfheid van membraanbuiging, de impact van domeinvorming op membraanschommelingen en de dynamische signatuur van membraan-eiwitinteracties.
Introduction
Het begrip van celmembranen en hun functie is de afgelopen decennia opmerkelijk geëvolueerd. De vroegere visie van celmembranen als passieve lipide-bilayers die celgrenzen definiëren en huismembraaneiwitten1 is geleidelijk veranderd in een dynamisch model waarin lipide-tweelagen een belangrijke rol spelen bij het reguleren van vitale biologische processen, waaronder cellulaire signalering, moleculaire uitwisseling en eiwitfunctie – om er maar een paar2,3,4,5,6te noemen . Dit besef dat celmembranen zeer dynamisch zijn en voortdurend een remodellering en moleculaire herverdeling ondergaan, heeft wetenschappelijke verkenningen aangedrongen die verder gaan dan evenwichtsstructuren van membranen7,8,9. Dienovereenkomstig zijn meerdere benaderingen ontwikkeld om de verschillende dynamische modi in biologische en bio-geïnspireerde lipidenmembranen te bestuderen. Tot op heden hebben de meeste van deze studies zich voornamelijk gericht op diffusieve moleculaire bewegingen10,11,12,13 en macroscopische vormschommelingen14,15,16, waardoor een aanzienlijke kloof is achtergelaten in het begrijpen van de intermediaire membraandynamiek, d.w.z. collectieve fluctuaties van lipidenassemblages bestaande uit enkele 10-100s lipidemoleculen. Deze dynamieken komen voor over lengteschalen van enkele tientallen tot enkele 100 Å en in de loop van de tijd schalen van sub-ns tot enkele honderden ns (zie figuur 1), hier aangeduid als mesoscopische schalen. Het is inderdaad op deze schalen dat belangrijke biologische activiteit plaatsvindt op membraanniveau17. Dit omvat virale ontluikende18, kanaal gating19, en membraan-eiwit interacties20. Het is ook belangrijk erop te wijzen dat het energielandschap van membraaneiwitten21,22 aantoont dat conformationele veranderingen in eiwitten — noodzakelijk voor hun regulerende rol — plaatsvinden in de tijdschalen23 van collectieve membraanschommelingen , waarbij het belang van mesoscopische dynamiek in de biologische functie van celmembranen en hun bio-geïnspireerde analogen verder wordt benadrukt20. Dit artikel richt zich op de twee primaire mesoscopische dynamische modi in lipidemembranen, namelijk buigschommelingen en dikteschommelingen.
De grootste uitdaging bij het direct onderzoeken van deze fluctuatiemodi is de moeilijkheid om tegelijkertijd toegang te krijgen tot hun ruimtelijke en temporele schalen met behulp van standaardspectroscopiemethoden. De andere uitdaging is dat directe contacttechnieken dezelfde fluctuaties kunnen beïnvloeden als die welke bedoeld zijn om16te meten . Dit wordt nog verergerd door de compositorische en structurele complexiteit van biologische membranen24,25, wat resulteert in niet-homogene membraankenmerken, waaronder lipidedomeinvorming26,27,28,29,30 en membraanasymmetrie31,32,33– die selectieve sondes vereisen om de dynamiek van verschillende membraankenmerken te begrijpen. Gelukkig kunnen deze uitdagingen worden overwonnen met niet-invasieve neutronenspectroscopiemethoden, zoals neutronenspinecho (NSE), die inherent toegang hebben tot de vereiste lengte- en tijdschalen, en verder studies van selectieve membraankenmerken mogelijk maken zonder hun fysisch-chemische omgeving te veranderen34. Inderdaad, in de afgelopen jaren is NSE spectroscopie geëvolueerd tot een unieke en krachtige sonde van collectieve membraandynamica35. Resultaten van NSE-studies over lipidemembranen hebben nieuwe inzichten opgeleverd in mechanische36,37 en visco-elastische38,39 eigenschappen van lipidemembranen en hebben nieuw licht geworpen op hun potentiële rol in biologische functie40,41.
De NSE-spectroscopietechniek is gebaseerd op een interferometrisch instrumentontwerp, voor het eerst voorgesteld door Mezei42, met behulp van een reeks spin-flippers en magnetische spoelen om de precessie van de neutronenspin te regelen terwijl neutronen het instrument doorkruisen. Het ontwerp berust op magnetische spiegeling van de magnetische veldelementen ten opzichte van de monsterpositie (figuur 1A). Dit houdt in dat bij gebrek aan energie-uitwisseling tussen het neutron en het monster, het neutron hetzelfde aantal spinprecessies uitvoert, in tegengestelde richting, in de eerste en tweede helft van het instrument (let op de π-flipper tussen de twee precessiespoelen). Als gevolg hiervan blijft de uiteindelijke spintoestand van het neutron ongewijzigd ten opzichte van de begintoestand – een fenomeen dat spin-echo wordt genoemd (zie transparant neutron in figuur 1A). Wanneer het neutron echter energetisch interageert met het monster, wijzigt de energie-uitwisseling het aantal spinprecessies in de tweede helft van het instrument, wat leidt tot een andere eindspintoestand (zie figuur 1A). Dit wordt experimenteel gedetecteerd als een verlies in polarisatie, zoals later in dit artikel zal worden getoond. Voor meer informatie over de NSE-techniek wordt de lezer verwezen naar speciale technische documenten42,43,44,45.
Hier wordt een vereenvoudigde beschrijving gepresenteerd om een ruwe schatting te geven van de lengte- en tijdschalen die toegankelijk zijn met NSE. De lengteschalen worden bepaald door het bereik van haalbare wavevectortransfers, Q = 4π sin θ/λ, waarbij 2θ de verstrooiingshoek is en λ de neutronengolflengte. Men kan zien dat Q wordt ingesteld door het golflengtebereik en de mate van rotatie van de tweede arm van de spectrometer (zie figuur 1A). Een typisch Q-bereikop NSE-spectrometers is ~0.02-2 Å-146,47en tot 0.01-4 Å-1 met recente upgrades48,49, overeenkomend met ruimtelijke schalen van ~1-600 Å. Aan de andere kant wordt de toegankelijke tijdschaal berekend op basis van de totale precessiehoek (of fase) die het neutron in de magnetische precessiespoelen heeft verkregen, en blijktdeze 50te zijn : 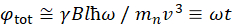 . In deze uitdrukking is t de Fouriertijd gedefinieerd als
. In deze uitdrukking is t de Fouriertijd gedefinieerd als 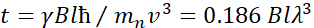 , waar is de
, waar is de 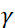 neutronen gyromagnetische verhouding,
neutronen gyromagnetische verhouding, 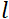 is de spoellengte, en
is de spoellengte, en 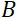 is de sterkte van het magnetisch veld van de spoel. Het is de moeite waard erop te wijzen dat de Fouriertijd een hoeveelheid is die strikt afhankelijk is van de instrumentgeometrie, de magnetische veldsterkte en de neutronengolflengte. Bijvoorbeeld, met behulp van neutronen van golflengte
is de sterkte van het magnetisch veld van de spoel. Het is de moeite waard erop te wijzen dat de Fouriertijd een hoeveelheid is die strikt afhankelijk is van de instrumentgeometrie, de magnetische veldsterkte en de neutronengolflengte. Bijvoorbeeld, met behulp van neutronen van golflengte 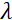 = 8 Å en instrumentinstellingen van
= 8 Å en instrumentinstellingen van 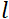 = 1,2 m en
= 1,2 m en 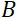 = 0,4 T, wordt de Fouriertijd berekend op t ~ 50 ns. Experimenteel wordt de Fouriertijd afgestemd door de stroom in de precessiespoelen (d.w.z. magnetische veldsterkte) te veranderen of verschillende neutronengolflengten te gebruiken, wat resulteert in typische NSE-tijdschalen van ~ 1 ps tot 100 ns. Recente upgrades in NSE-spectrometers hebben echter toegang gegeven tot langere Fourier-tijden, tot ~ 400 ns op de J-NSE-Phoenix spectrometer op de Heinz Maier-Leibnitz Zentrum51 en de SNS-NSE spectrometer in Oak Ridge National Lab48, en tot ~ 1.000 ns op de IN15 NSE spectrometer in het Institut Laue-Langevin (ILL).
= 0,4 T, wordt de Fouriertijd berekend op t ~ 50 ns. Experimenteel wordt de Fouriertijd afgestemd door de stroom in de precessiespoelen (d.w.z. magnetische veldsterkte) te veranderen of verschillende neutronengolflengten te gebruiken, wat resulteert in typische NSE-tijdschalen van ~ 1 ps tot 100 ns. Recente upgrades in NSE-spectrometers hebben echter toegang gegeven tot langere Fourier-tijden, tot ~ 400 ns op de J-NSE-Phoenix spectrometer op de Heinz Maier-Leibnitz Zentrum51 en de SNS-NSE spectrometer in Oak Ridge National Lab48, en tot ~ 1.000 ns op de IN15 NSE spectrometer in het Institut Laue-Langevin (ILL).
Naast directe toegang tot de lengte- en tijdschaal van membraandynamica heeft NSE de inherente mogelijkheden van neutronenisotopengevoeligheid52. Met name het vermogen van neutronen om verschillend te interageren met de isotopen van waterstof, het meest voorkomende element in biologische systemen, resulteert in een andere neutronenverstrooiingslengtedichtheid,34 of NSLD (het equivalent van de optische brekingsindex50), wanneer protium wordt vervangen door deuterium. Dit maakt een benadering mogelijk die bekend staat als contrastvariatie, die vaak wordt gebruikt om specifieke membraankenmerken te benadrukken of anderen te verbergen – het laatste scenario wordt contrastmatching genoemd. Een frequente toepassing van contrastvariatie/matching is de vervanging van water (NSLD = -0,56 × 10-6 Å-2) door zwaar water of D2O (NSLD = 6,4 × 10-6 Å-2) om het neutronensignaal van geprotieerde lipidemembranen (NSLD ~ 0 × 10-6 Å-2)te versterken. Deze aanpak is zeer effectief in studies van membraanstructuur omdat de penetratie van D2O in het hoofdgroepgebied van het membraan een nauwkeurige bepaling van de membraandikten mogelijk maakt (zie figuur 2A, linkerpaneel) en van de locatie van verschillende lipidesubgroepen wanneer meer geavanceerde modellen worden toegepast53,54. Dit artikel belicht enkele voorbeelden over het gebruik van contrastvariatie voor studies naar collectieve dynamiek in biomimetische membranen en bepaalde membraankenmerken.
Hier wordt de effectiviteit van NSE bij het bieden van unieke inzichten in dynamische en functionele membraaneigenschappen geïllustreerd aan de hand van tastbare voorbeelden van NSE-studies naar model- en biologisch relevante lipidenmembraansystemen met de nadruk op mesoschaaldynamiek in vrijstaande membranen, in de vorm van liposomale suspensuspensuspensussen. Voor NSE-metingen van membraandynamica in het vlak wordt de lezer verwezen naar speciale publicaties over beweidingsincidentie neutronenspin-echospectroscopie (GINSES)55,56 en andere studies van uitgelijnde multilamellar membraanstapels57,58,59,60.
Voor de eenvoud belicht dit artikel drie verschillende schema’s van membraandeuteratie geïllustreerd op een goed bestudeerd domeinvormend, of fasescheidend, lipidenbilayer-systeem van 1,2-dimyristoyl-sn-glycero-3-phosphocholine (DMPC) en 1,2-distearoyl-sn-glycero-3-phosphocholine (DSPC) mengsels61,62. De twee lipiden worden gekenmerkt door een mismatch in hun koolwaterstofketenlengte (14 carbons/tail in DMPC vs 18 carbons/tail in DSPC) en hun gel-fluid overgangstemperatuur (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C). Dit resulteert in laterale fasescheiding in DMPC:DSPC-membranen bij temperaturen tussen de bovenste en onderste overgangstemperaturen van het mengsel63. De hier overwogen deuteratieschema’s zijn gekozen om de verschillende dynamische modi aan te tonen die toegankelijk zijn in NSE-metingen op liposomale membranen, namelijk buigschommelingen, dikteschommelingen en selectieve buig-/dikteschommelingen van laterale domeinen. Alle lipidesamenstellingen worden gerapporteerd voor DMPC:DSPC-bilayers bereid met een molfractie van 70:30, met behulp van in de handel verkrijgbare protiated en perdeuterated varianten van DMPC en DSPC. Alle monstervoorbereidingsstappen zijn gebaseerd op 4 ml liposomale suspensie, in D2O, met een lipideconcentratie van 50 mg/ml, voor een totale lipidenmassa van Mtot = 200 mg per monster.
Protocol
Representative Results
Discussion
NSE is een krachtige en unieke techniek in het meten van mesoscopische dynamiek van lipidenmembranen onder verschillende omstandigheden. Het effectieve gebruik van NSE hangt af van de monsterkwaliteit, het neutronencontrast en het bereik van de toegankelijke dynamiek die voor een bepaald monster kan worden onderzocht. Er zijn dus verschillende kritieke stappen nodig voor het uitvoeren van succesvolle NSE-experimenten en het verzamelen van hoogwaardige gegevens. Een belangrijke stap in het waarborgen van het effectieve ge…
Divulgations
The authors have nothing to disclose.
Acknowledgements
R. Ashkar bedankt M. Nagao, L.-R. Stingaciu, en P. Zolnierczuk voor vele nuttige besprekingen en voor hun frequente hulp met NSE experimenten op hun respectieve beamlines. De auteurs erkennen het gebruik van neutronenspinechospectrometers bij NIST en ORNL. De NSE spectrometer bij NIST wordt ondersteund door het Center for High Resolution Neutron Scattering, een samenwerkingsverband tussen het National Institute of Standards and Technology en de National Science Foundation in het kader van overeenkomst nr. Dmr-1508249. De NSE spectrometer bij ORNL’s Spallation Neutron Source wordt ondersteund door de Scientific User Facilities Division, Office of Basic Energy Sciences, Us Department of Energy. Oak Ridge National Laboratory wordt beheerd door UT-Battelle, LLC onder US DOE Contract No. DE-AC05-00OR22725.
Materials
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes–a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich’s model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a “modeling manifesto”. Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles – as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O’Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O’Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).

