Spectroscopie d’écho de spin neutronique en tant que sonde unique pour la dynamique de la membrane lipidique et les interactions membrane-protéine
Summary
Cet article décrit les protocoles de préparation des échantillons, de réduction des données et d’analyse des données dans les études d’écho de spin neutronique (NSE) des membranes lipidiques. Le étiquetage judicieux des lipides au deutérium permet d’accéder à différentes dynamiques membranaires sur des échelles de longueur et de temps mésoscopiques, sur lesquelles se produisent des processus biologiques vitaux.
Abstract
Les bicouches lipidiques forment la matrice principale des membranes cellulaires et constituent la principale plate-forme pour l’échange de nutriments, les interactions protéine-membrane et le bourgeonnement viral, entre autres processus cellulaires vitaux. Pour une activité biologique efficace, les membranes cellulaires doivent être suffisamment rigides pour maintenir l’intégrité de la cellule et de ses compartiments, mais suffisamment fluides pour permettre aux composants membranaires, tels que les protéines et les domaines fonctionnels, de diffuser et d’interagir. Cet équilibre délicat des propriétés des membranes élastiques et fluides, et leur impact sur la fonction biologique, nécessitent une meilleure compréhension de la dynamique collective des membranes sur les échelles de longueur et de temps mésoscopiques des processus biologiques clés, par exemple les déformations membranaires et les événements de liaison aux protéines. Parmi les techniques qui peuvent sonder efficacement cette plage dynamique, il y a la spectroscopie par écho de spin neutronique (NSE). Combiné au labelage au deutérium, le NSE peut être utilisé pour accéder directement aux fluctuations de flexion et d’épaisseur ainsi qu’à la dynamique mésoscopique de certaines caractéristiques de la membrane. Cet article fournit une brève description de la technique NSE et décrit les procédures pour effectuer des expériences NSE sur les membranes liposomales, y compris des détails sur les schémas de préparation et de deutération des échantillons, ainsi que des instructions pour la collecte et la réduction des données. L’article présente également des méthodes d’analyse de données utilisées pour extraire des paramètres clés de la membrane, tels que le module de rigidité de flexion, le module de compressibilité de surface et la viscosité dans le plan. Pour illustrer l’importance biologique des études NSE, certains exemples de phénomènes membranaires sondés par NSE sont discutés, à savoir l’effet des additifs sur la rigidité de flexion de la membrane, l’impact de la formation de domaine sur les fluctuations membranaires et la signature dynamique des interactions membrane-protéine.
Introduction
La compréhension des membranes cellulaires et de leur fonction a remarquablement évolué au cours des dernières décennies. L’ancienne vision des membranes cellulaires en tant que bicouches lipidiques passives qui définissent les limites cellulaires et abritent les protéines membranaires1 s’est progressivement transformée en un modèle dynamique dans lequel les bicouches lipidiques jouent un rôle important dans la régulation des processus biologiques vitaux, y compris la signalisation cellulaire, l’échange moléculaire et la fonction protéique – pour n’en nommer que quelques-uns2,3,4,5,6. Cette prise de conscience que les membranes cellulaires sont très dynamiques, en constante évolution et redistribution moléculaire, a poussé les explorations scientifiques au-delà des structures d’équilibre des membranes7,8,9. En conséquence, de multiples approches ont été développées pour étudier les différents modes dynamiques dans les membranes lipidiques biologiques et bioinspirées. À ce jour, la majorité de ces études se sont principalement concentrées sur les mouvements moléculaires diffusifs10,11,12,13 et les fluctuations de forme macroscopiques14,15,16, laissant une lacune significative dans la compréhension de la dynamique membranaire intermédiaire, c’est-à-dire les fluctuations collectives des assemblages lipidiques constitués de quelques 10 à 100 molécules lipidiques. Ces dynamiques se produisent sur des échelles de longueur de quelques dizaines à quelques 100 Å et sur des échelles de temps de sous-ns à quelques centaines de ns (voir figure 1),appelées ici échelles mésoscopiques. C’est en effet à ces échelles que l’activité biologique clé a lieu au niveau de la membrane17. Cela inclut le bourgeonnement viral18, le canal gating19et les interactions membrane-protéine20. Il est également important de souligner que le paysage énergétique des protéines membranaires21,22 montre que les changements conformationnels dans les protéines – nécessaires à leur rôle régulateur – se produisent sur les échelles de temps ns23 des fluctuations membranaires collectives, soulignant davantage l’importance de la dynamique mésoscopique dans la fonction biologique des membranes cellulaires et de leurs analogues bioinspirés20. Cet article se concentre sur les deux principaux modes dynamiques mésoscopiques dans les membranes lipidiques, à savoir les fluctuations de flexion et les fluctuations d’épaisseur.
Le principal défi pour sonder directement ces modes de fluctuation est la difficulté d’accéder simultanément à leurs échelles spatiales et temporelles à l’aide de méthodes de spectroscopie standard. L’autre défi est que les techniques de contact direct pourraient avoir un impact sur les mêmes fluctuations qu’elles sont censées mesurer16. Ceci est encore exacerbé par la complexité compositionnelle et structurelle des membranes biologiques24,25, qui se traduit par des caractéristiques membranaires non homogènes, y compris la formation de domaine lipidique26,27,28,29,30 et l’asymétrie membranaire31,32,33– exigeant des sondes sélectives pour comprendre la dynamique des différentes caractéristiques de la membrane. Heureusement, ces défis peuvent être surmontés avec des méthodes de spectroscopie neutronique non invasives, telles que l’écho de spin neutronique (NSE), qui accèdent intrinsèquement aux échelles de longueur et de temps requises, et permettent d’autres études des caractéristiques sélectives de la membrane sans modifier leur environnement physico-chimique34. En effet, au cours des dernières années, la spectroscopie NSE a évolué pour devenir une sonde unique et puissante de dynamique membranaire collective35. Les résultats des études NSE sur les membranes lipidiques ont produit de nouvelles connaissances sur les propriétés mécaniques36,37 et viscoélastiques38,39 des membranes lipidiques et ont jeté un nouvel éclairage sur leur rôle potentiel dans la fonction biologique40,41.
La technique de spectroscopie NSE est basée sur une conception d’instrument interférométrique, proposée pour la première fois par Mezei42,utilisant une série de spin-flippers et de bobines magnétiques pour contrôler la précession du spin neutronique lorsque les neutrons traversent l’instrument. La conception repose sur la mise en miroir magnétique des éléments du champ magnétique par rapport à la position de l’échantillon(Figure 1A). Cela implique qu’en l’absence d’échange d’énergie entre le neutron et l’échantillon, le neutron effectue le même nombre de précessions de spin, dans des directions opposées, dans la première et la deuxième moitié de l’instrument (remarquez le π-flipper entre les deux bobines de précession). En conséquence, l’état de spin final du neutron reste inchangé par rapport à l’état initial – un phénomène appelé écho de spin (voir neutron transparent à la figure 1A). Cependant, lorsque le neutron interagit énergétiquement avec l’échantillon, l’échange d’énergie modifie le nombre de précessions de spin dans la seconde moitié de l’instrument, conduisant à un état de spin final différent (voir Figure 1A). Ceci est détecté expérimentalement comme une perte de polarisation, comme nous le verrons plus loin dans cet article. Pour plus de détails sur la technique NSE, le lecteur est référé aux documents techniques dédiés42,43,44,45.
Ici, une description simplifiée est présentée pour fournir une estimation approximative de la durée et des échelles de temps accessibles avec NSE. Les échelles de longueur sont déterminées par la gamme des transferts de vecteurs d’onde réalisables, Q = 4π sin θ/λ, où 2θ est l’angle de diffusion et λ est la longueur d’onde du neutron. On peut voir que Q est défini par la gamme de longueurs d’onde et l’étendue de rotation du deuxième bras du spectromètre (voir Figure 1A). Une plage Qtypique sur les spectromètres NSE est ~0,02-2 Å-146,47, et jusqu’à 0,01-4 Å-1 avec des mises à niveau récentes48,49, correspondant à des échelles spatiales de ~ 1-600 Å. D’autre part, l’échelle de temps accessible est calculée à partir de l’angle total de précession (ou phase) acquis par le neutron à l’intérieur des bobines de précession magnétique, et se trouve êtrede 50: 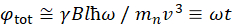 . Dans cette expression, t est le temps de Fourier défini comme
. Dans cette expression, t est le temps de Fourier défini comme 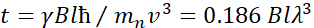 , où est le rapport
, où est le rapport 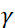 gyromagnétique des neutrons,
gyromagnétique des neutrons, 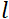 est la longueur de la bobine et est la force du champ magnétique de
est la longueur de la bobine et est la force du champ magnétique de 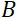 la bobine. Il convient de souligner que le temps de Fourier est une quantité qui dépend strictement de la géométrie de l’instrument, de l’intensité du champ magnétique et de la longueur d’onde des neutrons. Par exemple, en utilisant des neutrons de longueur d’onde
la bobine. Il convient de souligner que le temps de Fourier est une quantité qui dépend strictement de la géométrie de l’instrument, de l’intensité du champ magnétique et de la longueur d’onde des neutrons. Par exemple, en utilisant des neutrons de longueur d’onde 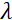 = 8 Å et des réglages d’instrument de
= 8 Å et des réglages d’instrument de 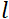 = 1,2 m et
= 1,2 m et 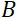 = 0,4 T, le temps de Fourier est calculé à t ~ 50 ns. Expérimentalement, le temps de Fourier est réglé en modifiant le courant dans les bobines de précession (c’est-à-dire l’intensité du champ magnétique) ou en utilisant différentes longueurs d’onde de neutrons, ce qui donne des échelles de temps NSE typiques de ~ 1 ps à 100 ns. Cependant, les récentes mises à niveau des spectromètres NSE ont permis d’accéder à des temps de Fourier plus longs, jusqu’à ~400 ns sur le spectromètre J-NSE-Phoenix au Heinz Maier-Leibnitz Zentrum51 et le spectromètre SNS-NSE au Oak Ridge National Lab48,et jusqu’à ~1 000 ns au spectromètre IN15 NSE à l’Institut Laue-Langevin (ILL)49.
= 0,4 T, le temps de Fourier est calculé à t ~ 50 ns. Expérimentalement, le temps de Fourier est réglé en modifiant le courant dans les bobines de précession (c’est-à-dire l’intensité du champ magnétique) ou en utilisant différentes longueurs d’onde de neutrons, ce qui donne des échelles de temps NSE typiques de ~ 1 ps à 100 ns. Cependant, les récentes mises à niveau des spectromètres NSE ont permis d’accéder à des temps de Fourier plus longs, jusqu’à ~400 ns sur le spectromètre J-NSE-Phoenix au Heinz Maier-Leibnitz Zentrum51 et le spectromètre SNS-NSE au Oak Ridge National Lab48,et jusqu’à ~1 000 ns au spectromètre IN15 NSE à l’Institut Laue-Langevin (ILL)49.
Outre l’accès direct à la durée et à l’échelle de temps de la dynamique membranaire, NSE a les capacités inhérentes de la sensibilité isotopiqueneutronique 52. Plus précisément, la capacité des neutrons à interagir différemment avec les isotopes de l’hydrogène, l’élément le plus abondant dans les systèmes biologiques, entraîne une densité de longueur de diffusion des neutrons différente,34 ou NSLD (l’équivalent de l’indice optique de réfraction50),lorsque le protium est substitué par du deutérium. Cela permet une approche connue sous le nom de variation de contraste, qui est couramment utilisée pour mettre en évidence des caractéristiques spécifiques de la membrane ou en dissimuler d’autres – ce dernier scénario est appelé correspondance de contraste. Une application fréquente de variation/correspondance de contraste est la substitution de l’eau (NSLD = -0,56 × 10-6 Å-2) par de l’eau lourde ouD2O (NSLD = 6,4 × 10-6 Å-2) pour amplifier le signal neutronique des membranes lipidiques protiées (NSLD ~ 0 × 10-6 Å-2). Cette approche est très efficace dans les études de la structure membranaire car la pénétration de D2O dans la région du groupe de tête de la membrane permet de déterminer avec précision les épaisseurs de membrane (voir Figure 2A,panneau de gauche) et de l’emplacement des différents sous-groupes lipidiques lorsque des modèles plus sophistiqués sont appliqués53,54. Cet article met en évidence quelques exemples sur l’utilisation de la variation de contraste pour les études de la dynamique collective dans les membranes biomimétiques et certaines caractéristiques membranaires.
Ici, l’efficacité de NSE à fournir des informations uniques sur les propriétés dynamiques et fonctionnelles des membranes est illustrée par des exemples tangibles d’études NSE sur des systèmes de membranes lipidiques modèles et biologiquement pertinents en mettant l’accent sur la dynamique à méso-échelle dans les membranes autoportantes, sous la forme de suspensions liposomales. Pour les mesures NSE de la dynamique des membranes dans le plan, le lecteur est référé à des publications dédiées sur la spectroscopie d’écho-spin neutronique d’incidence de pâturage (GINSES)55,56 et d’autres études de piles de membranes multilamellaires alignées57,58,59,60.
Pour simplifier, cet article met en évidence trois schémas différents de deutération membranaire illustrés sur un système de bicouche lipidique bien étudié formant ou séparant les phases de mélanges de 1,2-dimyristoyl-sn-glycero-3-phosphocholine (DMPC) et de 1,2-distéaroyl-sn-glycero-3-phosphocholine (DSPC)61,62. Les deux lipides sont caractérisés par un décalage dans leur longueur de chaîne hydrocarbonée (14 carbones/queue en DMPC vs 18 carbones/queue en DSPC) et leur température de transition gel-fluide (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C). Il en résulte une séparation latérale de phase dans les membranes DMPC:DSPC à des températures comprises entre les températures de transition supérieures et inférieures du mélange63. Les schémas de deutération considérés ici sont choisis pour démontrer les différents modes dynamiques accessibles dans les mesures NSE sur les membranes liposomales, à savoir les fluctuations de flexion, les fluctuations d’épaisseur et les fluctuations sélectives de flexion / épaisseur des domaines latéraux. Toutes les compositions lipidiques sont rapportées pour les bicouches DMPC:DSPC préparées à une fraction molaire de 70:30, en utilisant des variantes protiées et perdeuterées de DMPC et DSPC disponibles dans le commerce. Toutes les étapes de préparation de l’échantillon sont basées sur 4 mL de suspension liposomale, en D2O, avec une concentration lipidique de 50 mg/mL, pour une masse lipidique totale de Mtot = 200 mg par échantillon.
Protocol
Representative Results
Discussion
NSE est une technique puissante et unique dans la mesure de la dynamique mésoscopique des membranes lipidiques dans diverses conditions. L’utilisation efficace de la NSE dépend de la qualité de l’échantillon, du contraste des neutrons et de la gamme de dynamiques accessibles qui peuvent être sondées pour un échantillon donné. Ainsi, plusieurs étapes critiques sont nécessaires pour mener à bien des expériences NSE et collecter des données de haute qualité. Une étape clé pour assurer l’utilisation eff…
Divulgations
The authors have nothing to disclose.
Acknowledgements
R. Ashkar remercie M. Nagao, L.-R. Stingaciu et P. Zolnierczuk pour de nombreuses discussions utiles et pour leur aide fréquente dans les expériences NSE sur leurs lignes de faisceau respectives. Les auteurs reconnaissent l’utilisation de spectromètres d’écho de spin neutronique au NIST et à l’ORNL. Le spectromètre NSE du NIST est soutenu par le Center for High Resolution Neutron Scattering, un partenariat entre le National Institute of Standards and Technology et la National Science Foundation en vertu de l’accord no. DMR-1508249. Le spectromètre NSE de la source de neutrons de spallation de l’ORNL est soutenu par la Division des installations pour les utilisateurs scientifiques, Office of Basic Energy Sciences, Département de l’énergie des États-Unis. Oak Ridge National Laboratory est géré par UT-Battelle, LLC en vertu du contrat US DOE No. DE-AC05-00OR22725.
Materials
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes–a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich’s model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a “modeling manifesto”. Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles – as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O’Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O’Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).

