ספקטרוסקופיית הד ספין נויטרונים כגשוש ייחודי לדינמיקה של קרום השומנים ואינטראקציות בין חלבון ממברנה
Summary
מאמר זה מתאר את הפרוטוקולים להכנת מדגם, הפחתת נתונים וניתוח נתונים במחקרי הד ספין נויטרונים (NSE) של ממברנות השומנים. תיוג דיטריום נבון של שומנים מאפשר גישה לדינמיקה שונה של ממברנה על קשקשי אורך וזמן מזוסקופיים, שעליהם מתרחשים תהליכים ביולוגיים חיוניים.
Abstract
דו-שכבתיים שומנים מהווים את המטריצה העיקרית של קרום התא והם הפלטפורמה העיקרית לחילופי חומרים מזינים, אינטראקציות בין קרום חלבון וניצנים ויראליים, בין תהליכים תאיים חיוניים אחרים. לפעילות ביולוגית יעילה, קרום התא צריך להיות נוקשה מספיק כדי לשמור על שלמות התא ותאיו עדיין נוזל מספיק כדי לאפשר רכיבי ממברנה, כגון חלבונים ותחומים פונקציונליים, לפזר אינטראקציה. איזון עדין זה של תכונות ממברנה אלסטית וזורמת, והשפעתם על התפקוד הביולוגי, מחייבים הבנה טובה יותר של דינמיקת הממברנה הקולקטיבית על פני סולמות אורך וזמן מסוסקופיים של תהליכים ביולוגיים מרכזיים, למשל, עיוותי ממברנה ואירועי כריכת חלבונים. בין הטכניקות שיכולות לחקור ביעילות טווח דינמי זה הוא ספקטרוסקופיית הד ספין נויטרונים (NSE). בשילוב עם תיוג דיטריום, NSE יכול לשמש כדי לגשת ישירות כיפוף ותנודות עובי, כמו גם דינמיקה מסוסקופית של תכונות ממברנה נבחרות. מאמר זה מספק תיאור קצר של טכניקת NSE ומתאר את ההליכים לביצוע ניסויי NSE על ממברנות liposomal, כולל פרטים של הכנה מדגם תוכניות deuteration, יחד עם הוראות לאיסוף נתונים והפחתת. המאמר מציג גם שיטות ניתוח נתונים המשמשות לחילוץ פרמטרים מרכזיים של ממברנה, כגון מודולוס קשיחות כיפוף, מודולוס דחיסת שטח וצמיגות בתוך המטוס. כדי להמחיש את החשיבות הביולוגית של מחקרי NSE, דוגמאות נבחרות לתופעות ממברנה הנבדקות על ידי NSE נדונות, כלומר, ההשפעה של תוספים על קשיחות כיפוף הממברנה, ההשפעה של היווצרות תחום על תנודות ממברנה, ואת החתימה הדינמית של אינטראקציות חלבון ממברנה.
Introduction
ההבנה של קרום התא ותפקודם התפתחה להפליא בעשורים האחרונים. ההשקפה הקודמת של קרום התא כביבאי שומנים פסיביים המגדירים את גבולות התא וחלבוני קרום הבית1 הפכה בהדרגה למודל דינמי שבו דו-שכבתי השומנים ממלאים תפקיד חשוב בוויסות תהליכים ביולוגיים חיוניים, כולל איתות תאי, החלפה מולקולרית ותפקוד חלבונים – אם להזכיר כמה2,3,4,5,6. הבנה זו כי קרום התא הם דינמיים מאוד, כל הזמן עובר שיפוץ וחלוקה מולקולרית, דחף מחקרים מדעיים מעבר למבני שיווי משקל של ממברנות7,8,9. בהתאם, פותחו גישות מרובות כדי לחקור את המצבים הדינמיים השונים בממברנות שומנים ביולוגיות וביואינספיריות. עד כה, רוב המחקרים התמקדו בעיקר בתנועות מולקולריות מפוזרות10,11,12,13 ותנודות צורה מקרוסקופיות14,15,16, משאיר פער משמעותי בהבנת דינמיקת ממברנה ביניים, כלומר, תנודות קולקטיביות של מכלולי שומנים המורכבים כמה 10-100s של מולקולות השומנים. דינמיקה זו מתרחשת על פני סולמות אורך של כמה עשרות עד כמה 100 Å ועם הזמן סולמות של sub-ns עד כמה מאות ns (ראה איור 1), המכונה כאן קשקשים מסוסקופיים. זה אכן בקנה מידה זה כי פעילות ביולוגית מפתח מתרחשת ברמה הממברנה17. זה כולל ניצנים ויראליים18, ערוץ gating19, ו אינטראקציות חלבון ממברנה20. חשוב גם לציין כי הנוף האנרגטי של חלבוני הממברנה21,22 מראה כי שינויים קונפורמציה בחלבונים – הכרחיים לתפקידם הרגולטורי – מתרחשים לאורך סולמות הזמן ns23 של תנודות ממברנה קולקטיבית, תוך הדגשת החשיבות של דינמיקה מסוסקופית בתפקוד הביולוגי של קרום התא והאנלוגיות הביו-השראה שלהם20. מאמר זה מתמקד בשני המצבים הדינמיים המזוסקופיים העיקריים בממברנות השומנים, כלומר, תנודות כיפוף ותנודות עובי.
האתגר העיקרי בוחן ישיר מצבי תנודות אלה הוא הקושי לגשת בו זמנית לקנה המידה המרחבי והזמן שלהם בשיטות ספקטרוסקופיה סטנדרטיות. האתגר השני הוא שטכניקות מגע ישיר יכולות להשפיע על אותן תנודות שהן אמורות למדוד16. זה מחמיר עוד יותר על ידי המורכבות הרכב ומבנית של ממברנות ביולוגיות24,25, אשר התוצאה היא תכונות ממברנה לא הומוגנית, כולל היווצרות תחום השומנים26,27,28,29,30 ו אסימטריה ממברנה31,32,33– דורש בדיקות סלקטיביות כדי להבין את הדינמיקה של תכונות ממברנה שונות. למרבה המזל, ניתן להתגבר על אתגרים אלה בשיטות ספקטרוסקופיה לא פולשניות של נויטרונים, כגון הד ספין נויטרונים (NSE), אשר מטבען לגשת קשקשי האורך וזמן הנדרשים, ולאפשר עוד יותר מחקרים של תכונות ממברנה סלקטיבית מבלי לשנות את הסביבה הפיזיקוכימית שלהם34. ואכן, במהלך השנים האחרונות ספקטרוסקופיית NSE התפתחה לגשוש ייחודי ורב עוצמה של דינמיקת הממברנה הקולקטיבית35. תוצאות מחקרי NSE על ממברנות השומנים הפיקו תובנות חדשות על מכני36,37 ו צמיג38,39 תכונות של ממברנות השומנים לשפוך אור חדש על תפקידם הפוטנציאלי בתפקוד הביולוגי40,41.
טכניקת הספקטרוסקופיה של NSE מבוססת על עיצוב מכשיר אינטרפרומטרי, שהוצע לראשונה על ידי Mezei42, באמצעות סדרה של ספין סנפירים סלילים מגנטיים כדי לשלוט על הקדירה של ספין הנויטרונים כמו נייטרונים לחצות את המכשיר. העיצוב נשען על שיקוף מגנטי של יסודות השדה המגנטי ביחס למיקום המדגם(איור 1A). משמעות הדבר היא כי בהיעדר חילופי אנרגיה בין הנויטרונים לדגימה, הנויטרונים מבצעים את אותו מספר של נטיות ספין, בכיוונים מנוגדים, במחצית הראשונה והשנייה של המכשיר (שימו לב π-סנפיר בין שני סלילי ההקדמה). כתוצאה מכך, מצב הספין הסופי של הנויטרונים נותר ללא שינוי ביחס למצב ההתחלתי – תופעה המכונה ספין-הד (ראו נויטרונים שקופים באיור 1A). עם זאת, כאשר הנויטרונים מתקשרים במרץ עם המדגם, חילופי האנרגיה משנה את מספר ההטיות בסיבוב במחצית השנייה של המכשיר, מה שמוביל למצב ספין סופי אחר (ראו איור 1A). זה מזוהה באופן ניסיוני כהפסד בקיטוב, כפי שיוצג בהמשך מאמר זה. לקבלת פרטים נוספים על טכניקת NSE, הקורא מופנה מאמרים טכניים ייעודיים42,43,44,45.
כאן, תיאור פשוט מוצג כדי לספק הערכה גסה של סולמות האורך והשעה הנגישים עם NSE. סולמות האורך נקבעים על ידי טווח העברות wavevector בר השגה, Q = 4π sin θ/ λ, שבו2 θ הוא זווית הפיזור ו λ הוא אורך הגל הנויטרונים. ניתן לראות ש-Q מוגדר על ידי טווח אורך הגל והיקף הסיבוב של הזרוע השנייה של הספקטרומטר (ראו איור 1A). טווח Qטיפוסי על ספקטרומטרים NSE הוא ~ 0.02-2 Å-146,47, ועד 0.01-4 Å-1 עם שדרוגים אחרונים48,49, המקביל לקנה מידה מרחבי של ~ 1-600 Å. מצד שני, סולם הזמן הנגיש מחושב מזווית ההקדמה הכוללת (או השלב) שנרכשה על ידי הנויטרונים בתוך סלילי ההקדמה המגנטית, ונמצא50: 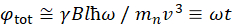 . בביטוי זה, t הוא זמן פורייה המוגדר
. בביטוי זה, t הוא זמן פורייה המוגדר 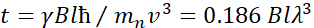 כ, איפה
כ, איפה 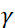 היחס הגירומגנטי של הנויטרונים,
היחס הגירומגנטי של הנויטרונים, 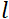 הוא אורך סליל,
הוא אורך סליל, 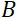 והוא כוחו של השדה המגנטי של סליל. ראוי לציין כי זמן פורייה הוא כמות התלויה לחלוטין בגיאומטריית המכשיר, חוזק השדה המגנטי ואורך הגל הנויטרונים. לדוגמה, באמצעות נייטרונים של אורך גל
והוא כוחו של השדה המגנטי של סליל. ראוי לציין כי זמן פורייה הוא כמות התלויה לחלוטין בגיאומטריית המכשיר, חוזק השדה המגנטי ואורך הגל הנויטרונים. לדוגמה, באמצעות נייטרונים של אורך גל 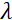 = 8 Å והגדרות מכשיר של
= 8 Å והגדרות מכשיר של 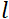 = 1.2 מ ‘ ו
= 1.2 מ ‘ ו 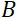 – 0.4 T, זמן פורייה מחושב להיות t ~ 50 ns. באופן ניסיוני, זמן פורייה מכוון על ידי שינוי הזרם בסלילי הקדם (כלומר, חוזק השדה המגנטי) או באמצעות אורכי גל נויטרונים שונים, וכתוצאה מכך סולמות זמן טיפוסיים של NSE של ~ 1 ps עד 100 ns. עם זאת, שדרוגים אחרונים בספקטרומטר NSE אפשרו גישה לזמנים ארוכים יותר של פורייה, עד כ-400 נ’ב בספקטרומטר J-NSE-Phoenix בספקטרומטר היינץ מאייר-לייבניץ זנטרום51 וספקטרומטר SNS-NSE במעבדה הלאומית אוק רידג’48, ועד ~ 1,000 ns בספקטרומטר IN15 NSE במכון לאו-לנגווין (ILL)49.
– 0.4 T, זמן פורייה מחושב להיות t ~ 50 ns. באופן ניסיוני, זמן פורייה מכוון על ידי שינוי הזרם בסלילי הקדם (כלומר, חוזק השדה המגנטי) או באמצעות אורכי גל נויטרונים שונים, וכתוצאה מכך סולמות זמן טיפוסיים של NSE של ~ 1 ps עד 100 ns. עם זאת, שדרוגים אחרונים בספקטרומטר NSE אפשרו גישה לזמנים ארוכים יותר של פורייה, עד כ-400 נ’ב בספקטרומטר J-NSE-Phoenix בספקטרומטר היינץ מאייר-לייבניץ זנטרום51 וספקטרומטר SNS-NSE במעבדה הלאומית אוק רידג’48, ועד ~ 1,000 ns בספקטרומטר IN15 NSE במכון לאו-לנגווין (ILL)49.
מלבד גישה ישירה לאורך ולקנה המידה של דינמיקת הממברנה, ל- NSE יש את היכולות הטבועות של רגישות איזוטופ נויטרונים52. באופן ספציפי, היכולת של נייטרונים לקיים אינטראקציה שונה עם האיזוטופים של מימן, היסוד הנפוץ ביותר במערכות ביולוגיות, גורמת לצפיפות אורך פיזור נויטרונים שונה,34 או NSLD (המקבילה של האינדקס האופטי של שבירה50), כאשר פרוטיום מוחלף על ידי דיטריום. זה מאפשר גישה המכונה וריאציה ניגודית, אשר משמש בדרך כלל כדי להדגיש תכונות ממברנה ספציפיות או להסתיר אחרים – התרחיש האחרון מכונה התאמת ניגוד. יישום תכוף של וריאציה /התאמה ניגודיות הוא החלפת מים (NSLD = -0.56 × 10-6 Å-2) על ידי מים כבדים או D2O (NSLD = 6.4 × 10-6 Å-2) כדי להגביר את אות הנויטרונים מקרום השומנים פרוטיים (NSLD ~ 0 × 10-6 Å-2). גישה זו יעילה מאוד במחקרים של מבנה הממברנה מכיוון שחדירת D2O לאזור קבוצת הראש של הממברנה מאפשרת קביעה מדויקת של עובי הממברנה (ראה איור 2A, פאנל שמאלי) ושל המיקום של קבוצות משנה שומנים שונות כאשר מודלים מתוחכמים יותר מוחלים53,54. מאמר זה מדגיש כמה דוגמאות על השימוש וריאציה ניגודיות למחקרים של דינמיקה קולקטיבית בממברנות biomimetic ותכונות ממברנה לבחור.
כאן, האפקטיביות של NSE במתן תובנות ייחודיות על תכונות ממברנה דינמית ופונקציונלית מודגמת באמצעות דוגמאות מוחשיות של מחקרי NSE על מודל ומערכות קרום שומנים רלוונטיות ביולוגית עם דגש על דינמיקה mesoscale בממברנות עצמאיות, בצורה של מתלים liposomal. עבור מדידות NSE של דינמיקת ממברנה בתוך המטוס, הקורא מתייחס פרסומים ייעודיים על ספקטרוסקופיית ספין-הד-הד של נויטרונים (GINSES)55,56 ומחקרים אחרים של ערימות ממברנה רב-שלמית מיושרות57,58,59,60.
לפשטות, מאמר זה מדגיש שלוש תוכניות שונות של פירוק ממברנה מאויר על דומיין נחקר היטב, או הפרדת פאזה, מערכת bilayer השומנים של 1,2-dimyristoyl-sn-גליצרי-3-פוספוצולין (DMPC) ו 1,2-distearoyl-sn-גלייצרו-3-פוספוצולין (DSPC) תערובות61,62. שני השומנים מאופיינים בחוסר התאמה באורך שרשרת הפחמימנים שלהם (14 פחמנים/זנב ב- DMPC לעומת 18 פחמנים /זנב ב- DSPC) וטמפרטורת המעבר של נוזל הג’ל שלהם (Tm, DMPC = 23 °C לעומת Tm, DSPC = 55 °C (C). התוצאה היא הפרדת פאזה לרוחב ב DMPC: DSPC ממברנות בטמפרטורות בין טמפרטורות המעבר העליונות והתחתונות של התערובת63. תוכניות ההסתה הנחשבות כאן נבחרות כדי להדגים את המצבים הדינמיים השונים הנגישים במדידות NSE על ממברנות ליפוזומליות, כלומר, תנודות כיפוף, תנודות עובי ותנודות כיפוף / עובי סלקטיביות של תחומים לרוחב. כל הרכבי השומנים מדווחים עבור DMPC:DSPC bilayers מוכן בשבר שומה של 70:30, באמצעות גרסאות פרוטציה ו perdeuterated זמין מסחרית של DMPC ו- DSPC. כל שלבי הכנת המדגם מבוססים על 4 מ”ל של השעיה ליפוזומית, ב D2O, עם ריכוז שומנים בדם של 50 מ”ג /מ”ל, עבור מסת שומנים כוללת של Mtot = 200 מ”ג לדגימה.
Protocol
Representative Results
Discussion
NSE היא טכניקה רבת עוצמה וייחודית במדידת דינמיקה מסוסקופית של ממברנות שומנים בדם בתנאים שונים. הניצול היעיל של NSE תלוי באיכות המדגם, בניגודיות הנויטרונים ובמגוון הדינמיקה הנגישת שניתן לבדוק עבור מדגם נתון. לכן, נדרשים מספר צעדים קריטיים לביצוע ניסויי NSE מוצלחים ואיסוף נתונים באיכות גבוהה. ?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
ר. אשקר תודה מ. נגאו, ל.ר. סטינצ’יו, ו P. Zolnierczuk לדיונים שימושיים רבים ועל עזרתם התכופה בניסויי NSE על קווי הקורה שלהם. המחברים מכירים בשימוש בספקטרומטרי הד ספין נויטרונים ב- NIST ו- ORNL. ספקטרומטר NSE ב- NIST נתמך על ידי המרכז לפיזור ניוטרון ברזולוציה גבוהה, שותפות בין המכון הלאומי לתקנים וטכנולוגיה לבין הקרן הלאומית למדע בהסכם לא. DMR-1508249. ספקטרומטר NSE במקור הנויטרונים של ORNL נתמך על ידי חטיבת מתקני המשתמש המדעי, המשרד למדעי האנרגיה הבסיסית, מחלקת האנרגיה של ארה”ב. המעבדה הלאומית אוק רידג’ מנוהלת על ידי UT-Battelle, LLC תחת חוזה DOE בארה”ב לא. DE-AC05-00OR22725.
Materials
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes–a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich’s model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a “modeling manifesto”. Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles – as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O’Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O’Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).

