Untersuchungen von Chaperon-Cochaperon-Interaktionen mit homogenem Bead-based Assay
Summary
Dieses Protokoll stellt eine Technik zur Untersuchung von Protein-Protein-Interaktionen unter Verwendung von Glutathion-verknüpften Spenderperlen mit GST-verschmolzenen TPR-Motiv-Co-Chaperonen und Akzeptorperlen in Verbindung mit einem Hsp90-abgeleiteten Peptid vor. Wir haben diese Technik verwendet, um kleine Moleküle zu screenen, um Hsp90-FKBP51- oder Hsp90-FKBP52-Interaktionen zu stören, und potente und selektive Hsp90-FKBP51-Interaktionshemmer identifiziert.
Abstract
Das Targeting der Wechselwirkungen mit dem Hitzeschockprotein 90 (Hsp90)-Cochaperon bietet die Möglichkeit, Hsp90-abhängige intrazelluläre Prozesse gezielt zu regulieren. Das konservierte MEEVD-Pentapeptid am C-Terminus von Hsp90 ist für die Wechselwirkung mit dem Tetratricopeptid-Wiederholungsmotiv (TPR) von Co-Chaperonen verantwortlich. FK506-bindendes Protein (FKBP) 51 und FKBP52 sind zwei ähnliche TPR-Motiv-Co-Chaperone, die an steroidhormonabhängigen Erkrankungen mit unterschiedlichen Funktionen beteiligt sind. Daher bietet die Identifizierung von Molekülen, die spezifisch Wechselwirkungen zwischen Hsp90 und FKBP51 oder FKBP52 blockieren, ein vielversprechendes therapeutisches Potenzial für mehrere menschliche Krankheiten. Hier beschreiben wir das Protokoll für einen amplifizierten lumineszierenden homogenen Näherungsassay zur Untersuchung von Wechselwirkungen zwischen Hsp90 und seinen Partner-Co-Chaperonen FKBP51 und FKBP52. Zunächst haben wir die TPR-motivhaltigen Proteine FKBP51 und FKBP52 in Glutathion-S-Transferase (GST)-getaggter Form gereinigt. Unter Verwendung der Glutathion-verknüpften Spenderperlen mit GST-verschmolzenen TPR-Motivproteinen und den Akzeptorperlen in Verbindung mit einem 10-mer C-terminalen Peptid von Hsp90 haben wir Protein-Protein-Interaktionen in einer homogenen Umgebung untersucht. Wir haben diesen Assay verwendet, um kleine Moleküle zu screenen, um Hsp90-FKBP51- oder Hsp90-FKBP52-Interaktionen zu stören, und potente und selektive Hsp90-FKBP51-Interaktionshemmer identifiziert.
Introduction
Molekulare Chaperone tragen zur Protein-Homöostase bei, einschließlich Proteinfaltung, Transport und Abbau. Sie regulieren mehrere zelluläre Prozesse und sind mit zahlreichen Krankheiten wie Krebs und neurodegenerativen Erkrankungen verbunden1. Das Hitzeschockprotein 90 (Hsp90) ist eines der wichtigsten Chaperone, dessen Funktion von Konformationsänderungen abhängt, die durch ATP-Hydrolyse und Bindung an Client-Proteine, die durch seine Co-Chaperone vermitteltwerden, angetriebenwerden 2 . Trotz eines offensichtlichen Potenzials von Hsp90 als therapeutisches Ziel stellt die Feinabstimmung seiner Funktion eine große Herausforderung dar. Es gibt mehrere Hsp90-Inhibitoren, die auf die N-terminale ATP-Bindungsregion abzielen, die in klinischen Studien untersucht wurden, aber keiner von ihnen wurde für die Vermarktung zugelassen3. Aufgrund des Fehlens einer gut definierten Ligandenbindungstasche4hatte das Targeting der C-terminalen Region von Hsp90 nur begrenzten Erfolg4. Kürzlich wurde die Störung von Hsp90-Cochaperon-Wechselwirkungen durch kleine Moleküle als alternative Strategie untersucht5. Die Ausrichtung auf die Hsp90-Cochaperon-Interaktionen würde keine allgemeine Zellstressreaktion hervorrufen und bietet die Möglichkeit, verschiedene intrazelluläre Prozesse spezifisch zu regulieren. Das konservierte MEEVD-Pentapeptid am C-Terminus von Hsp90 ist für die Wechselwirkung mit dem Tetratricopeptid-Wiederholungsmotiv (TPR) der Co-Chaperone verantwortlich6. Von den 736 TPR-motivhaltigen Proteinen, die in der humanen Proteindatenbank annotiert sind, interagieren ~20 verschiedene Proteine über dieses Peptid mit Hsp907. Moleküle, die um die MEEVD-Peptidbindung konkurrieren, würden die Wechselwirkungen zwischen Hsp90 und Co-Chaperonen, die eine TPR-Domäne enthalten, stören. Die Peptidbindungsstelle hat eine ähnliche tertiäre Struktur, aber die Gesamthomologie zwischen verschiedenen TPR-Motivdomänen ist relativ niedrig7, was die Möglichkeit bietet, Moleküle zu identifizieren, die spezifisch in der Lage sind, Wechselwirkungen zwischen Hsp90 und bestimmten TPR-Motiv-Co-Chaperonen zu blockieren. Unter diesen TPR-Motiv-Co-Chaperonen sind FK506-bindendes Protein (FKBP) 51 und FKBP52 Regulatoren der Steroidhormonrezeptor-Signalisierung (SHR) und an mehreren Steroidhormon-abhängigen Krankheiten beteiligt, darunter Krebs, stressbedingte Krankheiten, Stoffwechselerkrankungen und Alzheimer-Krankheit8. Obwohl FKBP51 und FKBP52 > Sequenzähnlichkeit von 80% teilen, unterscheiden sich ihre Funktionen: FKBP52 ist ein positiver Regulator der SHR-Aktivität, während FKBP51 in den meisten Fällen ein negativer Regulator ist8. Daher bietet die Identifizierung von Molekülen, die spezifisch Wechselwirkungen zwischen Hsp90 und FKBP51 oder FKBP52 blockieren, ein vielversprechendes therapeutisches Potenzial für verwandte Krankheiten.
Einmplified Luminescent Proximity Homogenous Assay (AlphaScreen) wurde erstmals 1994 von Ullman EF et al.9entwickelt. Jetzt wird es häufig verwendet, um verschiedene Arten von biologischen Interaktionen wie Peptid10, Protein11, DNA12, RNA13und Zucker14zu erkennen . Bei dieser Technik gibt es zwei Arten von Perlen (Durchmesser 200 nm), eine ist die Spenderperle und die andere ist die Akzeptorperle. Die Biomoleküle werden auf diesen Perlen immobilisiert; ihre biologischen Wechselwirkungen bringen Spender- und Akzeptorperlen in die Nähe. Bei 680 nm beleuchtet ein Photosensibilisator in der Spenderperle und wandelt Sauerstoff in Singulett-Sauerstoff um. Da der Singsingstoff eine kurze Lebensdauer hat, kann er nur bis zu 200 nm diffundieren. Befindet sich die Akzeptorperle in der Nähe, reagiert ihr Thioxenderivat mit dem Singulett Sauerstoff, der bei 370 nm Chemilumineszenz erzeugt. Diese Energie aktiviert weiterhin Fluorophore in derselben Akzeptorperle, um Licht bei 520-620 nm15zu emittieren. Wenn die biologischen Wechselwirkungen gestört sind, können die Akzeptorperle und die Spenderperle keine Nähe erreichen, was zum Sauerstoffzerfall des Singuletts und zu einem schwach produzierten Signal führt.
Hier beschreiben wir ein Protokoll, das diese Technik zum Screening kleiner Moleküle verwendet, die Wechselwirkungen zwischen Hsp90- und TPR-Co-Chaperonen hemmen, insbesondere FKBP51 und FKBP52. Die 10 Aminosäuren langen Peptide, die Hsp90 extremem C-Terminus entsprechen, sind an Akzeptorperlen gebunden. Gereinigte GST-markierte TPR-Co-Chaperone interagieren mit Glutathion-verknüpften Spenderperlen. Wenn die Wechselwirkung zwischen Hsp90-abgeleiteten Peptiden und TPR-Motiv-Co-Chaperonen die Perlen zusammenbringt, wird ein verstärktes Signal erzeugt (Abbildung 1A). Wenn die gescreenten kleinen Moleküle die Wechselwirkungen zwischen Hsp90- und TPR-Motiv-Co-Chaperonen hemmen können, wird dieses verstärkte Signal verringert (Abbildung 1B). Ihr IC50 kann durch quantitative Messung berechnet werden. Dieses Protokoll kann auf alle Chaperon-TPR-Motiv-Co-Chaperon-Interaktionen von Interesse erweitert werden und ist von großer Bedeutung für die Entwicklung neuartiger Moleküle, die insbesondere die Wechselwirkung zwischen Hsp90 und FKBP51 oder FKBP52 blockieren.
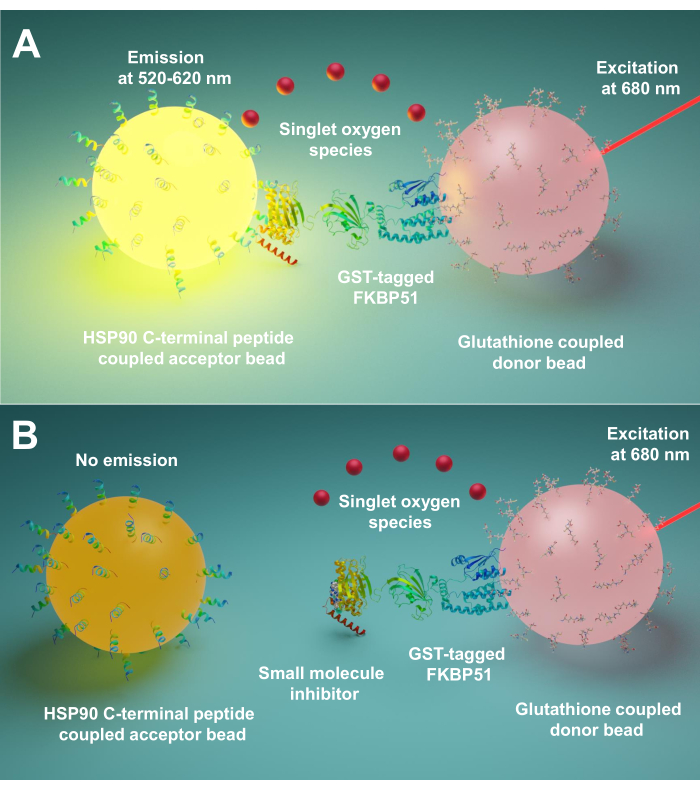
Abbildung 1: Das Grundprinzip dieses Assays. (A) Gereinigte GST-FKBP51 interagiert mit Glutathion-verknüpften Spenderperlen. Die 10 Aminosäuren langen Peptide, die dem extremen C-Terminus von Hsp90 entsprechen, sind an Akzeptorperlen gebunden. Die Interaktion zwischen Hsp90-abgeleiteten Peptiden und der TPR-Domäne von FKBP51 bringt die Spender- und Akzeptorperlen in die Nähe. Bei 680 nm beleuchtet ein Photosensibilisator in der Spenderperle und wandelt Sauerstoff in Singulett-Sauerstoff um. Das Thioxenderivat auf der Akzeptorperle reagiert mit dem Singulett Sauerstoff und erzeugt eine Chemilumineszenz bei 370 nm. Diese Energie aktiviert weiterhin Fluorophore in derselben Akzeptorperle, um Licht bei 520-620 nm zu emittieren. (B) Wenn kleine Moleküle die Wechselwirkungen zwischen Hsp90 und FKBP51 hemmen, können die Spender- und Akzeptorperlen keine Nähe erreichen. Dann zerfällt der Singulett-Sauerstoff mit kurzer Lebensdauer und es wird kein nachweisbares Signal erzeugt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protocol
Representative Results
Discussion
Hier beschreiben wir ein Protokoll mit dem Assay zum Screening kleiner Moleküle, die Wechselwirkungen zwischen Hsp90 und TPR-Motiv-Co-Chaperonen, insbesondere FKBP51 und FKBP52, hemmen. Sein hoher Z’-Wert (>0,8) zeigt die Robustheit und Zuverlässigkeit für ein Format mit hohem Durchsatz. Ergebnisse können innerhalb einer Stunde erzielt werden, und kleine Mengen an Perlen, Protein und Verbindungen sind erforderlich. Darüber hinaus könnte dieses Protokoll leicht auf alle Hsp90 / Hsp70 – TPR-Motiv-Co-Chaperon-Interakt…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Diese Studie wurde durch Zuschüsse des Schwedischen Forschungsrats (2018-02843), der Brain Foundation (Fo 2019-0140), der Foundation for Geriatric Diseases am Karolinska Institutet, der Gunvor and Josef Anérs Foundation, der Magnus Bergvalls Foundation, der Gun and Bertil Stohnes Foundation, der Tore Nilssons Foundation for Medical Research, der Margaretha af Ugglas Foundation und der Foundation for Old Servants unterstützt.
Materials
| 384-well plates | Perkin Elmer | 6008350 | Assay volume 25 ml |
| Amicon 10.000 MWCO centrifugation unit | Millipore | UFC901008 | Concentrate protein |
| Ampicillin | Sigma | A0166 | Antibiotics |
| Bacteria shaker Unimax 1010 | Heidolph | Culture bacteria | |
| cDNA clones for human FKBP51 | Source BioScience | clone id: 5723416 | pCMV-SPORT6 vector |
| cDNA clones for human FKBP52 | Source BioScience | clone id: 7474554 | pCMV-SPORT6 vector |
| Chemically Competent E. coli | Invitrogen | C602003 | One Shot BL21 Star (DE3) |
| Data analysis software | GraphPad Prism | 9.0.0 | Analysis data and make figures |
| Data analysis software | Excel | Analysis data | |
| DMSO | Supelco | 1.02952.1000 | Dilute compounds |
| DPBS | Gibco | 14190-144 | Prepare solution |
| EDTA | Calbiochem | 344504 | Prevent proteolysis during sonication |
| Glutathione | Sigma | G-4251 | Elute GST-tagged proteins |
| Glutathione donor beads | Perkin Elmer | 6765300 | Donor bead |
| GST-trap column | Cytiva (GE Healthcare) | 17528201 | Purify GST-tagged proteins |
| Isopropyl-β-D-thiogalactoside | Thermo Fisher Scientific | R0392 | Induce protein expression |
| LB Broth (Miller) | Sigma | L3522 | Microbial growth medium |
| PCR instrument | BIO-RAD | S1000 Thermal Cycler | Amplification/PCR |
| PD-10 column | Cytiva (GE Healthcare) | 17085101 | Solution exchange |
| pGEX-6P-1 vector | Cytiva (GE Healthcare) | 28954648 | Plasmid |
| pGEX-6P-2 vector | Cytiva (GE Healthcare) | 28954650 | Plasmid |
| Plate reader | Perkin Elmer | EnSpire 2300 Multilabel Reader | Read alpha plate |
| Plate reader software | Perkin Elmer | EnSpire Manager | Plate reader software |
| Plate reader software protocol | Perkin Elmer | Alpha 384-well Low volume | Use this protocol to read plate |
| PMSF | Sigma | P7626 | Prevent proteolysis during sonication |
| protease inhibitor cocktail | Sigma | S8830 | Prevent proteolysis during sonication |
| Sodium azide | Sigma | S2002 | As a preservative |
| Sodium cyanoborohydride (NaBH3CN) | Sigma | 156159 | Activates matrix for coupling |
| Ten amino acid peptide NH2-EDASRMEEVD-COOH corresponding to amino acids 714-724 of human Hsp90 beta isoform | Peptide 2.0 inc | Synthesize Hsp90 C-terminal peptide | |
| Test-Tube Rotator | LABINCO | Make end-over-end agitation | |
| Tris-HCl | Sigma | 10708976001 | Block unreacted sites of acceptor beads |
| Tween-20 | Sigma | P1379 | Prevent beads aggregation |
| Ultra centrifuge Avanti J-20 XP | Beckman Coulter | Centrifuge to get bacteria cell pellets | |
| Ultrasonic cell disruptor | Microson | Sonicate cells to release protein | |
| Unconjugated acceptor beads | Perkin Elmer | 6762003 | Acceptor beads |
| XCell SureLock Mini-Cell and XCell II Blot Module | Invitrogen | EI0002 | SDS-PAGE |
References
- Muchowski, P. J., Wacker, J. L. Modulation of neurodegeneration by molecular chaperones. Nature Reviews Neuroscience. 6 (1), 11-22 (2005).
- Eckl, J. M., Richter, K. Functions of the Hsp90 chaperone system: lifting client proteins to new heights. International Journal of Biochemistry and Molecular Biology. 4 (4), 157-165 (2013).
- Yuno, A. Clinical evaluation and biomarker profiling of Hsp90 inhibitors. Methods in Molecular Biology. 1709, 426-441 (2018).
- Dutta Gupta, S., Bommaka, M. K., Banerjee, A. Inhibiting protein-protein interactions of Hsp90 as a novel approach for targeting cancer. European Journal of Medicinal Chemistry. 178, 48-63 (2019).
- Pavlov, P. F., Hutter-Paier, B., Havas, D., Windisch, M., Winblad, B. Development of GMP-1 a molecular chaperone network modulator protecting mitochondrial function and its assessment in fly and mice models of Alzheimer’s disease. Journal of Cellular and Molecular Medicine. 22 (7), 3464-3474 (2018).
- Young, J. C., Obermann, W. M., Hartl, F. U. Specific binding of tetratricopeptide repeat proteins to the C-terminal 12-kDa domain of hsp90. Journal of Biological Chemistry. 273 (29), 18007-18010 (1998).
- Scheufler, C., et al. Structure of TPR domain-peptide complexes: critical elements in the assembly of the Hsp70-Hsp90 multichaperone machine. Cell. 101 (2), 199-210 (2000).
- Storer, C. L., Dickey, C. A., Galigniana, M. D., Rein, T., Cox, M. B. FKBP51 and FKBP52 in signaling and disease. Trends in Endocrinology & Metabolism. 22 (12), 481-490 (2011).
- Ullman, E. F., et al. Luminescent oxygen channeling immunoassay: measurement of particle binding kinetics by chemiluminescence. Proceedings of the National Academy of Sciences of the United States of America. 91 (12), 5426-5430 (1994).
- Wigle, T. J., et al. Screening for inhibitors of low-affinity epigenetic peptide-protein interactions: an AlphaScreen-based assay for antagonists of methyl-lysine binding proteins. Journal of Biomolecular Screening. 15 (1), 62-71 (2010).
- Guenat, S., et al. Homogeneous and nonradioactive high-throughput screening platform for the characterization of kinase inhibitors in cell lysates. Journal of Biomolecular Screening. 11 (8), 1015-1026 (2006).
- Sabatucci, A., et al. A new methodological approach for in vitro determination of the role of DNA methylation on transcription factor binding using AlphaScreen(R) analysis: Focus on CREB1 binding at hBDNF promoter IV. Journal of Neuroscience Methods. 341, 108720 (2020).
- Mills, N. L., Shelat, A. A., Guy, R. K. Assay Optimization and Screening of RNA-Protein Interactions by AlphaScreen. Journal of Biomolecular Screening. 12 (7), 946-955 (2007).
- Huang, X., et al. A competitive alphascreen assay for detection of hyaluronan. Glycobiology. 28 (3), 137-147 (2018).
- Principles of alphascreen amplified luinescent proximmity homogenous assay. PerkinElmer Life Sciences Available from: https://www.perkinelmer.com/lab-solutions/resources/docs/APP_AlphaScreen_Principles.pdf (2021)
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).

