זיהוי גלובלי של רשתות אינטראקציה בין תרגומים משותפים על ידי פרופיל ריבוזום סלקטיבי
Summary
אינטראקציות של תרגום משותף ממלאות תפקיד מכריע בשינויים בשרשרת המתהווה, במיקוד, בקיפול ובמסלולי הרכבה. כאן, אנו מתארים פרופיל ריבוזום סלקטיבי, שיטה לניתוח ישיר של in vivo, של אינטראקציות אלה במודל eukaryote Saccharomyces cerevisiae.
Abstract
בשנים האחרונות התברר כי ריבוזומים לא רק מפענחים את ה-mRNA שלנו אלא גם מנחים את הופעתה של שרשרת הפוליפפטידים לסביבה התאית הצפופה. ריבוזומים מספקים את הפלטפורמה לקשירה מבוקרת מרחבית וקינטית של גורמי מיקוד ממברנה, אנזימים משנים ומלווים מתקפלים. אפילו ההרכבה לקומפלקסים אוליגומריים מסדר גבוה, כמו גם לשלבי היווצרות רשת חלבונים-חלבונים, התגלו לאחרונה כמתואמים עם סינתזה.
כאן אנו מתארים פרופיל ריבוזום סלקטיבי, שיטה שפותחה כדי ללכוד אינטראקציות תרגום משותף in vivo. אנו נפרט את שלבי טיהור הזיקה השונים הנדרשים ללכידת קומפלקסים של שרשרת ריבוזום-מתהווה יחד עם אינטראקציות תרגום משותף, כמו גם את שלבי מיצוי ה-mRNA, הרחקת הגודל, השעתוק ההפוך, הריצוף העמוק וניתוח הביג-דאטה, הנדרשים כדי לפענח אינטראקציות של תרגום משותף ברזולוציה של כמעט קודון.
Introduction
Selective Ribosome Profiling (SeRP) היא השיטה היחידה, עד כה, הלוכדת ומאפיינת אינטראקציות תרגום משותף, in vivo, באופן ישיר 1,2,3,4,5,6. SeRP מאפשר פרופיל גלובלי של אינטראקציות של כל גורם עם תרגום ריבוזומים ברזולוציה של כמעט קודון 2,7.
השיטה מסתמכת על הקפאת הבזק של תאים גדלים ושמירה על תרגום פעיל. לאחר מכן, ליזטים של תאים מטופלים באמצעות RNase I כדי לעכל את כל ה-mRNA בתא, למעט שברי mRNA המוגנים על-ידי ריבוזומים המכונים “עקבות ריבוזומים”. לאחר מכן מתפצלים המדגם לשני חלקים; חלק אחד משמש ישירות לבידוד של כל עקבות הריבוזומליות התאיות, המייצגות את כל התרגום המתמשך בתא. החלק השני משמש לטיהור זיקה של תת-קבוצה ספציפית של ריבוזומים הקשורים לגורם עניין, לדוגמה: שינוי אנזימים, גורמי טרנסלוקציה, מלווים מתקפלים ואינטראקציות של הרכבה מורכבת. עקבות הריבוזומליות המטוהרות זיקה נקראות באופן קולקטיבי האינטראקציה. לאחר מכן, ה-mRNA המוגנים בריבוזום מופקים ומשמשים ליצירת ספריית cDNA, ולאחר מכן לריצוף עמוק.
ניתוח השוואתי של סך כל דגימות התרגום והאינטראקציה מאפשר זיהוי של כל האורפים המתקשרים עם גורם העניין, כמו גם אפיון של כל פרופיל אינטראקציה של orf. פרופיל זה מדווח על רצפי ההתחלה והסיום המדויקים של ההתקשרות שמהם ניתן להסיק את הקודונים המפוענחים ואת השאריות המתאימות של שרשרת הפוליפפטידים המתהווה, כמו גם על שינויי מהירות הריבוזומים במהלך האינטראקציה 7,8. איור 1 מתאר את הפרוטוקול כשרטוט.
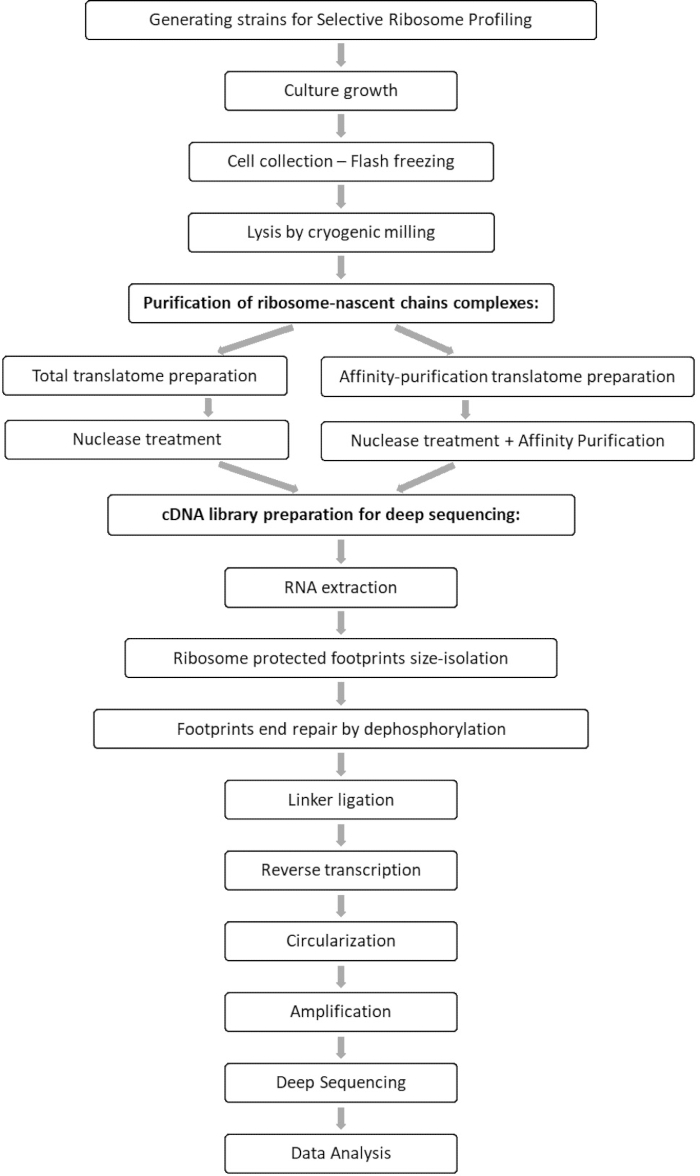
איור 1: סקירה כללית של פרוטוקול SeRP. פרוטוקול זה יכול להתבצע בשלמותו תוך 7-10 ימים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
Protocol
Representative Results
Discussion
כאן, הפרוטוקול מפרט את גישת פרופיל הריבוזומים הסלקטיבית ללכידת אינטראקציות של תרגום משותף ברזולוציה של כמעט קודון. ככל שהריבוזום עולה כמרכז לתיאום הופעת השרשרת המתהווה לתוך הציטופלסמה הצפופה, זוהי שיטה חיונית לזיהוי ואפיון האינטראקציות השונות של התרגום המשותף הנדרשות כדי להבטיח פרוטא?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
ברצוננו להודות לכל חברי המעבדה על הדיונים הפוריים ולמוחמד מחזומי על הקריאה הביקורתית בכתב היד. עבודה זו מומנה על ידי מענק ISF (הקרן הישראלית למדע) 2106/20.
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
References
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

