Global identifiering av saminterventionella interaktionsnätverk genom selektiv ribosomprofilering
Summary
Samöversättningsinteraktioner spelar en avgörande roll i begynnande kedjemodifieringar, inriktnings-, viknings- och monteringsvägar. Här beskriver vi Selektiv ribosomprofilering, en metod för in vivo, direkt analys av dessa interaktioner i modellen eukaryot Saccharomyces cerevisiae.
Abstract
Under de senaste åren har det blivit uppenbart att ribosomer inte bara avkodar vårt mRNA utan också styr framväxten av polypeptidkedjan i den trånga cellulära miljön. Ribosomer ger plattformen för rumsligt och kinetiskt kontrollerad bindning av membraninriktningsfaktorer, modifierande enzymer och vikbara chaperoner. Även sammansättningen i högordnade oligomera komplex, såväl som protein-proteinnätverksbildningssteg, upptäcktes nyligen samordnas med syntes.
Här beskriver vi Selective Ribosome Profiling, en metod utvecklad för att fånga sam-translationella interaktioner in vivo. Vi kommer att beskriva de olika affinitetsreningsstegen som krävs för att fånga ribosom-begynnande kedjekomplex tillsammans med sam-translationella interagerare, liksom mRNA-extraktion, storleksuteslutning, omvänd transkription, djupsekvensering och big-data-analyssteg, som krävs för att dechiffrera sam-translationella interaktioner i nära kodonupplösning.
Introduction
Selective Ribosome Profiling (SeRP) är den enda metoden hittills som fångar och karakteriserar sam-translationella interaktioner, in vivo, på ett direkt sätt 1,2,3,4,5,6. SeRP möjliggör global profilering av interaktioner mellan vilken faktor som helst med översättning av ribosomer i nära kodonupplösning 2,7.
Metoden bygger på blixtfrysning av växande celler och bevarande av aktiv översättning. Celllysater behandlas sedan med RNas I för att smälta allt mRNA i cellen utom ribosomskyddade mRNA-fragment som kallas “ribosomavtryck”. Provet delas sedan upp i två delar; en del används direkt för isolering av alla cellulära ribosomala fotavtryck, vilket representerar all pågående översättning i cellen. Den andra delen används för affinitetsrening av den specifika delmängden ribosomer associerade med en faktor av intresse, till exempel: modifierande enzymer, translokationsfaktorer, vikbara chaperoner och komplexa monteringsinteraktioner. De affinitetsrenade ribosomala fotavtrycken kallas kollektivt interaktionomet. Därefter extraheras de ribosomskyddade mRNA och används för cDNA-biblioteksgenerering, följt av djup sekvensering.
Jämförande analys av de totala translatom- och interaktionsproverna möjliggör identifiering av alla orfs som associerar med intressefaktorn, samt karakterisering av varje orf-interaktionsprofil. Denna profil rapporterar de exakta engagemangsstart- och avslutningssekvenserna från vilka man kan härleda de avkodade kodonerna och respektive rester av den framväxande polypeptidkedjan, liksom på ribosomhastighetsvariationerna under interaktionen 7,8. Figur 1 visar protokollet som ett schema.
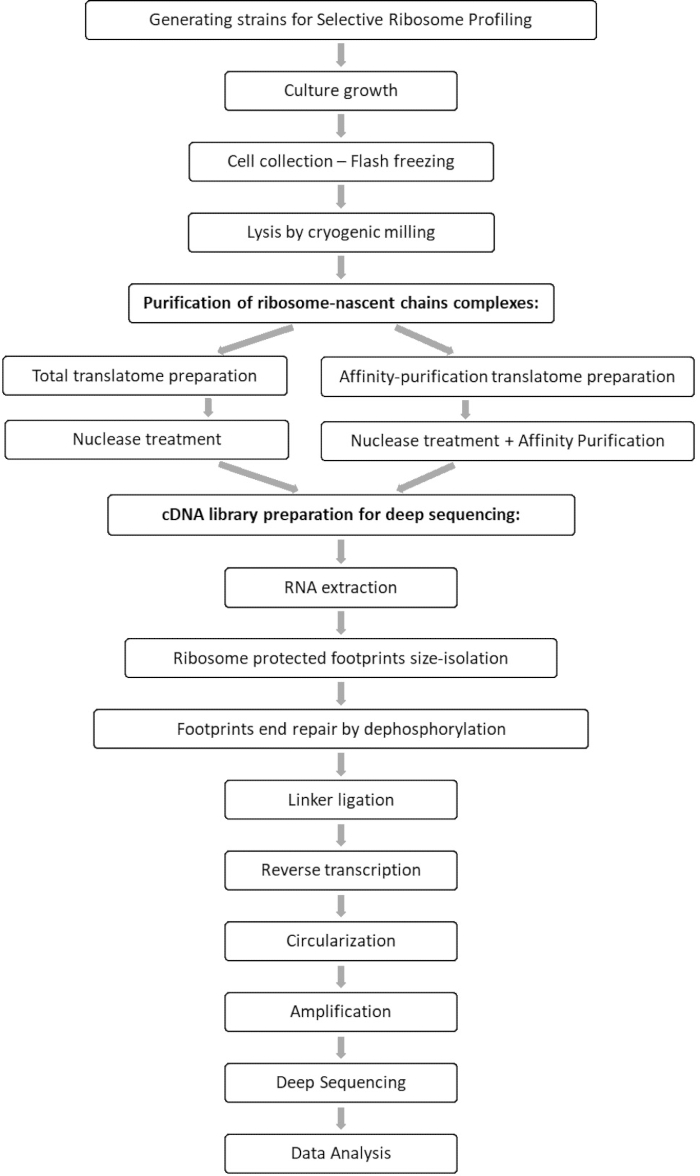
Bild 1: En översikt över SeRP-protokollet. Detta protokoll kan utföras i sin helhet inom 7-10 dagar. Klicka här för att se en större version av denna siffra.
Protocol
Representative Results
Discussion
Här beskriver protokollet den selektiva ribosomprofileringsmetoden för att fånga samöversättningsinteraktioner i nära kodonupplösning. När ribosomen stiger som ett nav för att samordna den framväxande kedjans uppkomst i den trånga cytoplasman, är detta en avgörande metod för att identifiera och karakterisera de olika sam-translationella interaktioner som krävs för att säkerställa ett funktionellt proteom, liksom för att studera olika sjukdomar. Hittills är SeRP den enda metoden som kan fånga och kar…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Vi vill tacka alla labbmedlemmar för givande diskussioner och Muhammad Makhzumy för den kritiska läsningen av manuskriptet. Detta arbete finansierades av ISF (Israeli Science Foundation) bidrag 2106/20.
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
References
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

