Bakteriel ekspression og oprensning af human matrixmetalloproteinase-3 ved anvendelse af affinitetskromatografi
Summary
His-tag oprensning, dialyse og aktivering anvendes til at øge udbyttet af opløselig, aktiv matrix metalloproteinase-3 katalytisk domæneproteinekspression i bakterier. Proteinfraktioner analyseres via SDS-PAGE geler.
Abstract
Matrixmetalloproteinaser (MMP’er) tilhører familien af metzincinproteaser med centrale roller i ekstracellulær matrix (ECM) nedbrydning og ombygning samt interaktioner med flere vækstfaktorer og cytokiner. Overekspression af specifikke MMP’er er ansvarlig for flere sygdomme såsom kræft, neurodegenerative sygdomme og hjerte-kar-sygdomme. MMP’er har været centrum for opmærksomhed for nylig som mål for at udvikle terapi, der kan behandle sygdomme, der er korreleret med MMP-overekspression.
For at undersøge MMP-mekanismen i opløsning er der behov for mere letkøbte og robuste rekombinante proteinekspressions- og oprensningsmetoder til fremstilling af aktive, opløselige MMP’er. Det katalytiske domæne for de fleste MMP’er kan imidlertid ikke udtrykkes i Escherichia coli (E. coli) i opløselig form på grund af mangel på posttranslationsmaskiner, mens pattedyrs ekspressionssystemer normalt er dyre og har lavere udbytter. MMP-inklusionsorganer skal gennemgå den kedelige og besværlige proces med omfattende oprensning og omfoldning, hvilket reducerer udbyttet af MMP’er i indfødt konformation betydeligt. Dette papir præsenterer en protokol, der bruger Rosetta2 (DE3) pLysS (i det følgende benævnt R2DP) celler til fremstilling af matrixmetalloproteinase-3 katalytisk domæne (MMP-3cd), som indeholder et N-terminal His-tag efterfulgt af pro-domæne (Hisx6-pro-MMP-3cd) til brug i affinitetsrensning. R2DP-celler forbedrer ekspressionen af eukaryote proteiner gennem et chloramphenicolresistent plasmid indeholdende kodoner, der normalt er sjældne i bakterielle ekspressionssystemer. Sammenlignet med den traditionelle cellelinje til valg af rekombinant proteinekspression, BL21 (DE3), forbedrede oprensning ved hjælp af denne nye stamme udbyttet af renset Hisx6-pro-MMP-3cd. Ved aktivering og afsaltning spaltes pro-domænet sammen med N-terminal His-tagget, hvilket giver aktiv MMP-3cd til øjeblikkelig brug i utallige in vitro-applikationer . Denne metode kræver ikke dyrt udstyr eller komplekse fusionsproteiner og beskriver hurtig produktion af rekombinante humane MMP’er i bakterier.
Introduction
De fleste komplekse eukaryote proteiner gennemgår udførlige posttranslationelle modifikationer efter ekspression, hvilket kræver stærkt assisteret proteinfoldning og co-faktorer for at være funktionelle1. Produktion af store mængder opløseligt humant protein i en bakterievært er fortsat en betydelig udfordring på grund af høje omkostninger og manglen på robuste ekspressions- og oprensningsmetoder, selv for laboratorieforsøg i mindre skala2,3. MMP’er, humane endopeptidaser med stor molekylvægt, udtrykkes normalt som uopløselige inklusionslegemer, når de udtrykkes i E. coli. Ekstraktion af opløselige humane MMP’er fører ofte til en besværlig, tidskrævende opløseligheds- og genfoldningsproces4.
MMP’er har kritiske roller i både fysiologiske og patogene processer. Humane MMP’er er en familie af 23 zinkendopeptidaser, kategoriseret efter struktur og substratspecikalitet og forskelligt udtrykt på trods af et stærkt bevaret katalytisk domæne5,6. MMP’er udskilles som inaktive zymogener, reguleret via posttranslationel aktivering og deres endogene hæmmere, vævshæmmere af metalloproteinaser (TIMP’er)7,8,9,10. Selvom MMP’er oprindeligt blev anerkendt for deres rolle i ECM-omsætningen, har de også været impliceret i udvikling, morfogenese, vævsreparation og ombygning8. Dysregulering af MMP’er har især været forbundet med kræft sammen med neurodegenerative, kardiovaskulære og fibrotiske sygdomme, blandt andre sygdomme5,7.
Udviklingen af robuste MMP-produktionsmetoder i stor skala er afgørende for at sikre succesen med fremtidige undersøgelser af MMP-mekanismer gennem biokemiske og cellebaserede assays. Forskellige MMP’er er tidligere blevet udtrykt i bakterier11, herunder Hisx6-mærkede MMP’er, uden at ændre MMP-aktivitet12,13,14,15. Disse metoder inkluderer dog kedelige, lange trin, der kan være vanskelige at replikere.
Pattedyrceller kan også bruges til at udtrykke mange forskellige humane proteiner, samtidig med at de sikrer de korrekte posttranslationelle modifikationer16. Selv om pattedyrs ekspressionssystem er et ideelt valg til fremstilling af rekombinante humane proteiner med korrekte postoverensstemmende modifikationer, er de største ulemper ved denne metode indledende lave udbytter, dyre vækstmedier og reagenser, lange tidslinjer for at nå stabile ekspressionslinjer og risiko for forurening med andre arter såsom svampe eller bakterier2,11 . Desuden giver MMP-produktion i pattedyrs cellelinjer urenheder fra associerede cellulære proteiner såsom TIMP’er eller fibronectiner11. I modsætning til den langsomme cellevækst, der observeres i pattedyrceller, tilbyder bakterieekspressionssystemet storskala proteinproduktion på kort tid sammen med enklere medie- og vækstkrav. På grund af manglen på andre associerede cellulære proteiner (dvs. TIMP’er) i bakterielle ekspressionssystemer udsættes aktive MMP’er i højere koncentrationer imidlertid for nedbrydning gennem autoproteolyse, hvilket resulterer i dårligt MMP-udbytte17.
Dette papir beskriver en detaljeret metode til bakteriel ekspression, oprensning og aktivering af rekombinant Hisx6-pro-MMP-3cd ved hjælp af E. coli som ekspressionsvært på grund af dets overkommelige priser, enkelhed og succes med at producere højere udbytter af MMP’er2,3,18. Da E. coli mangler proteinfoldemaskineri og posttranslationel behandling, der kræves til rekombinante MMP’er og andre komplekse proteiner, er mange E. coli-stammer blevet konstrueret til at overvinde disse begrænsninger, hvilket gør E. coli til en mere egnet vært til ekspression af rekombinant human MMP-3cd,19,20 . For eksempel forbedrer R2DP-stammen, der anvendes i denne undersøgelse, eukaryot ekspression ved at levere et chloramphenicolresistent plasmid indeholdende kodoner, der sjældent anvendes i E. coli.
Som beskrevet i denne protokol ekstraheres hisx6-pro-MMP-3 katalytiske domæneproteiner (MMP-3cd) efter overekspression af relativt rene inklusionslegemer fra pET-3a-vektoren (figur 1) i R2DP-celler4. Hisx6-pro-MMP-3cd3,19 blev renset ved hjælp af affinitetsmærkekromatografi. Ved omfoldning og dialyse blev pro-MMP-3cd (zymogen) aktiveret af 4-aminophenylmercuric acetat (APMA), og SDS-PAGE-analyse anvendes til at evaluere udbytter og behovet for yderligere oprensning5,21. Denne protokol beskriver ekspression, oprensning og aktivering af opløselig mMP-3cd som et eksempel. Det kan dog også anvendes som vejledning til ekspression af andre MMP’er og humane proteaser med lignende ekspression og aktiveringsmekanismer (figur 2). For andre proteiner end MMP-3cd rådes læseren til at bestemme optimale buffersammensætninger og metoder til deres målprotein, inden denne protokol forsøges.
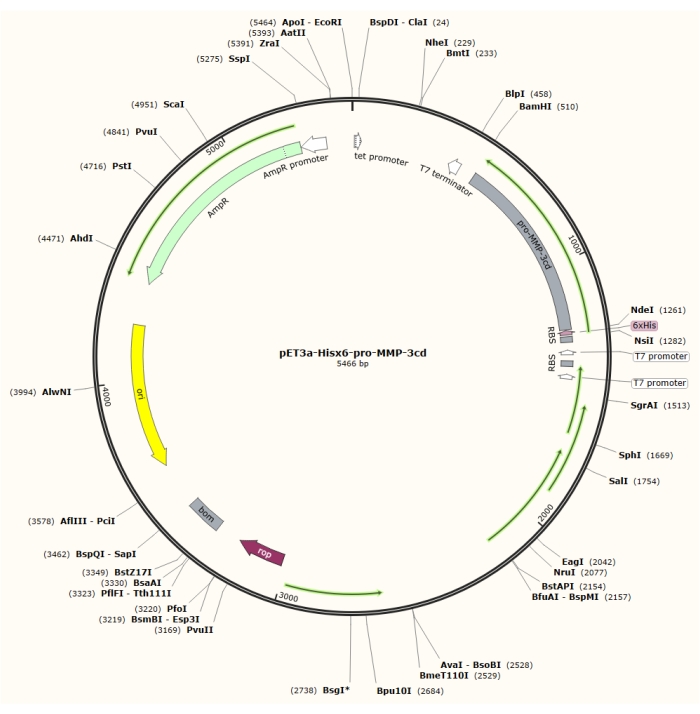
Figur 1: Plasmidkort over pET-3a-Hisx6-pro-MMP-3cd-plasmidet. PET-3a-vektoren indbefatter et ampicillinresistensgen. En N-terminal Hisx6-tag-sekvens klones ind i den pET-3a-baserede vektor, herunder pro-MMP-3cd, for at give pET-3a-Hisx6-pro-MMP-3cd-konstruktionen under kontrol af T7-promotor mellem BamHI- og NdeI-begrænsningssteder. Klik her for at se en større version af denne figur.
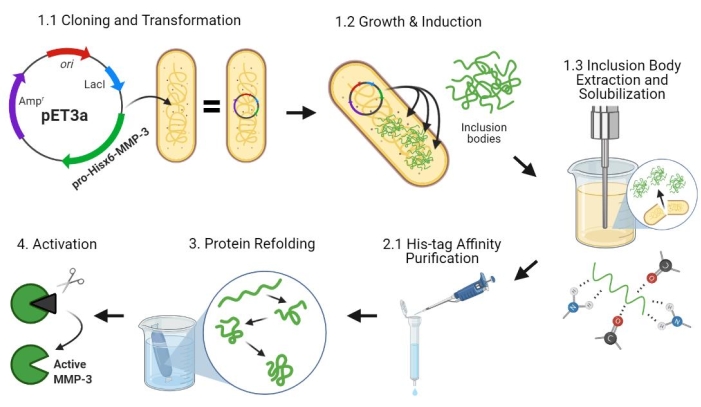
Figur 2: Bakteriel ekspression af pro-MMP-3cd, oprensning, omfoldning og aktivering. 1.1: pET-3a-Hisx6-pro-MMP-3cd plasmid blev omdannet til BL21 (DE3) eller R2DP-celler. 1.2: Pro-MMP-3cd-proteinekspression blev induceret under anvendelse af IPTG. 1.3: Kemisk lysis og sonikering bruges til at ekstrahere Hisx6-pro-MMP-3cd-proteiner, der hovedsageligt er uopløselige og findes i inklusionsorganerne. Urea blev brugt til at denaturere og opløse protein fra inklusionslegemer. 2.1. Denatureret Hisx6-pro-MMP-3cd-protein blev oprenset via affinitetskromatografirensning. 3. Den eluerede Hisx6-pro-MMP-3cd blev langsomt foldet om under dialyse gennem gradvis fjernelse af urinstof fra bufferen. 4. Endelig blev genfoldet MMP-3cd-protein aktiveret ved hjælp af APMA ved at fjerne det N-terminale propeptiddomæne. APMA fjernes senere fra opløsningen ved afsaltning. Tallene svarer til protokolafsnit, der beskriver disse trin. Forkortelser: MMP-3cd = Matrix metalloproteinase-3 katalytisk domæne; APMA = 4-aminophenylmercuric acetat. Klik her for at se en større version af denne figur.
Protocol
Representative Results
Discussion
Den storstilede produktion af opløselige, humane, rekombinante MMP’er er fortsat en udfordrende opgave. Pattedyrceller kan udtrykke funktionelle MMP’er til høje omkostninger og lange ventetider, mens E. coli hurtigt producerer store mængder MMP-inklusionsorganer, der skal renses og genfoldes11,16. R2DP-celler øger udbyttet af MMP-inklusionsorganer betydeligt, hvilket muliggør en mere omkostningseffektiv og produktiv MMP-omfoldningsproces. E. col…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Forfatterne vil gerne anerkende Dr. Evette Radisky og Alexandra Hockla på Mayo Clinic i Jacksonville, Florida, for at have leveret pET-3a-pro-MMP-3cd-plasmidet som skabelon til kloning af Hisx6pro-MMP-3cd-genet og deres kommentarer sammen med Dr. Paul Hartley fra Nevada Genomics Center ved University of Nevada, Reno, til DNA-sekventering. Forfatterne vil også gerne takke Cassandra Hergenrader for at hjælpe med en del af proteinekspressionen. M.R.-S. vil gerne takke NIH-P20 GM103650-COBRE Integrative Neuroscience-bevillingen og UNR R&D mICRO SEED Grant Award.
Materials
| 0.22 µm sterile filter | Sigma Aldrich | SLGP033RS | Used to remove some contaminants from the protein extract before purification, and prevent the Ni-NTA column from clogging |
| 1 L Erlenmeyer flasks | Thermo Fisher Scientific | S76106F | n/a |
| 1 L glass bottles | Thermo Fisher Scientific | 06-414-1D | n/a |
| 1.5 mL microfuge tubes | Thermo Fisher Scientific | 02-682-002 | n/a |
| 15 mL conical tubes | Thermo Fisher Scientific | 339650 | n/a |
| 18 G, 1-in. beveled needle | Amazon | B07S7VBHM2 | Used in combination with the dialysis casette |
| 2 mL desalting column | Thermo Fisher Scientific | 89890 | Removes APMA following activation |
| 2-(N-Morpholino)ethanesulfonic acid (MES) | Thermo Fisher Scientific | AAA1610422 | n/a |
| 250 mL conical bottle cushions | Thermo Fisher Scientific | 05-538-53A | Stabilize conical bottles during large-volume centrifugation |
| 250 mL conical bottles | Thermo Fisher Scientific | 05-538-53 | n/a |
| 400 mL stirred cell | Sigma Aldrich | UFSC40001 | Re-concentrates a much larger volume than the centrifugal filter unit. Rosetta2(DE3)pLysS cells produce high volumes of protein that may exceed the 15 mL limit of the centrifugal filter unit |
| 4-aminophenylmercuric acetate (APMA) | Sigma Aldrich | A9563-5G | Activates MMP-3 by cleaving the propeptide |
| 5 mL syringe | Thermo Fisher Scientific | NC0829167 | Used in combination with the dialysis casette |
| 50 mL conical tubes | Thermo Fisher Scientific | 339650 | Used for storage in many purification steps |
| 50 mL re-concentration tube | Sigma Aldrich | UFC901024D | Used for re-concentrating protein samples after dialysis or removing contaminants |
| Agar | Thermo Fisher Scientific | BP1423-500 | Buffer ingredient that solidifies autoclaved LB media upon cooling |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | Antibiotic used with pET3a vector; used at 100 µg/mL in LB media |
| BamHI | NEB | R3136S | Restriction enzyme to be used with the pET3a vector |
| Calcium chloride (CaCl2) | Thermo Fisher Scientific | 600-30-23 | The calcium ion stabilizes MMP structure |
| Cell spreaders | Thermo Fisher Scientific | 50-189-7544 | Can be used to spread cells across a petri dish after transformation |
| Chloramphenicol | Thermo Fisher Scientific | 22-055-125GM | Antibiotic used with pET3a vector; used at 34 µg/mL in LB media |
| Dialysis Buffer 1 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 4 M Urea. |
| Dialysis Buffer 2 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 2 M Urea. |
| Dialysis Buffer 3 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2 , 1 µM ZnCl2. |
| Dialysis clips | Thermo Fisher Scientific | 68011 | Used in combination with snakeskin dialysis tubing |
| Dialysis tubing | Thermo Fisher Scientific | 88243 | Alternative dialysis method that holds much larger sample volumes, but with higher risk of sample loss |
| Digest buffer | NEB | B7204S | Buffer used in digesting the pET3a vector |
| Disposable cuvettes | Thermo Fisher Scientific | 21-200-257 | Used to measure the bacterial culture OD during growth and expression |
| Dithiothreitol (DTT) | Thermo Fisher Scientific | D107125G | Assists with protein denaturation by reducing any disulfide bonds |
| DNA assembly mix | NEB | E2621S | Used to ligate the Hisx6-pro-MMP-3cd PCR product and digested pET3a vector |
| DNase I | NEB | M0303S | Endonuclease for degrading unfavorable DNA contaminants that could later affect protein purification |
| Ethanol | Thermo Fisher Scientific | A995-4 | n/a |
| Ethylenediaminetetraacetic acid (EDTA) | Thermo Fisher Scientific | J15694-AE | Used in denaturation. Prevents oxidation and subsequent formation of disulfide bonds |
| Gel recovery kit | Promega | A9281 | Isolates and purifies DNA from agarose gels |
| Glycerol | Thermo Fisher Scientific | G33-500 | Used for making glycerol stocks, which are frozen at -80 °C |
| Gravity flow column | BioRad | 7321010 | Used for Ni-NTA purification of recombinantly His-tagged proteins |
| Guanidine hydrochloride (GdnHCl) | Thermo Fisher Scientific | AAA135430B | Second chaotropic agent used for disrupting protein secondary structure. |
| High-transformation efficiency cells | NEB | C2987 | High-transformation efficiency cells with greater chance of success for cloning the N-terminal His-tag into the pET3a-pro-MMP-3cd construct |
| HT Elution Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 250 mM imidazole. Adjust pH to 7.4 |
| HT Equilibration Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea. Adjust pH to 7.4 |
| HT Regeneration Buffer | n/a | n/a | 20 mM MES, 0.1 M NaCl. Adjust pH to 5.0 |
| HT Wash Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 25 mM imidazole. Adjust pH to 7.4 |
| Hydrochloric acid (HCl) | Thermo Fisher Scientific | A144C-212 | Used to pH buffers |
| Imidazole | Thermo Fisher Scientific | AAA1022122 | Mimics the histidine side group. Used to separate non-specifically binding proteins from the his-tagged target protein |
| Inclusion Body Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 5 mM DTT, 2% v/v Triton X 100, 0.5 M Urea. Adjust pH to 8.0 |
| Isopropyl-ß-D-thiogalactopyranoside (IPTG) | Thermo Fisher Scientific | FERR0392 | A reagent that induces target gene expression in pET3a. Make 0.5 mL 1 M aliquots, filter sterilize and store in -20 °C |
| LB Amp CamR media | n/a | n/a | To be poured into a sterible 1 L bottle or 1 L flask. For 1 L, add 25 g LB Broth. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Amp CamR plates | n/a | n/a | To be poured into sterile petri dishes. Pour until the petri dish lid is completely covered. 1 L of media yields 40-60 plates. For 1 L: 25 g LB Broth, 16 g Agar. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Broth | Thermo Fisher Scientific | BP1426-2 | Pre-mixed with tryptone, yeast extract, and sodium chloride |
| Lysis Buffer | n/a | n/a | 50 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 0.133 g/mL lysozyme, 0.49% v/v Triton X-100. Adjust pH to 8.0 |
| Lysozyme | MP Biomedicals | 195303 | Used in protein extraction. Enzyme that lyses bacterial cell walls |
| Miniprep kit | Promega | A1330 | If successful, extracts the pET3a-pro-MMP-3cd construct from transformants |
| NdeI | NEB | R0111S | Restriction enzyme to be used with the pET3a vector |
| Ni-NTA resin | Thermo Fisher Scientific | PI88221 | Used to bind recombinant his-tagged proteins. This strong interaction can be displaced with higher concentrations of imidazole |
| PCR mix | NEB | M0492S | A PCR reagent for inserting an N-terminal his-tag into the pET3a-pro-MMP-3cd vector |
| pET plasmid | Addgene | n/a | The pET3a vector offers ampicillin resistance, inducible expression of a target gene, and sequencing with T7 primers |
| Petri dishes | VWR | 25384-342 | Used for plating transformants on LB agar media |
| R2DP cells | Novagen | 714033 | BL21 derivatives with enhanced expression of eukaryotic proteins. Contain tRNAs of codons found to be rare in e. coli |
| SOC growth media | NEB | B9020S | Non-selective growth media for rapid growth during transformation |
| Sodium chloride (NaCl) | Thermo Fisher Scientific | BP358-1 | Used in buffers and helps with protein stability |
| Sodium deoxycholate | Thermo Fisher Scientific | PI89905 | Detergent used in protein extraction. Lyses cell walls |
| Solubilization Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 10 mM DTT, 6 M Urea. Adjust pH to 8.0 |
| Tris base | Thermo Fisher Scientific | BP152-1 | Common buffer used in the physiological pH range. Temperature-sensitive |
| Triton X-100 | Thermo Fisher Scientific | M1122980101 | Detergent used for cell lysis |
| Urea | Thermo Fisher Scientific | AAJ75826A7 | First chaotropic agent for disrupting protein secondary structure |
| Zinc chloride (ZnCl2) | Thermo Fisher Scientific | AAA162810E | Stabilizes MMP structure. The zinc ion is found in the catalytic site of MMP-3 |
References
- Portolano, N., et al. Recombinant protein expression for structural biology in HEK 293F suspension cells: A novel and accessible approach. Journal of Visualized Experiments: JoVE. (92), e51897 (2014).
- Subedi, G. P., Johnson, R. W., Moniz, H. A., Moremen, K. W., Barb, A. High yield expression of recombinant human proteins with the transient transfection of HEK293 cells in suspension. Journal of Visualized Experiments: JoVE. (106), e53568 (2015).
- Nilvebrant, J., Alm, T., Hober, S. Orthogonal protein purification facilitated by a small bispecific affinity tag. Journal of Visualized Experiments: JoVE. (59), e3370 (2012).
- Yang, Z., et al. Highly efficient production of soluble proteins from insoluble inclusion bodies by a two-step-denaturing and refolding method. PLoS One. 6 (7), 22981 (2011).
- Hu, X., Beeton, C. Detection of functional matrix metalloproteinases by zymography. Journal of Visualized Experiments: JoVE. (45), e2445 (2010).
- Radisky, E. S., Raeeszadeh-Sarmazdeh, M., Radisky, D. C. Therapeutic potential of matrix metalloproteinase inhibition in breast cancer. Journal of Cellular Biochemistry. 118 (11), 3531-3548 (2017).
- Raeeszadeh-Sarmazdeh, M., Do, L. D., Hritz, B. G. Metalloproteinases and their inhibitors: Potential for the development of new therapeutics. Cells. 9 (5), 1313 (2020).
- Nagase, H., Visse, R., Murphy, G. Structure and function of matrix metalloproteinases and TIMPs. Cardiovascular Research. 69 (3), 562-573 (2006).
- Raeeszadeh-Sarmazdeh, M., et al. Directed evolution of the metalloproteinase inhibitor TIMP-1 reveals that its N- and C-terminal domains cooperate in matrix metalloproteinase recognition. Journal of Biological Chemistry. 294 (24), 9476-9488 (2019).
- Batra, J., et al. Matrix metalloproteinase-10 (MMP-10) interaction with tissue inhibitors of metalloproteinases TIMP-1 and TIMP-2. Journal of Biological Chemistry. 287 (19), 15935-15946 (2012).
- Singh, K. K., Jain, R., Ramanan, H., Saini, D. K., Galea, C. A. Expression and purification of matrix metalloproteinases in Escherichia coli. Matrix Metalloproteases. , 3-16 (2017).
- Manka, S. W., et al. Structural insights into triple-helical collagen cleavage by matrix metalloproteinase 1. Proceedings of the National Academy of Sciences of the United States of America. 109 (31), 12461-12466 (2012).
- Gomis-Ruth, F. X., et al. Mechanism of inhibition of the human matrix metalloproteinase stromelysin-1 by TIMP-1. Nature. 389 (6646), 77-81 (1997).
- Shirian, J., et al. Converting a broad matrix metalloproteinase family inhibitor into a specific inhibitor of MMP-9 and MMP-14. FEBS Letters. 592 (7), 1122-1134 (2018).
- Li, C., et al. Purification of recombinant histidine-tagged catalytic domain of MMP-13 in one step using affinity column and renaturation of it with histidine tag. Journal of Liquid Chromatography & Related Technologies. 37 (15), 2118-2130 (2014).
- Aydin, H., Azimi, F. C., Cook, J. D., Lee, J. E. A convenient and general expression platform for the production of secreted proteins from human cells. Journal of Visualized Experiments: JoVE. (65), e4041 (2012).
- McNiff, M. L., Haynes, E. P., Dixit, N., Gao, F. P., Laurence, J. S. Thioredoxin fusion construct enables high-yield production of soluble, active matrix metalloproteinase-8 (MMP-8) in Escherichia coli. Protein Expression and Purification. 122, 64-71 (2016).
- Maity, R., et al. GST-His purification: A two-step affinity purification protocol yielding full-length purified proteins. Journal of Visualized Experiments: JoVE. (80), e50320 (2013).
- Stefan, A., Ceccarelli, A., Conte, E., Montón Silva, A., Hochkoeppler, A. The multifaceted benefits of protein co-expression in Escherichia coli. Journal of Visualized Experiments: JoVE. (96), e52431 (2015).
- Yadavalli, R., Sam-Yellowe, T. HeLa based cell free expression systems for expression of Plasmodium rhoptry proteins. Journal of Visualized Experiments: JoVE. (100), e52772 (2015).
- Zeytuni, N., Zarivach, R. Purification of the M. magneticum strain AMB-1 magnetosome associated protein MamAΔ41. Journal of Visualized Experiments: JoVE. (37), e1844 (2010).

