Lichtgesteuerte Fermentationen für die mikrobielle Chemikalien- und Proteinproduktion
Summary
Die optogenetische Kontrolle des mikrobiellen Stoffwechsels bietet eine flexible dynamische Kontrolle über Fermentationsprozesse. Das Protokoll zeigt hier, wie man blaulichtregulierte Fermentationen für die chemische und Proteinproduktion auf verschiedenen volumetrischen Skalen aufbaut.
Abstract
Mikrobielle Zellfabriken bieten eine nachhaltige Alternative zur Herstellung von Chemikalien und rekombinanten Proteinen aus nachwachsenden Rohstoffen. Die Überlastung eines Mikroorganismus mit genetischen Veränderungen kann jedoch die Fitness und Produktivität des Wirts verringern. Dieses Problem kann durch dynamische Kontrolle überwunden werden: induzierbare Expression von Enzymen und Signalwegen, typischerweise unter Verwendung von chemischen oder nährstoffbasierten Zusätzen, um das Zellwachstum und die Zellproduktion auszugleichen. Die Optogenetik bietet eine nicht-invasive, hochgradig abstimmbare und reversible Methode zur dynamischen Regulierung der Genexpression. Hier beschreiben wir, wie man lichtgesteuerte Fermentationen von technisch hergestellten Escherichia coli und Saccharomyces cerevisiae zur Herstellung von Chemikalien oder rekombinanten Proteinen aufbaut. Wir diskutieren, wie Licht zu ausgewählten Zeiten und Dosierungen angewendet werden kann, um mikrobielles Wachstum und Produktion für eine verbesserte Fermentationskontrolle und Produktivität zu entkoppeln, sowie die wichtigsten Optimierungsüberlegungen für beste Ergebnisse. Darüber hinaus beschreiben wir, wie Lichtsteuerungen für Bioreaktorexperimente im Labormaßstab implementiert werden können. Diese Protokolle erleichtern die Einführung optogenetischer Kontrollen in technisch hergestellten Mikroorganismen zur Verbesserung der Fermentationsleistung.
Introduction
Die Optogenetik, die Steuerung biologischer Prozesse mit lichtempfindlichen Proteinen, bietet eine neue Strategie zur dynamischen Steuerung mikrobieller Fermentationen für die chemische und Proteinproduktion1,2. Die Belastung durch technische Stoffwechselwege und die Toxizität einiger Zwischenprodukte und Produkte beeinträchtigen häufig das Zellwachstum3. Solche Belastungen können zu einer schlechten Biomasseakkumulation und einer verminderten Produktivität führen3. Dieser Herausforderung kann begegnet werden, indem die Fermentationen zeitlich in eine Wachstums- und Produktionsphase unterteilt werden, die metabolische Ressourcen der Biomasseakkumulation bzw. Produktsynthese widmen4. Wir haben kürzlich gezeigt, dass der Übergang vom Wachstum zur Produktion in dieser zweiphasigen Fermentation mit Änderungen der Beleuchtungsbedingungen induziert werden kann5,6,7. Die hohe Abstimmbarkeit, Reversibilität und Orthogonalität von Lichteinträgen8 bieten einzigartige Vorteile für lichtgesteuerte Fermentationen, die mit chemischen Induktoren, die zur dynamischen Steuerung herkömmlicher Zweiphasenfermentationen eingesetzt werden, schwer oder gar nicht repliziert werden können4,9,10,11.
Das blaulichtempfindliche Protein EL222, das aus Erythrobacter litoralis gewonnen wird, wurde verwendet, um mehrere optogenetische Schaltkreise für das Metabolic Engineering in Saccharomyces cerevisiae5,7,12,13 zu entwickeln. EL222 enthält eine LOV-Domäne (Light-Oxygen-Voltage-Sensor), die bei Blaulichtaktivierung (465 nm) eine Konformationsverschiebung erfährt, wodurch sie an ihre verwandte DNA-Sequenz (C120)13 binden kann. Die Fusion von EL222 mit der viralen VP16-Aktivierungsdomäne (VP16-EL222) führt zu einem auf Blaulicht reagierenden Transkriptionsfaktor, der die Genexpression in S. cerevisiae7 und anderen Organismen14 aus dem synthetischen Promotor PC120 reversibel aktivieren kann. Mehrere Schaltkreise, die auf EL222 basieren, wurden für die chemische Produktion in S. cerevisiae entwickelt und verwendet, wie das grundlegende lichtaktivierte OptoEXP-System7, in dem das Gen von Interesse direkt aus PC120 exprimiert wird (Abbildung 1A). Bedenken hinsichtlich der Lichtdurchdringung bei den hohen Zelldichten, die typischerweise in der Produktionsphase von Fermentationen auftreten, motivierten uns jedoch, invertierte Schaltkreise zu entwickeln, die im Dunkeln induziert werden, wie die OptoINVRT- und OptoQ-INVRT-Schaltungen (Abbildung 1B) 5,7,13. Diese Systeme nutzen die Galactose- (GAL) oder Chinainsäure (Q)-Regulone aus S. cerevisiae bzw. N. crassa und kontrollieren ihre entsprechenden Repressoren (GAL80 und QS) mit VP16-EL222, um die Genexpression im Licht zu unterdrücken und im Dunkeln stark zu induzieren. Die Kombination von OptoEXP- und OptoINVRT-Schaltkreisen führt zu einer bidirektionalen Kontrolle der Genexpression und ermöglicht zweiphasige Fermentationen, bei denen die Wachstumsphase mit blauem Licht und die Produktionsphase mit Dunkelheit induziert wird (Abbildung 2A) 5,7.
Die Verwendung von Licht anstelle von Dunkelheit zur Induktion der Genexpression während der Produktionsphase würde die Fähigkeiten der optogenetischen Kontrollen erheblich erweitern, würde aber auch die Überwindung der Lichtdurchdringungsbeschränkungen der hohen Zelldichten erfordern, die typischerweise in dieser Phase der Fermentation auftreten. Zu diesem Zweck haben wir Schaltkreise entwickelt, die als OptoAMP und OptoQ-AMP bekannt sind und die transkriptionelle Reaktion auf die Blaulichtstimulation verstärken. Diese Schaltkreise verwenden Wildtyp- oder überempfindliche Mutanten von VP16-EL222, um die Produktion der transkriptionellen Aktivatoren Gal4p oder QF2 der GAL- bzw. Q-Regulonen zu kontrollieren und eine erhöhte Empfindlichkeit und stärkere Genexpression mit Licht zu erreichen12,13 (Abbildung 1C). OptoAMP-Schaltungen können eine vollständige und homogene Lichtinduktion in 5-L-Bioreaktoren bei einer optischen Dichte (gemessen bei 600 nm; OD600) Werte von mindestens 40 mit nur ~0,35% der Beleuchtung (5% Lichtdosis auf nur ~7% der Schüttfläche). Dies zeigt eine höhere Empfindlichkeit im Vergleich zu OptoEXP, das eine Beleuchtung von nahezu 100% erfordert12. Die Fähigkeit, die Genexpression mit Licht bei hohen Zelldichten effektiv zu induzieren, eröffnet neue Möglichkeiten zur dynamischen Steuerung von Fermentationen. Dazu gehören Betriebsfermentationen in mehr als zwei zeitlichen Phasen, wie z. B. dreiphasige Fermentationen, bei denen Wachstums-, Induktions- und Produktionsphasen mit einzigartigen Lichtplänen zur Optimierung der chemischen Produktion festgelegt werden (Abbildung 2B)12.
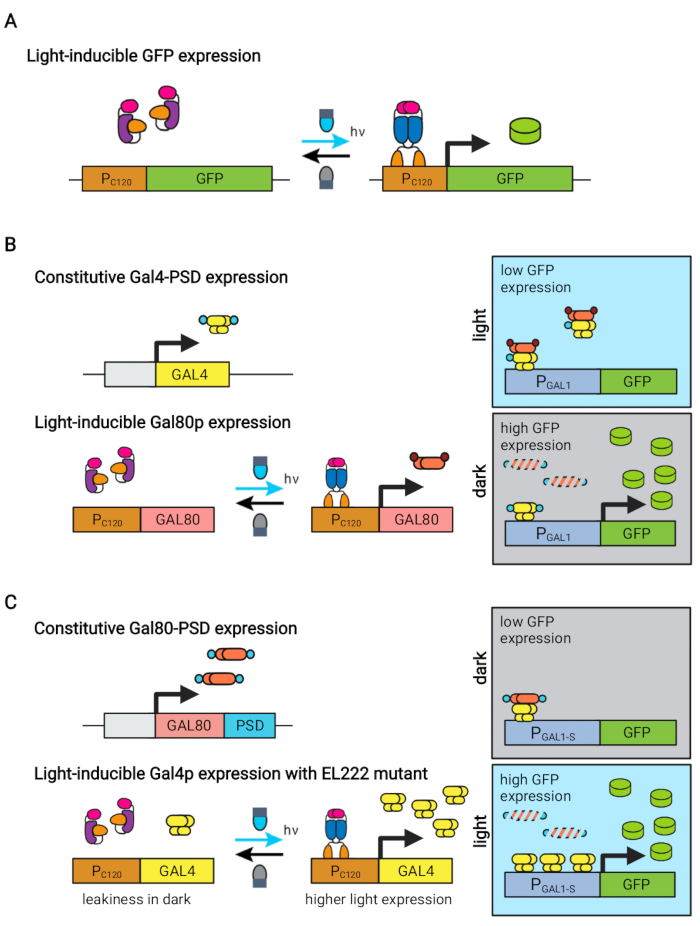
Abbildung 1: Optogenetische Schaltkreise zur dynamischen Regelung von S. cerevisiae. Die Schaltungen OptoEXP, OptoINVRT und OptoAMP basieren auf dem lichtempfindlichen VP16-EL222-System. (A) Im OptoEXP-Kreislauf führt die Exposition gegenüber blauem Licht zu einer Konformationsänderung und Dimerisierung von VP16-EL222, die eine DNA-bindende Domäne freilegt und eine Transkription von PC120 ermöglicht. Die Figur wurde von Zhao et al.7 modifiziert. (B) OptoINVRT-Schaltungen nutzen die GAL (siehe abgebildeten) oder Q-Regulinen, um die Expression im Dunkeln zu induzieren. In GAL-basierten Schaltungen werden VP16-EL222 und GAL4 konstitutiv ausgedrückt, während PC120-Laufwerke Ausdruck des GAL80-Repressors erzeugen (in Q-basierten Schaltungen werden GAL4 und GAL80 durch QF2 bzw. QS ersetzt, und anstelle eines GAL-Promotors wird ein synthetischer QUAS-haltiger Promotor verwendet). Im Licht verhindert Gal80p die Aktivierung des interessierenden Gens aus PGAL1. Im Dunkeln wird GAL80 nicht exprimiert und schnell abgebaut, indem es zu einer konstitutiven Degron-Domäne (kleine braune Domäne) verschmolzen wird, was die Aktivierung von PGAL1 durch Gal4p ermöglicht. Die Figur wurde von Zhao et al.5 modifiziert. (C) OptoAMP-Schaltungen verwenden auch VP16-EL222 zur Steuerung der GAL- (siehe abgebildeten) oder Q-Regulien. In diesen Schaltkreisen wird der GAL80-Repressor (oder QS) konstitutiv ausgedrückt und mit einem lichtempfindlichen Degron (kleine blaue Domäne) verschmolzen, der eine strenge Unterdrückung im Dunkeln gewährleistet. PC120 und eine überempfindliche VP16-EL222-Mutantenkontrollexpression von GAL4 (oder QF2) mit Licht, die PGAL1 (oder einen QUAS-haltigen Promotor) im Licht stark aktiviert. GAL-abgeleitete Schaltungen können technische Formen von PGAL1 verwenden, wie PGAL1-M oder PGAL1-S, die eine erhöhte Aktivität aufweisen, sowie Wildtyp-Promotoren, die von der GAL-Regulierung kontrolliert werden (PGAL1, PGAL10, PGAL2, PGAL7). Die Figur wurde von Zhao et al.12 modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
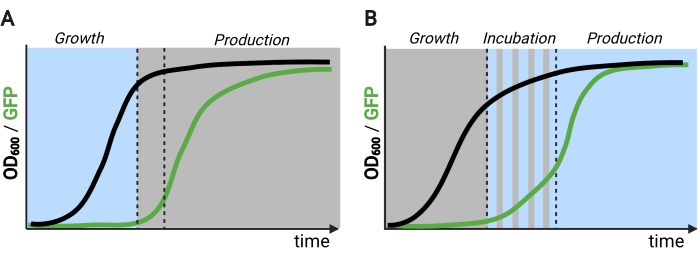
Abbildung 2: Zwei- und dreiphasige Fermentationen im Laufe der Zeit . (A) Zweiphasige Fermentationen, die mit invertierten Schaltkreisen betrieben werden, bestehen aus einer lichtgetriebenen Wachstumsphase und einer dunklen Produktionsphase. In der Wachstumsphase reichert sich Biomasse an, da der Produktionsweg unterdrückt bleibt. Nach Erreichen des gewünschten OD600 werden die Zellen ins Dunkel verschoben, um sich metabolisch anzupassen, bevor sie für die Produktionsphase in frischen Medien resuspendiert werden. (B) In einem dreiphasigen Prozess werden die Wachstums-, Inkubations- und Produktionsphasen durch einzigartige Lichtpläne definiert, die aus einer dunklen Wachstumsperiode, einer gepulsten Inkubation und einer vollständig beleuchteten Produktionsphase bestehen können. Mit Biorender erstellte Figur. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Optogenetische Schaltkreise wurden auch für die dynamische Steuerung der chemischen und Proteinproduktion in E. coli entwickelt. OptoLAC-Schaltungen steuern den bakteriellen LacI-Repressor über die lichtempfindliche pDawn-Schaltung, die auf dem Zweikomponentensystem YF1/FixJ6 basiert (Abbildung 3). Ähnlich wie OptoINVRT5 sind OptoLAC-Schaltkreise so konzipiert, dass sie die Genexpression im Licht unterdrücken und im Dunkeln induzieren. Die Expressionsniveaus, die OptoLAC-Schaltungen verwenden, können mit denen der Standard-Isopropyl-β-d-1-thiogalactopyranosid-Induktion (IPTG) mithalten oder diese übertreffen, wodurch die Festigkeit der chemischen Induktion erhalten bleibt und gleichzeitig eine verbesserte Abstimmbarkeit und Reversibilität geboten wird6. Daher ermöglichen OptoLAC-Schaltkreise eine effektive optogenetische Kontrolle für das Metabolic Engineering in E. coli.
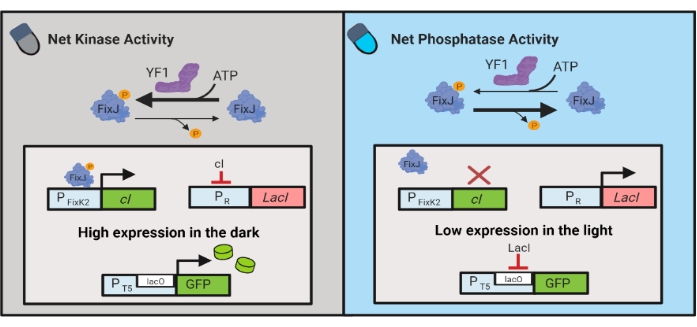
Abbildung 3: OptoLAC-Schaltungen zur dynamischen Regelung von E. coli. Die OptoLAC-Schaltungen passen das pDawn-System und das lac-Operon an, um eine Aktivierung im Dunkeln und eine Unterdrückung im Licht zu erreichen. Im Dunkeln phosphoryliert YF1 FixJ, wodurch dann der PFixK2-Promotor aktiviert wird, um den cI-Repressor auszudrücken. Der cI-Repressor verhindert die Expression des lacI-Repressors aus dem PR-Promotor, der die Transkription des interessierenden Gens von einem lacO-haltigen Promotor ermöglicht. Umgekehrt reduziert blaues Licht die YF1-Nettokinaseaktivität, kehrt die FixJ-Phosphorylierung und damit die cI-Expression um, was die Expression von lacI unterdrückt und die Expression durch den lacO-haltigen Promotor verhindert. Die Figur wurde von Lalwani et al.6 modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Wir beschreiben hier die grundlegenden Protokolle für lichtgesteuerte Fermentationen von S. cerevisiae und E. coli zur chemischen oder Proteinproduktion. Sowohl für Hefe als auch für Bakterien konzentrieren wir uns zunächst auf Fermentationen mit einer lichtgetriebenen Wachstumsphase und einer dunkelheitsinduzierten Produktionsphase, die durch OptoINVRT- und OptoLAC-Schaltkreise ermöglicht wird. Anschließend beschreiben wir ein Protokoll für eine dreiphasige (Wachstum, Induktion, Produktion) lichtgesteuerte Fermentation, die durch OptoAMP-Schaltungen ermöglicht wird. Darüber hinaus beschreiben wir, wie optogenetisch kontrollierte Fermentationen von Mikrotiterplatten auf Bioreaktoren im Labormaßstab hochskaliert werden können. Mit diesem Protokoll wollen wir einen vollständigen und leicht reproduzierbaren Leitfaden für die Durchführung lichtgesteuerter Fermentationen für die chemische oder Proteinproduktion bereitstellen.
Protocol
Representative Results
Discussion
Dynamische Kontrolle wird seit langem angewendet, um die Ausbeute für metabolisches Engineering und rekombinante Proteinproduktion zu verbessern4. Verschiebungen in der enzymatischen Expression werden am häufigsten mit chemischen Induktoren wie IPTG21, Galactose22 und Tetracyclin23 durchgeführt, aber auch unter Verwendung von Prozessbedingungen wie Temperatur und pH-Wert vermittelt. Die optogenetische Kontrolle der Genexpre…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Diese Forschung wurde vom U.S. Department of Energy, Office of Science, Office of Biological and Environmental Research Award Number DE-SC0019363, dem NSF CAREER Award CBET-1751840, The Pew Charitable Trusts und dem Camille Dreyfus Teacher-Scholar Award unterstützt.
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
References
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bio-ingénierie. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

