Protéomique de spectrométrie de masse guidée par lignée cellulaire dans l’embryon (grenouille) en développement
Summary
Nous décrivons ici une caractérisation protéomique basée sur la spectrométrie de masse de lignées cellulaires avec des destins tissulaires connus chez l’embryon vertébré Xenopus laevis .
Abstract
La caractérisation des événements moléculaires lorsque les cellules donnent naissance à des tissus et des organes permet de mieux comprendre le développement normal et de concevoir des remèdes efficaces contre les maladies. Les technologies permettant l’identification et la quantification précises de divers types et d’un grand nombre de protéines fourniraient encore des informations manquantes sur les mécanismes moléculaires orchestrant le développement des tissus et des organismes dans l’espace et le temps. Ici, nous présentons un protocole basé sur la spectrométrie de masse qui permet de mesurer des milliers de protéines dans des lignées cellulaires identifiées dans des embryons Xenopus laevis (grenouille). L’approche s’appuie sur des cartes reproductibles du devenir cellulaire et des méthodes établies pour identifier, marquer, suivre et échantillonner par fluorescence les cellules et leur descendance (clones) à partir de ce modèle de développement des vertébrés. Après avoir recueilli le contenu cellulaire à l’aide de micro-échantillonnage ou isolé des cellules par dissection ou tri cellulaire activé par fluorescence, les protéines sont extraites et traitées pour une analyse protéomique ascendante. La chromatographie liquide et l’électrophorèse capillaire sont utilisées pour fournir une séparation évolutive pour la détection et la quantification des protéines avec la spectrométrie de masse à haute résolution (HRMS). Des exemples représentatifs sont fournis pour la caractérisation protéomique des cellules destinées au tissu neural. La protéomique HRMS guidée par lignée cellulaire est adaptable à différents tissus et organismes. Il est suffisamment sensible, spécifique et quantitatif pour examiner la dynamique spatio-temporelle du protéome au cours du développement des vertébrés.
Introduction
Notre compréhension de la différenciation cellulaire et de la genèse des tissus et des organes est le résultat de décennies de criblages ciblés élaborés de gènes et de leurs produits. Accroître notre connaissance de toutes les biomolécules et de leurs quantités lors d’événements cellulaires importants aiderait à démêler les mécanismes moléculaires qui contrôlent la structuration spatiale et temporelle du plan corporel des vertébrés. Les technologies permettant l’amplification moléculaire et le séquençage sont maintenant en mesure de rendre compte régulièrement d’un grand nombre de gènes et de transcrits à l’appui d’études fondées sur des hypothèses dans la recherche biologique fondamentale et translationnelle. Pour comprendre les systèmes en développement, une relation complexe entre la transcription et la traduction plaide en faveur de l’analyse directe de multiples protéines et de leurs modifications post-traductionnelles. La protéomique globale utilisant des systèmes biologiques in vitro, tels que les cellules souches pluripotentes induites, a commencé à délimiter les mécanismes d’induction tissulaire 1,2. Dans les organismes complexes, tels que l’embryon de vertébrés, le développement repose sur les gradients morphogènes dans le contexte de l’espace et du temps3. Il s’ensuit que l’acquisition de connaissances sur les changements protéomiques à mesure que les cellules se différencient pour former des tissus spécialisés, tels que les tissus neuraux, offre une clé pour débloquer des programmes moléculaires contrôlant le développement normal et défectueux et guider les thérapies de prochaine génération.
La grenouille à griffes vertébrée d’Afrique du Sud (Xenopus laevis) est un modèle bien établi en biologie cellulaire et développementale, neuro- et régénérative. Le prix Nobel de physiologie ou médecine 4,5 de Sir John Gurdon en 2012 pour la découverte de la pluripotence du noyau somatique a souligné l’importance de ce modèle pour les découvertes en études fondamentales et translationnelles. Les embryons Xenopus se développent à l’extérieur de la mère, facilitant ainsi la manipulation directe des cellules, des clones cellulaires et de l’expression génique à différents stades de développement. La pigmentation asymétrique et les divisions cellulaires stéréotypées ont permis de cartographier des cartes de devenir reproductibles à partir de l’embryon de stade 7,8 à 16-6 et 32 cellules. Pour la protéomique basée sur la spectrométrie de masse à haute résolution (SGRH), les avantages supplémentaires du modèle comprennent une taille relativement grande (~1 mm de diamètre), qui donne une teneur abondante en protéines pour l’analyse (~130 μg chez les embryons au stade de clivage précoce, ~10 μg de teneur en protéines dans les cellules individuelles de l’embryon à 16 cellules)9,10.
À l’heure actuelle, le SGRH est la principale technologie de choix pour la détection des protéines. Cette technologie permet la détection et la quantification directes, sensibles et spécifiques de multiples, généralement des centaines, voire des milliers de protéines différentes11. La protéomique ascendante du SGRH comporte une série d’étapes interconnectées. Après l’extraction de l’échantillon de cellule/tissu, les protéines sont digérées avec une enzyme protéolytique, telle que la trypsine (protéomique ascendante). Les peptides résultants sont séparés en fonction de leurs différentes propriétés physico-chimiques, y compris l’hydrophobicité (chromatographie liquide en phase inverse, LC), la charge nette (chromatographie par échange d’ions), la taille (chromatographie d’exclusion de taille) ou la mobilité électrophorétique (électrophorèse capillaire, CE). Les peptides sont ensuite chargés (ionisés), généralement à l’aide d’une ionisation par électronébulisation (ESI), et les ions peptidiques sont détectés et séquencés par fragmentation en phase gazeuse par HRMS en tandem. Les données peptidiques résultantes sont mises en correspondance avec le protéome de l’organisme étudié. Avec l’intensité du signal d’ions peptidiques spécifiques aux protéines (protéotypiques) corrélée à la concentration, la quantification des protéines peut être effectuée sans marquage ou sur la base du marquage (quantification multiplexage). La protéomique du SGRH fournit une riche source d’information sur l’état moléculaire du système à l’étude, ce qui permet de générer des hypothèses et des études fonctionnelles de suivi.
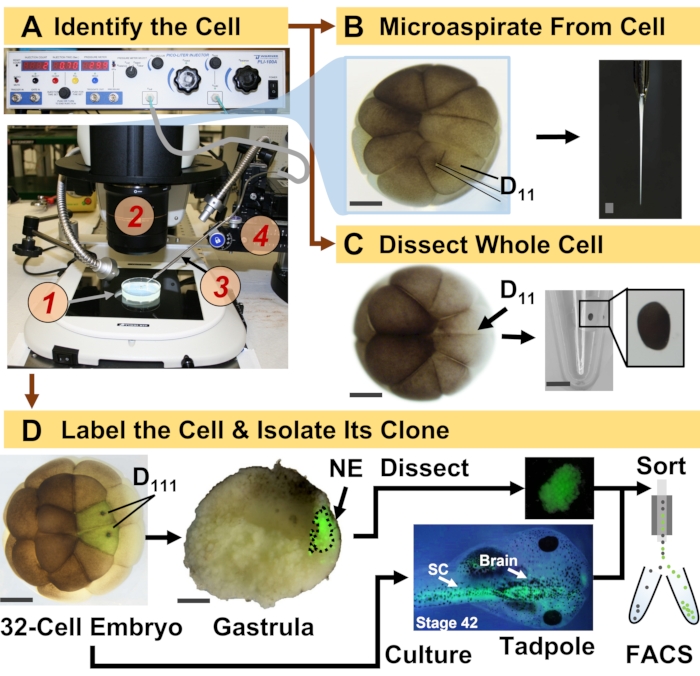
Figure 1 : Protéomique spatio-temporellement évolutive permettant la protéomique HRMS guidée par lignée cellulaire dans l’embryon (grenouille) en développement. (A) Visualisation de l’échantillon (1) à l’aide d’un stéréomicroscope (2) pour l’injection d’une cellule identifiée (encadré), à l’aide d’une micropipette fabriquée (3) sous contrôle par un étage de translation (4). (B) Prélèvement subcellulaire de la cellule D 11 gauche identifiée dans un embryon à16 cellules. (C) Dissection d’une cellule D 11 entière à partir d’un embryon à16 cellules. (D) Traçage fluorescent (vert) des descendants D111 gauche et droit d’un embryon à 32 cellules pour guider la dissection de l’ectoderme neural (NE) dans la gastrule (stade 10) et isolement du tissu descendant du têtard à l’aide de FACS. Barres d’échelle : 200 μm pour les embryons, 1,25 mm pour le flacon. Les figures ont été adaptées avec la permission des références 15,19,21,59. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Le protocole présenté ici permet de quantifier un grand nombre de protéines dans des cellules/tissus identifiés dans des embryons de X. laevis en développement basés sur le SGRH. L’approche s’appuie sur une identification cellulaire précise, des cartes de destin cellulaire reproductibles et des méthodologies établies pour suivre les lignées cellulaires dans ce modèle biologique 6,7,8. Comme le montre la figure 1, nous étudions les protéomes de cellules individuelles en utilisant la dissection de cellules entières ou le microéchantillonnage capillaire pour aspirer le contenu cellulaire. Le suivi de la lignée d’une cellule nous permet d’étudier l’évolution spatio-temporelle du protéome lorsque les cellules forment des tissus pendant la gastrulation. La descendance cellulaire est marquée par fluorescence par injection d’un fluorophore conjugué à du dextrane inerte ou à l’ARNm pour la protéine fluorescente (p. ex. protéine fluorescente verte, ou GFP). La descendance marquée est isolée aux moments de développement souhaités. Pendant la gastrulation, les clones cellulaires étroitement regroupés peuvent être isolés par dissection. Après gastrulation, les clones cellulaires peuvent être distribués dans l’embryon en raison des mouvements migratoires et peuvent être isolés des tissus dissociés par tri cellulaire activé par fluorescence (FACS). Les protéines de ces cellules et tissus sont mesurées par protéomique ascendante utilisant HPLC ou CE pour la séparation et ESI tandem HRMS pour l’identification. La protéomique HRMS guidée par lignée cellulaire est évolutive à différentes tailles de cellules et lignées au sein de l’embryon et est spécifique, sensible et quantitative. À travers des exemples sélectionnés présentés ici, nous démontrons également que ce protocole est évolutif et largement adaptable à différents types de cellules et de lignées cellulaires.
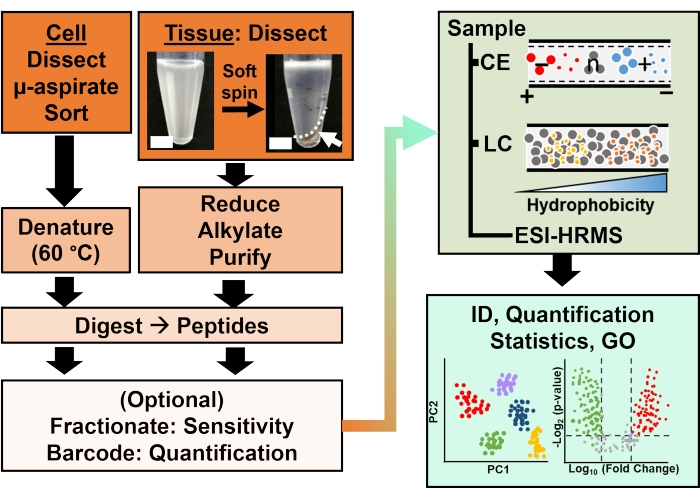
Figure 2 : Le flux de travail bioanalytique. La micro-dissection et l’aspiration capillaire, ou FACS, ont facilité l’échantillonnage de la teneur en protéines cellulaires et clonales. Épuisement des protéines vitellines abondantes et séparation par électrophorèse capillaire (EC) ou chromatographie liquide à nanoflux (LC) sensibilité à l’identification améliorée (ID) à l’aide de la spectrométrie de masse à haute résolution (HRMS) par ionisation électronébulaire (ESI). La quantification a révélé une dysrégulation, fournissant de nouvelles informations pour des études basées sur des hypothèses en conjonction avec les informations disponibles à partir de l’ontologie génique (GO). Les chiffres ont été adaptés avec la permission de la référence15. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocol
Representative Results
Discussion
Ce protocole permet de caractériser l’expression des protéines dans les lignées cellulaires identifiées chez les embryons de l’espèce Xenopus . Issue du SGRH, la méthodologie combine une spécificité exquise dans l’identification moléculaire, une capacité de détection multi-protéines sans sondes moléculaires (généralement des centaines à des milliers de protéines différentes) et une capacité de quantification. L’adaptabilité aux outils et flux de travail classiques en (neuro)biologie c…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Nous sommes reconnaissants à Jie Li (Université du Maryland, College Park) pour ses précieuses discussions sur la dissociation embryonnaire et le FACS. Nous remercions Vi M. Quach et Camille Lombard-Banek pour leur aide à la préparation des échantillons et à la collecte de données dans les études antérieures illustrant les applications protéomiques mises en évidence dans ce protocole. Certaines parties de ce travail ont été soutenues par la National Science Foundation sous le numéro d’attribution IOS-1832968 CAREER (à P.N.), les National Institutes of Health sous le numéro d’attribution R35GM124755 (à P.N.), le programme de partenariat entre l’Université du Maryland et le National Cancer Institute (à P.N.) et les bourses de recherche de la COSMOS Club Foundation (à A.B.B. et L.R.P.).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Biologie du développement. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Biologie du développement. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Biologie du développement. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

