ספקטרומטריית מסה מונחית שושלת תאים פרוטאומיקה בעובר המתפתח (צפרדע)
Summary
כאן אנו מתארים אפיון פרוטאומי מבוסס ספקטרומטריית מסות של שושלות תאים עם גורלות רקמות ידועים בעובר בעל חוליות Xenopus laevis .
Abstract
אפיון אירועים מולקולריים כתאים המולידים רקמות ואיברים מעלה פוטנציאל להבין טוב יותר התפתחות תקינה ולתכנן תרופות יעילות למחלות. טכנולוגיות המאפשרות זיהוי וכימות מדויקים של סוגים שונים ומספר גדול של חלבונים יספקו מידע שעדיין חסר על מנגנונים מולקולריים המתזמרים התפתחות רקמות ואורגניזמים במרחב ובזמן. כאן אנו מציגים פרוטוקול מבוסס ספקטרומטריית מסות המאפשר מדידה של אלפי חלבונים בשושלות תאים מזוהות בעוברי צפרדע (Xenopus laevis ). הגישה מתבססת על מפות גורל התא הניתנות לשחזור ושיטות מבוססות לזיהוי, תיוג, מעקב ודגימה פלואורסצנטית של תאים וצאצאיהם (שיבוטים) ממודל זה של התפתחות בעלי חוליות. לאחר איסוף תוכן תאי באמצעות מיקרו-דגימה או בידוד תאים על ידי דיסקציה או מיון תאים המופעלים על ידי פלואורסצנטיות, חלבונים מופקים ומעובדים לאנליזה פרוטאומית מלמטה למעלה. כרומטוגרפיה נוזלית ואלקטרופורזה נימית משמשות כדי לספק הפרדה ניתנת להרחבה לזיהוי וכימות חלבונים באמצעות ספקטרומטריית מסות ברזולוציה גבוהה (HRMS). דוגמאות מייצגות ניתנות לאפיון פרוטאומי של תאים בעלי גורל לרקמה עצבית. פרוטאומיקה HRMS מונחית שושלת תאים ניתנת להתאמה לרקמות ואורגניזמים שונים. הוא רגיש, ספציפי וכמותי מספיק כדי להציץ לתוך הדינמיקה המרחבית-טמפורלית של הפרוטאום במהלך התפתחות בעלי חוליות.
Introduction
הבנתנו את התמיינות התאים ואת היווצרותם של רקמות ואיברים היא תוצאה של עשרות שנים של בדיקות ממוקדות מורכבות של גנים ותוצריהם. הגדלת הידע שלנו על כל הביומולקולות וכמותן במהלך אירועים תאיים חשובים תסייע לפענח מנגנונים מולקולריים השולטים בתבניות המרחביות והזמניות של תוכנית הגוף של בעלי החוליות. טכנולוגיות המאפשרות הגברה מולקולרית וריצוף מסוגלות כיום לדווח באופן שגרתי על מספר רב של גנים ותעתיקים, ותומכות במחקרים מונחי השערות במחקר ביולוגי ותרגומי בסיסי. כדי להבין מערכות מתפתחות, מערכת יחסים מורכבת בין שעתוק לתרגום דוגלת בניתוח ישיר של חלבונים מרובים ובשינויים שלאחר התרגום. פרוטאומיקה גלובלית המשתמשת במערכות ביולוגיות במבחנה, כגון תאי גזע פלוריפוטנטיים מושרים, החלה לשרטט מנגנונים של השראת רקמות 1,2. באורגניזמים מורכבים, כגון עובר בעל חוליות, ההתפתחות מסתמכת על שיפועים מורפוגניים בהקשר של מרחב וזמן3. מכאן נובע כי רכישת ידע על שינויים פרוטאומיים כאשר תאים מתמיינים ליצירת רקמות מיוחדות, כגון רקמות עצביות, מציעה מפתח לפתיחת תוכניות מולקולריות השולטות בהתפתחות נורמלית ופגומית ומנחה את הדור הבא של הטיפולים.
צפרדע בעלת טפרים דרום אפריקאית (Xenopus laevis) היא מודל מבוסס היטב בביולוגיה תאית והתפתחותית, נוירו ורגנרטיבית. פרס נובללפיזיולוגיה או לרפואה 4,5 של סר ג’ון גורדון לשנת 2012 על גילוי הפלוריפוטנטיות של הגרעין הסומטי הדגיש את חשיבותו של מודל זה לתגליות במחקרים בסיסיים ותרגומיים. עוברי קסנופוס מתפתחים חיצונית לאם, ובכך מאפשרים מניפולציה ישירה של תאים, שיבוטים של תאים וביטוי גנים בשלבי התפתחות שונים. פיגמנטציה אסימטרית וחלוקות תאים סטריאוטיפיות אפשרו לשרטט מפות גורל הניתנות לשחזור מהעובר בן 16-6 ו-32 תאים 7,8. עבור פרוטאומיקה מבוססת ספקטרומטריית מסה ברזולוציה גבוהה (HRMS), יתרונות נוספים של המודל כוללים גודל גדול יחסית (~ 1 מ”מ קוטר), אשר מניב תכולת חלבון בשפע לניתוח (~ 130 מיקרוגרם בעוברים בשלב המחשוף המוקדם, ~ 10 מיקרוגרם של תכולת חלבון בתאים בודדים של עובר בן 16 תאים)9,10.
כיום, HRMS היא הטכנולוגיה המובילה המועדפת לגילוי חלבונים. טכנולוגיה זו מאפשרת איתור וכימות ישיר, רגיש וספציפי של חלבונים מרובים, בדרך כלל מאות עד אלפי חלבונים שונים11. פרוטאומיקה מלמטה למעלה על ידי HRMS כוללת סדרה של שלבים הקשורים זה בזה. לאחר מיצוי מדגימת התא/רקמה, חלבונים מתעכלים עם אנזים מפרקי חלבון, כגון טריפסין (פרוטאומיקה מלמטה למעלה). הפפטידים המתקבלים מופרדים בהתבסס על התכונות הפיזיקוכימיות השונות שלהם, כולל הידרופוביות (כרומטוגרפיה נוזלית הפוכה, LC), מטען נטו (כרומטוגרפיה של החלפת יונים), גודל (כרומטוגרפיה של אי הכללת גודל), או ניידות אלקטרופורטית (אלקטרופורזה נימית, CE). לאחר מכן פפטידים נטענים (מיוננים), בדרך כלל באמצעות יינון אלקטרוספריי (ESI), ויונים פפטידים מזוהים ומרוצפים באמצעות פיצול פאזת גז על ידי טנדם HRMS. הנתונים הפפטידים המתקבלים ממופים לפרוטאום של האורגניזם הנחקר. עם מתאם בין עוצמת אות יון פפטידי ספציפי לחלבון (פרוטטיפי) לבין ריכוז, ניתן לבצע כימות חלבון ללא תווית או מבוסס תווית (כימות ריבוב). פרוטאומיקה HRMS מניבה משאב עשיר של מידע על המצב המולקולרי של המערכת הנחקרת, ומאפשרת יצירת השערות ומחקרים פונקציונליים עוקבים.
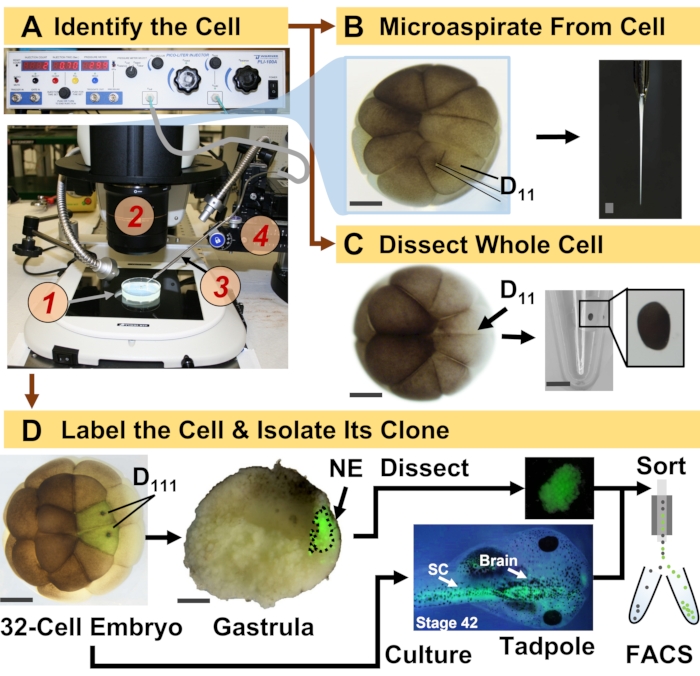
איור 1: פרוטאומיקה מרחבית-זמנית המאפשרת פרוטאומיקה מונחית שושלת תאים HRMS בעובר המתפתח (צפרדע). (A) הדמיה של הדגימה (1) באמצעות סטריאומיקרוסקופ (2) להזרקה של תא מזוהה (inset), באמצעות מיקרופיפטה מפוברקת (3) תחת בקרה על ידי שלב תרגום (4). (B) דגימה תת-תאית של תא D 11 השמאלי שזוהה בעובר בן16 תאים. (C) דיסקציה של תא D 11 שלם מעובר בן16 תאים. (D) מעקב פלואורסצנטי (ירוק) של צאצאי D111 משמאל ומימין מעובר בן 32 תאים כדי להנחות דיסקציה של האקטודרם העצבי (NE) בגסטרולה (שלב 10) ובידוד הרקמה היורדת מהראשנים באמצעות FACS. מוטות קנה מידה: 200 מיקרומטר לעוברים, 1.25 מ”מ לבקבוקון. הנתונים אומצו באישור הפניות 15,19,21,59. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
הפרוטוקול המוצג כאן מאפשר כימות מבוסס HRMS של מספר רב של חלבונים בתאים/רקמות מזוהים בעוברי X. laevis מתפתחים. הגישה מתבססת על זיהוי מדויק של תאים, מפות גורל תאים הניתנות לשחזור, ומתודולוגיות מבוססות למעקב אחר שושלות תאים במודל ביולוגיזה 6,7,8. כפי שניתן לראות באיור 1, אנו חוקרים פרוטאומים מתאים בודדים על-ידי שימוש בדיסקציה של תאים שלמים או במיקרו-דגימה נימית כדי לשאוף תוכן תאי. ניטור השושלת של התא מאפשר לנו לחקור את האבולוציה המרחבית-זמנית של הפרוטאום כאשר תאים יוצרים רקמות במהלך גסטרולציה. צאצאי התא מסומנים באופן פלואורסצנטי על ידי הזרקת פלואורופור מצומד לדקסטרן אינרטי או mRNA עבור חלבון פלואורסצנטי (למשל, חלבון פלואורסצנטי ירוק, או GFP). הצאצא המסומן מבודד בנקודות זמן התפתחותיות רצויות. במהלך הגסטרולציה, שיבוטים של תאים המקובצים היטב עשויים להיות מבודדים על ידי דיסקציה. לאחר הגסטרולציה, שיבוטים של תאים עשויים להיות מופצים בתוך העובר עקב תנועות נדידה וניתן לבודד אותם מרקמות מנותקות על ידי מיון תאים המופעלים על ידי פלואורסצנטיות (FACS). חלבונים בתאים וברקמות אלה נמדדים באמצעות פרוטאומיקה מלמטה למעלה המשתמשת ב- HPLC או CE להפרדה ו- ESI טנדם HRMS לזיהוי. פרוטאומיקה HRMS מונחית שושלת תאים ניתנת להרחבה לגדלי תאים ושושלות שונות בתוך העובר והיא ספציפית, רגישה וכמותית. באמצעות דוגמאות נבחרות המוצגות כאן, אנו גם מדגימים שפרוטוקול זה ניתן להרחבה ולהתאמה רחבה לסוגים שונים של תאים ושושלות תאים.
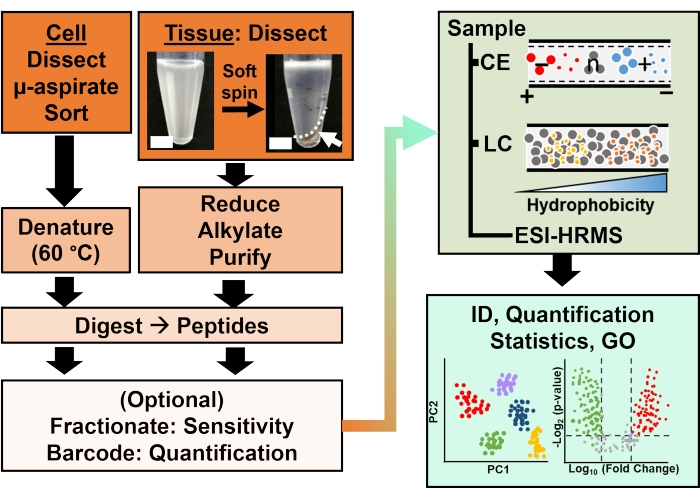
איור 2: תהליך העבודה הביואנליטי. מיקרו-דיסקציה ושאיפה נימית, או FACS הקל על דגימה של תכולת חלבון תאי ושבטי. דלדול חלבוני חלמון בשפע והפרדה על ידי אלקטרופורזה נימית (CE) או כרומטוגרפיה נוזלית ננו-זרימה (LC) רגישות זיהוי משופר (ID) באמצעות יינון אלקטרוספריי (ESI) ספקטרומטריית מסה ברזולוציה גבוהה (HRMS). הכימות חשף חוסר ויסות, וסיפק מידע חדש למחקרים מונחי השערות בשילוב עם מידע זמין מאונטולוגיה גנטית (GO). האיורים עובדו באישור הפניה15. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Protocol
Representative Results
Discussion
פרוטוקול זה מאפשר אפיון ביטוי חלבונים בשושלות תאים מזוהות בעוברים מזן הקסנופוס . המתודולוגיה, המבוססת על HRMS, משלבת ספציפיות מעולה בזיהוי מולקולרי, יכולת לגילוי רב-חלבוני ללא בדיקות מולקולריות (בדרך כלל מאות עד אלפי חלבונים שונים), ויכולת כימות. הסתגלות לכלים קלאסיים ולתהליכי עבודה בב…
Divulgations
The authors have nothing to disclose.
Acknowledgements
אנו אסירי תודה לג’יה לי (אוניברסיטת מרילנד, קולג’ פארק) על דיונים חשובים על דיסוציאציה עוברית ו- FACS. אנו מודים ל- Vi M. Quach ול- Camille Lombard-Banek על הסיוע בהכנת הדגימות ואיסוף הנתונים במחקרים קודמים המדגימים את היישומים הפרוטאומיים המודגשים בפרוטוקול זה. חלקים מעבודה זו נתמכו על ידי הקרן הלאומית למדע תחת פרס מספר IOS-1832968 CAREER (ל- P.N.), המכונים הלאומיים לבריאות תחת פרס מספר R35GM124755 (ל- P.N.), תוכנית השותפות של אוניברסיטת מרילנד-המכון הלאומי לסרטן (ל- P.N.), ופרסי המחקר של קרן מועדון קוסמוס (ל- A.B.B. ו- L.R.P).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Biologie du développement. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Biologie du développement. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Biologie du développement. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

