विकासशील (मेंढक) भ्रूण में सेल-वंश निर्देशित मास स्पेक्ट्रोमेट्री प्रोटिओमिक्स
Summary
यहां हम कशेरुक जेनोपस लेविस भ्रूण में ज्ञात ऊतक भाग्य के साथ सेल वंश के मास स्पेक्ट्रोमेट्री-आधारित प्रोटिओमिक लक्षण वर्णन का वर्णन करते हैं।
Abstract
आणविक घटनाओं का लक्षण वर्णन क्योंकि कोशिकाएं ऊतकों और अंगों को जन्म देती हैं, सामान्य विकास को बेहतर ढंग से समझने और रोगों के लिए कुशल उपचार डिजाइन करने की क्षमता बढ़ाती हैं। विभिन्न प्रकारों और बड़ी संख्या में प्रोटीन की सटीक पहचान और परिमाणीकरण को सक्षम करने वाली प्रौद्योगिकियां अंतरिक्ष और समय में ऊतक और जीव विकास को व्यवस्थित करने वाले आणविक तंत्र पर अभी भी लापता जानकारी प्रदान करेंगी। यहां, हम एक मास स्पेक्ट्रोमेट्री-आधारित प्रोटोकॉल प्रस्तुत करते हैं जो जेनोपस लेविस (मेंढक) भ्रूण में पहचाने गए सेल वंश में हजारों प्रोटीन के माप को सक्षम बनाता है। दृष्टिकोण प्रजनन योग्य सेल-भाग्य मानचित्रों और कशेरुक विकास के इस मॉडल से कोशिकाओं और उनकी संतान (क्लोन) की पहचान, फ्लोरोसेंटली लेबल, ट्रैक और नमूना करने के लिए स्थापित तरीकों पर आधारित है। माइक्रोसैंपलिंग का उपयोग करके सेलुलर सामग्री एकत्र करने या विच्छेदन या प्रतिदीप्ति-सक्रिय सेल सॉर्टिंग द्वारा कोशिकाओं को अलग करने के बाद, प्रोटीन को निकाला जाता है और नीचे-ऊपर प्रोटिओमिक विश्लेषण के लिए संसाधित किया जाता है। तरल क्रोमैटोग्राफी और केशिका वैद्युतकणसंचलन का उपयोग उच्च-रिज़ॉल्यूशन मास स्पेक्ट्रोमेट्री (एचआरएमएस) के साथ प्रोटीन का पता लगाने और परिमाणीकरण के लिए स्केलेबल पृथक्करण प्रदान करने के लिए किया जाता है। तंत्रिका-ऊतक वसा कोशिकाओं के प्रोटिओमिक लक्षण वर्णन के लिए प्रतिनिधि उदाहरण प्रदान किए जाते हैं। सेल-वंश-निर्देशित एचआरएमएस प्रोटिओमिक्स विभिन्न ऊतकों और जीवों के लिए अनुकूलनीय है। कशेरुक विकास के दौरान प्रोटिओम की स्थानिक-अस्थायी गतिशीलता में देखने के लिए यह पर्याप्त रूप से संवेदनशील, विशिष्ट और मात्रात्मक है।
Introduction
सेल भेदभाव और ऊतकों और अंगों की उत्पत्ति की हमारी समझ जीन और उनके उत्पादों की विस्तृत लक्षित स्क्रीन के दशकों का परिणाम है। महत्वपूर्ण सेलुलर घटनाओं के दौरान सभी बायोमोलेक्यूल्स और उनकी मात्रा के बारे में हमारे ज्ञान को बढ़ाने से आणविक तंत्र को उजागर करने में मदद मिलेगी जो कशेरुक शरीर योजना के स्थानिक और लौकिक पैटर्निंग को नियंत्रित करते हैं। आणविक प्रवर्धन और अनुक्रमण को सक्षम करने वाली प्रौद्योगिकियां अब बड़ी संख्या में जीन और प्रतिलेख पर नियमित रूप से रिपोर्ट करने में सक्षम हैं, जो बुनियादी जैविक और अनुवाद अनुसंधान में परिकल्पना-संचालित अध्ययनों का समर्थन करती हैं। विकासशील प्रणालियों को समझने के लिए, प्रतिलेखन और अनुवाद के बीच एक जटिल संबंध कई प्रोटीनों और उनके पोस्ट-ट्रांसलेशनल संशोधनों के प्रत्यक्ष विश्लेषण की वकालत करता है। इन विट्रो जैविक प्रणालियों का उपयोग करके वैश्विक प्रोटिओमिक्स, जैसे कि प्रेरित प्लुरिपोटेंट स्टेम सेल, ऊतक प्रेरण 1,2 के तंत्र को चित्रित करना शुरू कर दिया। जटिल जीवों में, जैसे कशेरुक भ्रूण, विकास अंतरिक्ष और समय 3 के संदर्भ में मोर्फोजेन ग्रेडिएंट पर निर्भर करताहै। यह इस प्रकार है कि प्रोटिओमिक परिवर्तनों का ज्ञान प्राप्त करना क्योंकि कोशिकाएं विशेष ऊतकों, जैसे तंत्रिका ऊतकों को बनाने के लिए अंतर करती हैं, सामान्य और दोषपूर्ण विकास को नियंत्रित करने वाले आणविक कार्यक्रमों को अनलॉक करने और अगली पीढ़ी के चिकित्सीय मार्गदर्शन के लिए एक कुंजी प्रदान करती हैं।
कशेरुक दक्षिण अफ्रीकी पंजे वाला मेंढक (जेनोपस लेविस) सेल और विकासात्मक, न्यूरो-और पुनर्योजी जीव विज्ञान में एक अच्छी तरह से स्थापित मॉडल है। दैहिक नाभिक की प्लूरिपोटेंसी की खोज के लिए सर जॉन गुरडन के 2012 के फिजियोलॉजी या मेडिसिन 4,5 में नोबेल पुरस्कार ने बुनियादी और ट्रांसलेशनल अध्ययनों में खोजों के लिए इस मॉडल के महत्व पर प्रकाश डाला। जेनोपस भ्रूण मां के लिए बाहरी रूप से विकसित होते हैं, इस प्रकार विकास के विभिन्न चरणों में कोशिकाओं, सेल क्लोन और जीन अभिव्यक्ति के प्रत्यक्ष हेरफेर की सुविधा प्रदान करते हैं। विषम रंजकता और रूढ़िवादी कोशिका विभाजन ने 16-6 और 32-सेल 7,8 चरण भ्रूण से प्रतिलिपि प्रस्तुत करने योग्य भाग्यमानचित्रों के चार्टिंग को सक्षम किया। उच्च-रिज़ॉल्यूशन मास स्पेक्ट्रोमेट्री (एचआरएमएस) आधारित प्रोटिओमिक्स के लिए, मॉडल के अतिरिक्त लाभों में अपेक्षाकृत बड़ा आकार (~ 1 मिमी व्यास) शामिल है, जो विश्लेषण के लिए प्रचुर मात्रा में प्रोटीन सामग्री पैदा करता है (प्रारंभिक दरार-चरण भ्रूण में ~ 130 μg, 16-सेल भ्रूण की एकल कोशिकाओं में ~ 10 μg प्रोटीन सामग्री)9,10।
वर्तमान में, एचआरएमएस प्रोटीन का पता लगाने के लिए पसंद की अग्रणी तकनीक है। यह तकनीक कई, आमतौर पर सैकड़ों से हजारों विभिन्न प्रोटीनों का प्रत्यक्ष, संवेदनशील और विशिष्ट पता लगाने और परिमाणीकरण करने में सक्षम बनातीहै। एचआरएमएस द्वारा बॉटम-अप प्रोटिओमिक्स में परस्पर जुड़े चरणों की एक श्रृंखला शामिल है। ऊतक के नमूने से निष्कर्षण के बाद, प्रोटीन को प्रोटियोलिटिक एंजाइम के साथ पचाया जाता है, जैसे ट्रिप्सिन (बॉटम-अप प्रोटिओमिक्स)। परिणामी पेप्टाइड्स को उनके विभिन्न भौतिक रासायनिक गुणों के आधार पर अलग किया जाता है, जिसमें हाइड्रोफोबिसिटी (उलट-चरण तरल क्रोमैटोग्राफी, एलसी), नेट चार्ज (आयन-एक्सचेंज क्रोमैटोग्राफी), आकार (आकार बहिष्करण क्रोमैटोग्राफी), या इलेक्ट्रोफोरेटिक गतिशीलता (केशिका वैद्युतकणसंचलन, सीई) शामिल हैं। पेप्टाइड्स को तब चार्ज (आयनित) किया जाता है, आमतौर पर इलेक्ट्रोस्प्रे आयनीकरण (ईएसआई) का उपयोग करके, और पेप्टाइड आयनों का पता लगाया जाता है और अग्रानुक्रम एचआरएमएस द्वारा गैस-चरण विखंडन के माध्यम से अनुक्रमित किया जाता है। परिणामी पेप्टाइड डेटा का अध्ययन किए जा रहे जीव के प्रोटिओम में मैप किया जाता है। प्रोटीन-विशिष्ट (प्रोटिओटिपिक) पेप्टाइड आयन सिग्नल तीव्रता एकाग्रता के साथ सहसंबंध के साथ, प्रोटीन परिमाणीकरण लेबल-मुक्त या लेबल-आधारित (मल्टीप्लेक्सिंग मात्रा) किया जा सकता है। एचआरएमएस प्रोटिओमिक्स अध्ययन के तहत सिस्टम की आणविक स्थिति पर जानकारी का एक समृद्ध संसाधन उत्पन्न करता है, जिससे परिकल्पनाओं और अनुवर्ती कार्यात्मक अध्ययनों की पीढ़ी की अनुमति मिलती है।
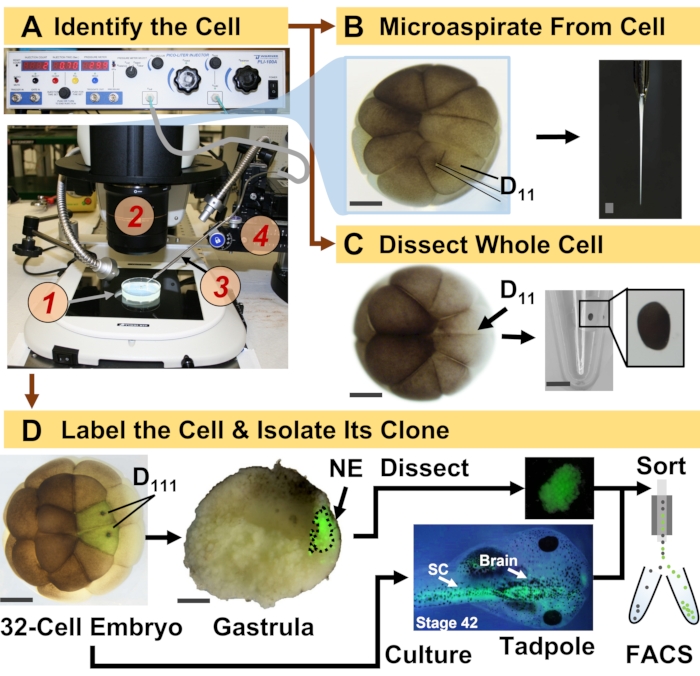
चित्रा 1: विकासशील (मेंढक) भ्रूण में सेल-वंश निर्देशित एचआरएमएस प्रोटिओमिक्स को सक्षम करने वाले स्थानिक स्केलेबल प्रोटिओमिक्स। (ए) एक पहचान किए गए सेल (इनसेट) के इंजेक्शन के लिए स्टीरियोमाइक्रोस्कोप (2) का उपयोग करके नमूने का विज़ुअलाइज़ेशन, अनुवाद-चरण द्वारा नियंत्रण में एक निर्मित माइक्रोपिपेट (3) का उपयोग करना (4). (बी) 16-सेल भ्रूण में पहचाने गए बाएं डी11 सेल का उपकोशिकीय नमूना। (सी) 16-सेल भ्रूण से पूरे डी 11 सेल का विच्छेदन। (डी) गैस्ट्रुला (चरण10) में तंत्रिका एक्टोडर्म (एनई) के विच्छेदन और एफएसीएस का उपयोग करके टैडपोल से वंशज ऊतक के अलगाव का मार्गदर्शन करने के लिए 32-सेल भ्रूण से बाएं और दाएं डी 111 संतानों का फ्लोरोसेंट (हरा) अनुरेखण। स्केल बार: भ्रूण के लिए 200 μm, शीशी के लिए 1.25 मिमी। आंकड़ेसंदर्भ 15,19,21,59 से अनुमति के साथ अनुकूलित किए गए थे। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
यहां प्रस्तुत प्रोटोकॉल एचआरएमएस-आधारित मात्रा में एक्स लेविस भ्रूण विकसित करने में पहचाने गए कोशिकाओं / ऊतकों में प्रोटीन की बड़ी संख्या का परिमाणीकरण करने में सक्षम बनाता है। दृष्टिकोण इस जैविक मॉडल 6,7,8 में सेल वंशावली को ट्रैक करने के लिए सटीक सेल पहचान, प्रतिलिपि प्रस्तुत करने योग्य सेल भाग्य मानचित्र और स्थापित पद्धतियों पर आधारित है। जैसा कि चित्रा 1 में दिखाया गया है, हम पूरे सेल विच्छेदन या केशिका माइक्रोसैंपलिंग को एस्पिरेट सेलुलर सामग्री को नियोजित करके एकल कोशिकाओं से प्रोटिओम का अध्ययन करते हैं। एक कोशिका के वंश की निगरानी हमें प्रोटिओम के स्थानिक विकास का अध्ययन करने की अनुमति देती है क्योंकि कोशिकाएं गैस्ट्रुलेशन के दौरान ऊतक बनाती हैं। सेल संतान को फ्लोरोसेंट प्रोटीन (जैसे, हरे फ्लोरोसेंट प्रोटीन, या जीएफपी) के लिए निष्क्रिय डेक्सट्रान या एमआरएनए के संयुग्मित फ्लोरोफोरे को इंजेक्ट करके फ्लोरोसेंट रूप से चिह्नित किया जाता है। लेबल की गई संतान को वांछित विकास समय बिंदुओं पर अलग किया जाता है। गैस्ट्रुलेशन के दौरान, सेल क्लोन जो कसकर क्लस्टर होते हैं, उन्हें विच्छेदन द्वारा अलग किया जा सकता है। गैस्ट्रुलेशन के बाद, सेल क्लोन को प्रवासी आंदोलनों के कारण भ्रूण के भीतर वितरित किया जा सकता है और प्रतिदीप्ति-सक्रिय सेल सॉर्टिंग (एफएसीएस) द्वारा अलग ऊतकों से अलग किया जा सकता है। इन कोशिकाओं और ऊतकों में प्रोटीन को पृथक्करण के लिए एचपीएलसी या सीई और पहचान के लिए ईएसआई टेंडम एचआरएमएस का उपयोग करके बॉटम-अप प्रोटिओमिक्स के माध्यम से मापा जाता है। सेल-वंश-निर्देशित एचआरएमएस प्रोटिओमिक्स भ्रूण के भीतर विभिन्न सेल आकारों और वंशों के लिए स्केलेबल है और विशिष्ट, संवेदनशील और मात्रात्मक है। यहां दिखाए गए चुनिंदा उदाहरणों के माध्यम से, हम इस प्रोटोकॉल को स्केलेबल और व्यापक रूप से विभिन्न प्रकार की कोशिकाओं और सेल वंशों के लिए अनुकूलनीय होने का भी प्रदर्शन करते हैं।
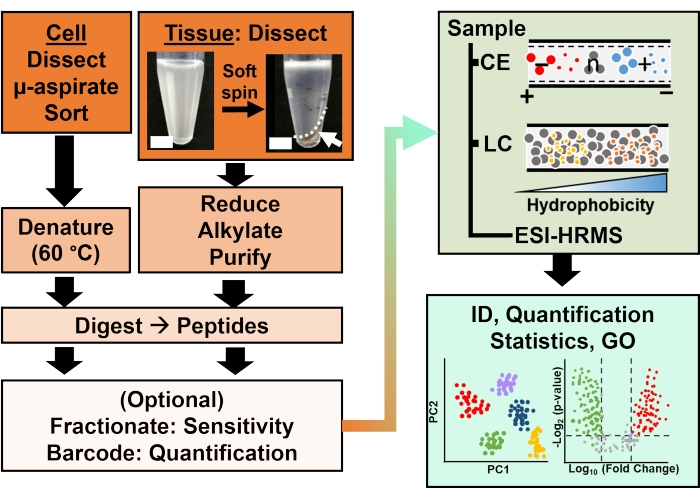
चित्रा 2: जैव-विश्लेषणात्मक वर्कफ़्लो। सूक्ष्म विच्छेदन और केशिका आकांक्षा, या एफएसीएस ने सेलुलर और क्लोनल प्रोटीन सामग्री के नमूने की सुविधा प्रदान की। इलेक्ट्रोस्प्रे आयनीकरण (ईएसआई) उच्च-रिज़ॉल्यूशन मास स्पेक्ट्रोमेट्री (एचआरएमएस) का उपयोग करके प्रचुर मात्रा में जर्दी प्रोटीन की कमी और केशिका वैद्युतकणसंचलन (सीई) या नैनो-प्रवाह तरल क्रोमैटोग्राफी (एलसी) बढ़ी हुई पहचान (आईडी) संवेदनशीलता द्वारा पृथक्करण। परिमाणीकरण ने डिसरेग्यूलेशन का खुलासा किया, जीन ऑन्कोलॉजी (जीओ) से उपलब्ध जानकारी के साथ संयोजन में परिकल्पना-संचालित अध्ययनों के लिए नई जानकारी प्रदान की। आंकड़े संदर्भ15 से अनुमति के साथ अनुकूलित किए गए थे। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
Protocol
Representative Results
Discussion
यह प्रोटोकॉल जेनोपस प्रजातियों के भ्रूण में पहचाने गए सेल वंश में प्रोटीन अभिव्यक्ति के लक्षण वर्णन को सक्षम बनाता है। एचआरएमएस से उपजी, कार्यप्रणाली आणविक पहचान में उत्तम विशिष्टता, आणविक जांच क…
Divulgations
The authors have nothing to disclose.
Acknowledgements
हम भ्रूण पृथक्करण और एफएसीएस पर मूल्यवान चर्चा के लिए जी ली (मैरीलैंड विश्वविद्यालय, कॉलेज पार्क) के आभारी हैं। क्वाच और केमिली लोम्बार्ड-बैनेक को पिछले अध्ययनों में नमूना तैयार करने और डेटा संग्रह के साथ सहायता के लिए धन्यवाद देते हैं जो इस प्रोटोकॉल में हाइलाइट किए गए प्रोटिओमिक अनुप्रयोगों का उदाहरण देते हैं। इस काम के कुछ हिस्सों को पुरस्कार संख्या आईओएस -1832968 करियर (पीएन को), पुरस्कार संख्या आर 35 जीएम 124755 (पीएन को) के तहत राष्ट्रीय स्वास्थ्य संस्थान, मैरीलैंड विश्वविद्यालय-राष्ट्रीय कैंसर संस्थान साझेदारी कार्यक्रम (पीएन को), और कॉसमॉस क्लब फाउंडेशन अनुसंधान पुरस्कार (एबीबी और एलआरपी के लिए) द्वारा समर्थित किया गया था।
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Biologie du développement. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Biologie du développement. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Biologie du développement. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

