Cellelinjeveiledet massespektrometriproteomikk i det utviklende (froske) embryoet
Summary
Her beskriver vi en massespektrometribasert proteomisk karakterisering av cellelinjer med kjente vevsskjebner i virveldyret Xenopus laevis-embryoet .
Abstract
Karakterisering av molekylære hendelser som celler gir opphav til vev og organer øker et potensial for å bedre forstå normal utvikling og designe effektive rettsmidler for sykdommer. Teknologier som muliggjør nøyaktig identifisering og kvantifisering av ulike typer og et stort antall proteiner, vil fortsatt gi manglende informasjon om molekylære mekanismer som orkestrerer vev og organismeutvikling i rom og tid. Her presenterer vi en massespektrometribasert protokoll som muliggjør måling av tusenvis av proteiner i identifiserte cellelinjer i Xenopus laevis (frosk) embryoer. Tilnærmingen bygger på reproduserbare celleskjebnekart og etablerte metoder for å identifisere, fluorescerende merke, spore og prøveceller og deres avkom (kloner) fra denne modellen for virveldyrutvikling. Etter å ha samlet cellulært innhold ved hjelp av mikrosampling eller isolering av celler ved disseksjon eller fluorescensaktivert cellesortering, blir proteiner ekstrahert og behandlet for nedenfra og opp proteomisk analyse. Væskekromatografi og kapillær elektroforese brukes til å gi skalerbar separasjon for proteindeteksjon og kvantifisering med høyoppløselig massespektrometri (HRMS). Representative eksempler er gitt for proteomisk karakterisering av nevralvevsskjebner. Cellelinjestyrt HRMS-proteomikk kan tilpasses forskjellige vev og organismer. Det er tilstrekkelig følsomt, spesifikt og kvantitativt til å kikke inn i den romlige-temporale dynamikken til proteomet under vertebratutvikling.
Introduction
Vår forståelse av celledifferensiering og opprinnelsen til vev og organer er resultatet av flere tiår med forseggjorte målrettede skjermer av gener og deres produkter. Å øke vår kunnskap om alle biomolekylene og deres mengder under viktige cellulære hendelser vil bidra til å avdekke molekylære mekanismer som styrer romlig og tidsmessig mønster av virveldyrets kroppsplan. Teknologier som muliggjør molekylær forsterkning og sekvensering er nå i stand til rutinemessig å rapportere om et stort antall gener og transkripsjoner, og støtter hypotesedrevne studier i grunnleggende biologisk og translasjonsforskning. For å forstå utvikling av systemer, tar et komplekst forhold mellom transkripsjon og oversettelse til orde for direkte analyse av flere proteiner og deres posttranslasjonelle modifikasjoner. Global proteomikk ved bruk av in vitro biologiske systemer, slik som induserte pluripotente stamceller, begynte å avgrense mekanismer for vevsinduksjon 1,2. I komplekse organismer, som virveldyrembryoet, er utviklingen avhengig av morfogengradienter i sammenheng med rom og tid3. Det følger at å få kunnskap om proteomiske forandringer når celler skiller seg for å danne spesialiserte vev, for eksempel nevrale vev, gir en nøkkel til å låse opp molekylære programmer som kontrollerer normal og defekt utvikling og veilede neste generasjons terapi.
Virveldyret sørafrikansk klørfrosk (Xenopus laevis) er en veletablert modell innen celle- og utviklings-, nevro- og regenerativ biologi. Sir John Gurdons 2012 Nobelpris i fysiologi eller medisin 4,5 for oppdagelsen av pluripotens av den somatiske kjernen fremhevet betydningen av denne modellen for funn i grunnleggende og translasjonsstudier. Xenopus-embryoer utvikler seg eksternt til moren, og letter dermed direkte manipulering av celler, cellekloner og genuttrykk over ulike utviklingsstadier. Asymmetrisk pigmentering og stereotype celledelinger gjorde det mulig å kartlegge reproduserbare skjebnekart fra 16-6 og 32-celle 7,8 stadium embryo. For høyoppløselig massespektrometri (HRMS) basert proteomikk inkluderer ytterligere fordeler med modellen relativt stor størrelse (~ 1 mm i diameter), noe som gir rikelig proteininnhold for analyse (~ 130 μg i tidlige spaltningsstadiumembryoer, ~ 10 μg proteininnhold i enkeltceller i 16-celleembryoet) 9,10.
For tiden er HRMS den ledende teknologien som er valgt for å oppdage proteiner. Denne teknologien muliggjør direkte, sensitiv og spesifikk deteksjon og kvantifisering av flere, vanligvis hundrevis-til-tusenvis av forskjellige proteiner11. Bottom-up proteomikk av HRMS innebærer en rekke sammenkoblede trinn. Etter ekstraksjon fra celle-/vevsprøven fordøyes proteiner med et proteolytisk enzym, slik som trypsin (bottom-up proteomics). De resulterende peptidene separeres basert på deres forskjellige fysisk-kjemiske egenskaper, inkludert hydrofobicitet (reversert fasevæskekromatografi, LC), nettoladning (ionebytterkromatografi), størrelse (størrelsesekskluderingskromatografi) eller elektroforetisk mobilitet (kapillær elektroforese, CE). Peptider blir deretter ladet (ionisert), vanligvis ved bruk av elektrosprayionisering (ESI), og peptidioner detekteres og sekvenseres via gassfasefragmentering av tandem-HRMS. De resulterende peptiddataene kartlegges til proteomet til organismen som studeres. Med proteinspesifikk (proteotypisk) peptidionsignalintensitet korrelert med konsentrasjon, kan proteinkvantifisering utføres etikettfri eller etikettbasert (multipleksingskvantifisering). HRMS proteomikk gir en rik ressurs av informasjon om molekylær tilstand av systemet under studien, noe som gjør det mulig å generere hypoteser og oppfølging funksjonelle studier.
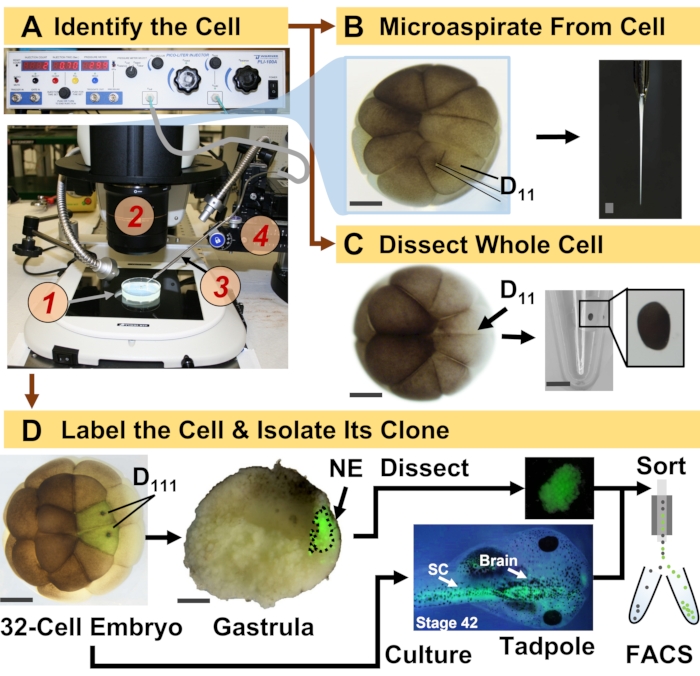
Figur 1: Spatiotemporalt skalerbar proteomikk som muliggjør cellelinjeveiledet HRMS-proteomikk i det utviklende (froske) embryoet. (A) Visualisering av prøven (1) ved hjelp av et stereomikroskop (2) for injeksjon av en identifisert celle (innfelt), ved bruk av en fabrikert mikropipette (3) under kontroll av et oversettelsestrinn (4). (B) Subcellulær prøvetaking av den identifiserte venstre D11-cellen i et 16-celleembryo. (C) Disseksjon av en hel D11 celle fra et 16-celle embryo. (D) Fluorescerende (grønn) sporing av venstre og høyre D111 progenier fra et 32-celleembryo for å lede disseksjon av nevralektoderm (NE) i gastrula (trinn 10) og isolering av etterkommervevet fra rumpetroll ved hjelp av FACS. Skalastenger: 200 μm for embryoer, 1,25 mm for hetteglasset. Tallene ble tilpasset med tillatelse fra referansene 15,19,21,59. Klikk her for å se en større versjon av denne figuren.
Protokollen som presenteres her muliggjør HRMS-basert kvantifisering av et stort antall proteiner i identifiserte celler/vev i utviklingen av X. laevis-embryoer. Tilnærmingen bygger på nøyaktig celleidentifikasjon, reproduserbare celleskjebnekart og etablerte metoder for å spore cellelinjer i denne biologiske modellen 6,7,8. Som vist i figur 1 studerer vi proteomer fra enkeltceller ved å bruke helcelledisseksjon eller kapillær mikrosampling for å aspirere cellulært innhold. Overvåking av avstamningen til en celle tillater oss å studere den spatiotemporale utviklingen av proteomet når celler danner vev under gastrulasjon. Celleavkommet er fluorescerende merket ved å injisere en fluorofor konjugert til inert dextran eller mRNA for fluorescerende protein (f.eks. grønt fluorescerende protein eller GFP). Det merkede avkommet isoleres ved ønskede utviklingstidspunkter. Under gastrulering kan cellekloner som er tett gruppert isoleres ved disseksjon. Etter gastrulering kan cellekloner distribueres i embryoet på grunn av migrerende bevegelser og kan isoleres fra dissosierte vev ved fluorescensaktivert cellesortering (FACS). Proteiner i disse cellene og vevene måles via bottom-up-proteomikk som bruker HPLC eller CE for separasjon og ESI-tandem HRMS for identifikasjon. Cellelinjestyrt HRMS-proteomikk er skalerbar til forskjellige cellestørrelser og linjer i embryoet og er spesifikk, sensitiv og kvantitativ. Gjennom utvalgte eksempler vist her, demonstrerer vi også at denne protokollen er skalerbar og bredt tilpasningsdyktig til forskjellige typer celler og cellelinjer.
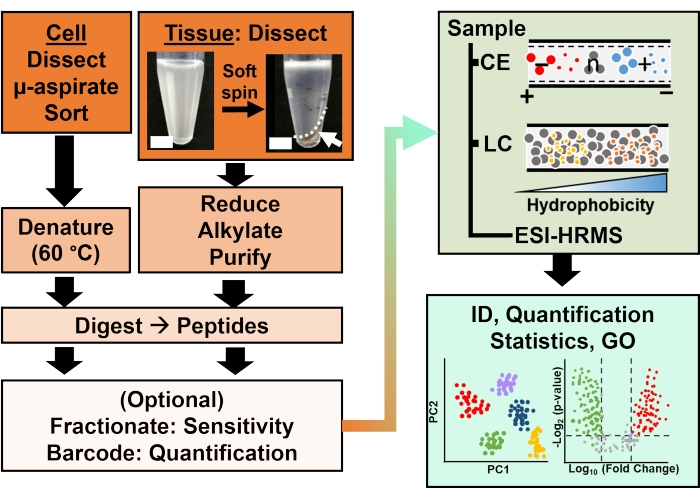
Figur 2: Den bioanalytiske arbeidsflyten. Mikrodisseksjon og kapillær aspirasjon, eller FACS muliggjorde prøvetaking av cellulært og klonalt proteininnhold. Uttømming av rikelig eggeplommeproteiner og separasjon ved kapillær elektroforese (CE) eller nano-flow væskekromatografi (LC) forbedret identifikasjon (ID) følsomhet ved bruk av elektrosprayionisering (ESI) høyoppløselig massespektrometri (HRMS). Kvantifisering avslørte dysregulering, og ga ny informasjon for hypotesedrevne studier i forbindelse med informasjon tilgjengelig fra genontologi (GO). Figurene ble bearbeidet med tillatelse frareferanse 15. Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
Denne protokollen muliggjør karakterisering av proteinuttrykk i identifiserte cellelinjer i embryoer av Xenopus-arten. Metodikken stammer fra HRMS, og kombinerer utsøkt spesifisitet i molekylær identifikasjon, evne til multiproteindeteksjon uten molekylære prober (vanligvis hundrevis til tusenvis av forskjellige proteiner) og en evne til kvantifisering. Tilpasningsevne til klassiske verktøy og arbeidsflyter i celle- og utviklingsbiologi (nevro) utvider HRMS-proteomikk til spennende applikasjoner, inkludert …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Vi er takknemlige for Jie Li (University of Maryland, College Park) for verdifulle diskusjoner om embryonal dissosiasjon og FACS. Vi takker Vi M. Quach og Camille Lombard-Banek for hjelp med prøvepreparering og datainnsamling i tidligere studier som eksemplifiserer de proteomiske applikasjonene som er fremhevet i denne protokollen. Deler av dette arbeidet ble støttet av National Science Foundation under tildelingsnummer IOS-1832968 KARRIERE (til P.N.), National Institutes of Health under prisnummer R35GM124755 (til P.N.), University of Maryland-National Cancer Institute Partnership Program (til P.N.), og COSMOS Club Foundation forskningspriser (til ABB og LRP).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Biologie du développement. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Biologie du développement. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Biologie du développement. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

