Протеомика масс-спектрометрии под контролем клеточных линий у развивающегося эмбриона (лягушки)
Summary
Здесь мы описываем основанную на масс-спектрометрии протеомную характеристику клеточных линий с известными тканевыми судьбами эмбриона позвоночных Xenopus laevis .
Abstract
Характеристика молекулярных событий, когда клетки дают начало тканям и органам, повышает потенциал для лучшего понимания нормального развития и разработки эффективных средств от болезней. Технологии, обеспечивающие точную идентификацию и количественную оценку различных типов и большого количества белков, предоставят до сих пор недостающую информацию о молекулярных механизмах, организующих развитие тканей и организмов в пространстве и времени. Здесь мы представляем протокол на основе масс-спектрометрии, который позволяет измерять тысячи белков в идентифицированных клеточных линиях эмбрионов Xenopus laevis (лягушек). Этот подход основан на воспроизводимых картах клеточной судьбы и установленных методах идентификации, флуоресцентной маркировки, отслеживания и отбора образцов клеток и их потомства (клонов) из этой модели развития позвоночных. После сбора клеточного содержимого с помощью микровыборки или выделения клеток путем диссекции или сортировки клеток, активированной флуоресценцией, белки извлекаются и обрабатываются для восходящего протеомного анализа. Жидкостная хроматография и капиллярный электрофорез используются для обеспечения масштабируемого разделения для обнаружения и количественного определения белка с помощью масс-спектрометрии высокого разрешения (HRMS). Приведены репрезентативные примеры протеомной характеристики клеток нервной ткани. Протеомика HRMS, управляемая клеточными линиями, адаптируется к различным тканям и организмам. Он достаточно чувствителен, специфичен и количественный, чтобы заглянуть в пространственно-временную динамику протеома во время развития позвоночных.
Introduction
Наше понимание дифференцировки клеток и генезиса тканей и органов является результатом десятилетий сложных целевых скринингов генов и их продуктов. Расширение наших знаний обо всех биомолекулах и их количествах во время важных клеточных событий поможет разгадать молекулярные механизмы, которые контролируют пространственную и временную структуру плана тела позвоночных. Технологии, обеспечивающие молекулярную амплификацию и секвенирование, теперь могут регулярно сообщать о большом количестве генов и транскриптов, поддерживая исследования, основанные на гипотезах, в фундаментальных биологических и трансляционных исследованиях. Чтобы понять развивающиеся системы, сложная взаимосвязь между транскрипцией и трансляцией требует прямого анализа нескольких белков и их посттрансляционных модификаций. Глобальная протеомика с использованием биологических систем in vitro, таких как индуцированные плюрипотентные стволовые клетки, начала очерчивать механизмы тканевой индукции 1,2. У сложных организмов, таких как эмбрион позвоночных, развитие зависит от градиентов морфогена в контексте пространства и времени3. Из этого следует, что получение знаний о протеомных изменениях по мере того, как клетки дифференцируются с образованием специализированных тканей, таких как нервные ткани, дает ключ к разблокированию молекулярных программ, контролирующих нормальное и дефектное развитие, и направляет терапию следующего поколения.
Позвоночная южноафриканская когтистая лягушка (Xenopus laevis) является хорошо зарекомендовавшей себя моделью в клеточной и развивающейся, нейро- и регенеративной биологии. Нобелевская премия сэра Джона Гердона по физиологии и медицине 2012 года 4,5 за открытие плюрипотентности соматического ядра подчеркнула важность этой модели для открытий в фундаментальных и трансляционных исследованиях. Эмбрионы Xenopus развиваются внешне по отношению к матери, тем самым облегчая прямое манипулирование клетками, клеточными клонами и экспрессией генов на различных стадиях развития. Асимметричная пигментация и стереотипное деление клеток позволили составить карту воспроизводимых карт судьбы 16-6 и 32-клеточногоэмбриона на 7,8 стадии. Для протеомики на основе масс-спектрометрии высокого разрешения (HRMS) дополнительные преимущества модели включают относительно большой размер (~1 мм в диаметре), что дает обильное содержание белка для анализа (~130 мкг у эмбрионов на ранней стадии расщепления, ~10 мкг белка в одиночных клетках 16-клеточного эмбриона)9,10.
В настоящее время HRMS является ведущей технологией выбора для обнаружения белков. Эта технология позволяет прямо, чувствительно и специфически обнаруживать и количественно определять множественные, обычно от сотен до тысяч различныхбелков11. Восходящая протеомика HRMS включает в себя ряд взаимосвязанных этапов. После экстракции из образца клетки / ткани белки перевариваются протеолитическим ферментом, таким как трипсин (восходящая протеомика). Полученные пептиды разделяют на основе их различных физико-химических свойств, включая гидрофобность (обращенно-фазовая жидкостная хроматография, LC), суммарный заряд (ионообменная хроматография), размер (эксклюзионная хроматография) или электрофоретическую подвижность (капиллярный электрофорез, CE). Затем пептиды заряжаются (ионизируются), как правило, с помощью электрораспыления ионизации (ESI), а пептидные ионы обнаруживаются и секвенируются посредством газофазной фрагментации с помощью тандемной HRMS. Полученные пептидные данные сопоставляются с протеомом исследуемого организма. При том, что интенсивность сигнала пептид-ионов специфического белка (протеотипа) коррелирует с концентрацией, количественное определение белка может быть выполнено без меток или на основе меток (мультиплексирующее количественное определение). Протеомика HRMS дает богатый источник информации о молекулярном состоянии исследуемой системы, что позволяет генерировать гипотезы и проводить последующие функциональные исследования.
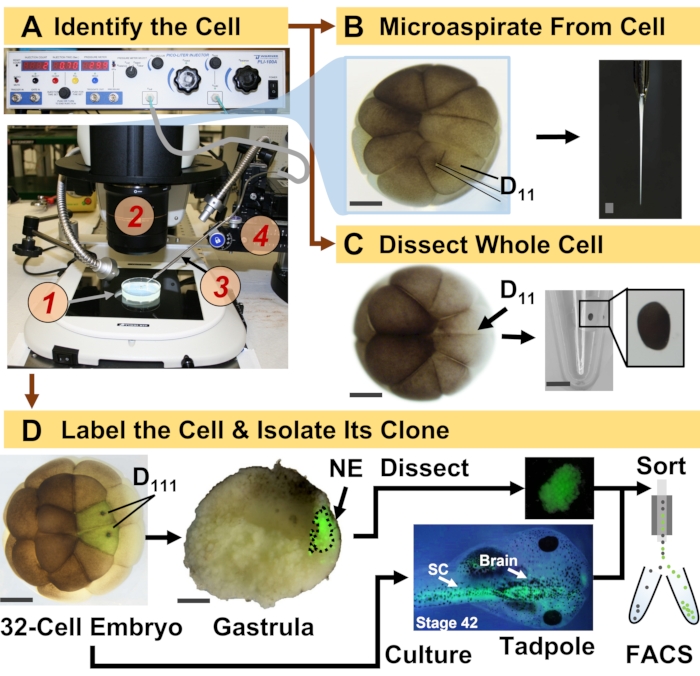
Рисунок 1: Пространственно-временная масштабируемая протеомика, обеспечивающая протеомику HRMS, управляемую клеточной линией, в развивающемся эмбрионе (лягушке). (A) Визуализация образца (1) с использованием стереомикроскопа (2) для инъекции идентифицированной клетки (врезка) с использованием изготовленной микропипетки (3) под контролем трансляционной стадии (4). (B) Субклеточный отбор идентифицированных левых клеток D11 в 16-клеточном эмбрионе. (C) Рассечение целой клетки D11 из 16-клеточного эмбриона. (D) Флуоресцентное (зеленое) отслеживание левого и правого потомства D111 от 32-клеточного эмбриона для направления рассечения невральной эктодермы (NE) в гаструле (стадия 10) и выделения ткани-потомка от головастика с использованием FACS. Масштабные линейки: 200 мкм для эмбрионов, 1,25 мм для флакона. Рисунки были адаптированы с разрешения ссылок 15,19,21,59. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Представленный здесь протокол позволяет количественно оценить большое количество белков в идентифицированных клетках/тканях развивающихся эмбрионов X. laevis на основе HRMS. Подход основан на точной идентификации клеток, воспроизводимых картах клеточной судьбы и установленных методологиях отслеживания клеточных линий в этой биологической модели 6,7,8. Как показано на рисунке 1, мы изучаем протеомы из отдельных клеток, используя диссекцию целых клеток или капиллярный микропробоотбор для аспирации клеточного содержимого. Наблюдение за происхождением клетки позволяет нам изучать пространственно-временную эволюцию протеома по мере того, как клетки образуют ткани во время гаструляции. Клеточное потомство флуоресцентно маркируют путем введения флуорофора, конъюгированного с инертным декстраном или мРНК для флуоресцентного белка (например, зеленого флуоресцентного белка или GFP). Меченое потомство изолируют в желаемые моменты развития. Во время гаструляции клоны клеток, которые плотно сгруппированы, могут быть выделены путем рассечения. После гаструляции клеточные клоны могут быть распределены внутри эмбриона за счет миграционных движений и могут быть выделены из диссоциированных тканей с помощью флуоресцентно-активированной сортировки клеток (FACS). Белки в этих клетках и тканях измеряются с помощью восходящей протеомики с использованием ВЭЖХ или КЭ для разделения и тандемной HRMS ESI для идентификации. Протеомика HRMS, управляемая клеточными линиями, масштабируется для различных размеров клеток и линий внутри эмбриона и является специфической, чувствительной и количественной. На отдельных примерах, показанных здесь, мы также демонстрируем, что этот протокол является масштабируемым и широко адаптируемым к различным типам клеток и клеточных линий.
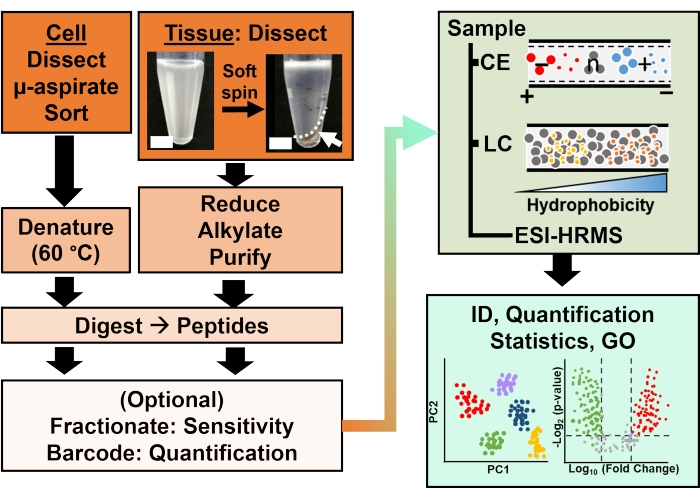
Рисунок 2: Биоаналитический рабочий процесс. Микродиссекция и капиллярная аспирация, или FACS, облегчают отбор проб клеточного и клонального белка. Истощение обильных белков желтка и разделение с помощью капиллярного электрофореза (CE) или нанопоточной жидкостной хроматографии (LC) с повышенной чувствительностью идентификации (ID) с использованием масс-спектрометрии высокого разрешения (HRMS) с помощью ионизации электрораспылением (ESI). Количественная оценка выявила дисрегуляцию, предоставив новую информацию для исследований, основанных на гипотезах, в сочетании с информацией, доступной из онтологии генов (GO). Рисунки были адаптированы с разрешения ссылки15. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
Этот протокол позволяет охарактеризовать экспрессию белка в идентифицированных клеточных линиях эмбрионов вида Xenopus . Методология, основанная на HRMS, сочетает в себе исключительную специфичность в молекулярной идентификации, способность к обнаружению нескольких белков без молек…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Мы благодарны Цзе Ли (Университет Мэриленда, Колледж-Парк) за ценные дискуссии об эмбриональной диссоциации и FACS. Мы благодарим Ви М. Куача и Камиллу Ломбард-Банек за помощь в подготовке образцов и сборе данных в предыдущих исследованиях, иллюстрирующих протеомные приложения, выделенные в этом протоколе. Часть этой работы была поддержана Национальным научным фондом под номером награды IOS-1832968 CAREER (для P.N.), Национальными институтами здравоохранения под номером R35GM124755 (для P.N.), Партнерской программой Университета Мэриленда и Национального института рака (для P.N.) и исследовательскими наградами Фонда клуба COSMOS (для ABB и L.R.P.).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Biologie du développement. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Biologie du développement. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Biologie du développement. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

