Detección de alto rendimiento de aislados microbianos con impacto en la salud de Caenorhabditis elegans
Summary
Los microbios intestinales pueden afectar positiva o negativamente la salud de su huésped a través de mecanismos específicos o conservados. Caenorhabditis elegans es una plataforma conveniente para detectar tales microbios. El presente protocolo describe el cribado de alto rendimiento de 48 aislados bacterianos para determinar su impacto en la resistencia al estrés de los nematodos, utilizados como sustitutos de la salud de los gusanos.
Abstract
Con su pequeño tamaño, corta vida útil y genética fácil, Caenorhabditis elegans ofrece una plataforma conveniente para estudiar el impacto de los aislados microbianos en la fisiología del huésped. También fluoresce en azul cuando muere, proporcionando un medio conveniente para identificar la muerte. Esta propiedad se ha explotado para desarrollar ensayos de supervivencia de C. elegans (LFASS) de alto rendimiento y sin etiquetas. Estos implican el registro de fluorescencia de lapso de tiempo de poblaciones de gusanos colocadas en placas de múltiples pocillos, de las cuales se puede derivar el tiempo medio de muerte de la población. El presente estudio adopta el enfoque LFASS para detectar múltiples aislados microbianos a la vez para detectar los efectos sobre la susceptibilidad de C. elegans al calor severo y al estrés oxidativo. Aquí se informa de esta tubería de detección microbiana, que se puede utilizar en particular para preseleccionar probióticos, utilizando resistencia severa al estrés como un indicador de la salud del huésped. El protocolo describe cómo cultivar colecciones aisladas de microbiota intestinal de C. elegans y poblaciones de gusanos sincrónicos en matrices de pozos múltiples antes de combinarlos para los ensayos. El ejemplo proporcionado cubre la prueba de 47 aislados bacterianos y una cepa de control en dos cepas de gusanos, en dos ensayos de estrés en paralelo. Sin embargo, la tubería de enfoque es fácilmente escalable y aplicable a la selección de muchas otras modalidades. Por lo tanto, proporciona una configuración versátil para estudiar rápidamente un paisaje multiparamétrico de condiciones biológicas y bioquímicas que afectan la salud de C. elegans.
Introduction
El cuerpo humano alberga un estimado de 10-100 billones de células microbianas vivas (bacterias, hongos arquea), que se encuentran principalmente en el intestino, la piel y los ambientes de la mucosa1. En un estado saludable, estos proporcionan beneficios a su huésped, incluida la producción de vitaminas, la maduración del sistema inmunológico, la estimulación de las respuestas inmunes innatas y adaptativas a los patógenos, la regulación del metabolismo de las grasas, la modulación de las respuestas al estrés y más, con un impacto en el crecimiento y el desarrollo, el inicio de la enfermedad y el envejecimiento 2,3,4,5 . La microbiota intestinal también evoluciona considerablemente a lo largo de la vida. La evolución más drástica ocurre durante la infancia y la primera infancia6, pero también ocurren cambios significativos con la edad, incluyendo una disminución en la abundancia de Bifidobacterium y un aumento en las especies de Clostridium, Lactobacillus, Enterobacteriaceae y Enterococcus 7. El estilo de vida puede alterar aún más la composición microbiana intestinal que conduce a la disbiosis (pérdida de bacterias beneficiosas, crecimiento excesivo de bacterias oportunistas), lo que resulta en diversas patologías como la enfermedad inflamatoria intestinal, la diabetes y la obesidad5, pero también contribuye a las enfermedades de Alzheimer y Parkinson 8,9,10,11.
Esta comprensión ha contribuido críticamente a refinar el concepto del eje intestino-cerebro (GBA), donde las interacciones entre la fisiología intestinal (ahora incluyendo los microbios dentro de ella) y el sistema nervioso se consideran el principal regulador del metabolismo animal y las funciones fisiológicas12. Sin embargo, el papel preciso de la microbiota en la señalización intestino-cerebro y los mecanismos de acción asociados están lejos de ser completamente comprendidos13. Dado que la microbiota intestinal es un determinante clave del envejecimiento saludable, la forma en que las bacterias modulan el proceso de envejecimiento se ha convertido en un tema de intensa investigación y controversia 6,14,15.
Con la demostración de que el gusano redondo Caenorhabditis elegans alberga una microbiota intestinal de buena fe dominada, como en otras especies, por Bacteroidetes, Firmicutes y Actinobacteria 16,17,18,19,20, su rápido aumento como plataforma experimental para estudiar las interacciones comensales huésped-intestino21,22,23,24 ,25,26 ha ampliado significativamente nuestro arsenal de investigación26,27,28,29. En particular, los enfoques experimentales de alto rendimiento disponibles para que C. elegans estudien las interacciones gen-dieta, gen-fármaco, gen-patógeno, etc., se pueden adaptar para explorar rápidamente cómo los aislados bacterianos y los cócteles afectan la salud y el envejecimiento de C. elegans.
El presente protocolo describe una tubería experimental para examinar a la vez matrices de aislados o mezclas bacterianas colocadas en placas de múltiples pocillos para detectar efectos sobre la resistencia al estrés de C. elegans como un indicador de la salud, que puede usarse para identificar probióticos. Detalla cómo cultivar grandes poblaciones de gusanos y manejar matrices bacterianas en formatos de placas de 96 y 384 pocillos antes de procesar gusanos para el análisis automatizado de resistencia al estrés utilizando un lector de placas de fluorescencia (Figura 1). El enfoque se basa en ensayos automatizados de supervivencia sin etiquetas (LFASS)30 que explotan el fenómeno de la fluorescencia de muerte31, mediante el cual los gusanos moribundos producen una explosión de fluorescencia azul que puede usarse para determinar el momento de la muerte. La fluorescencia azul es emitida por ésteres glucosílicos de ácido antranílico almacenados en gránulos intestinales de C. elegans (un tipo de orgánulo relacionado con lisosomas), que estallan cuando se desencadena una cascada necrótica en el intestino del gusano al morir31.
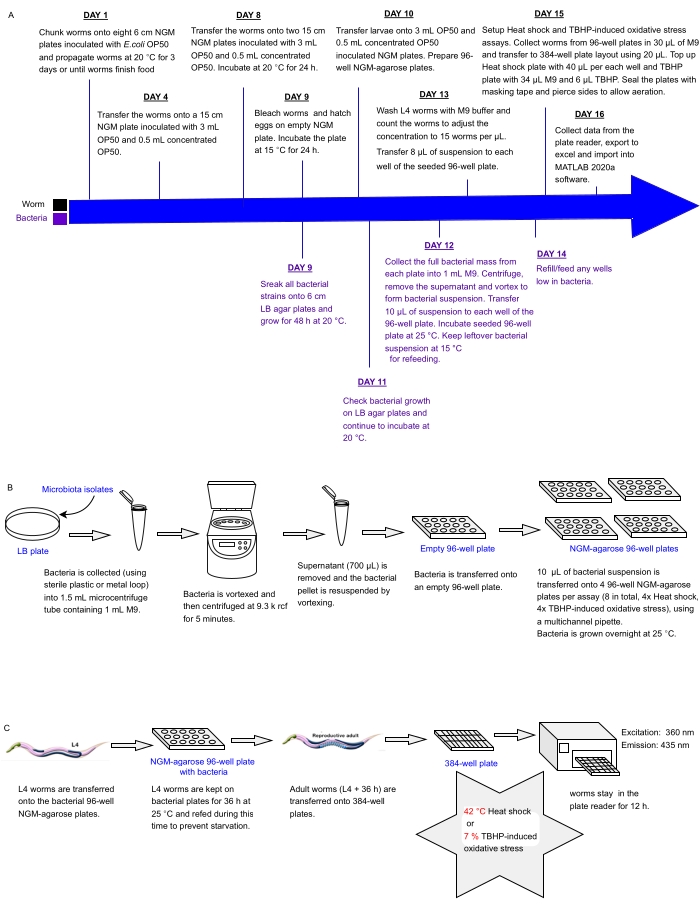
Figura 1: Flujo de trabajo experimental para el cribado de alto rendimiento de aislados bacterianos con impacto en la resistencia de C. elegans al estrés . (A) Línea de tiempo para el mantenimiento de gusanos y bacterias y la configuración del ensayo. (B) Configuración y manejo de la matriz de placas bacterianas de 96 pocillos. (C) Configuración de placa de tornillo sin fin de 384 pocillos. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
C. elegans ofrece muchas ventajas para examinar rápidamente múltiples parámetros experimentales a la vez, debido a su pequeño tamaño, transparencia, rápido desarrollo, corta vida útil, bajo costo y facilidad de manejo. Su genoma, plan corporal, sistema nervioso, intestino y microbioma considerablemente más simples, pero lo suficientemente complejos y similares a los humanos, lo convierten en un poderoso modelo preclínico, donde se puede obtener una visión mecanicista mientras se prueba la eficacia o to…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Agradecemos al CGC Minnesota (Madison, EUA, NIH – P40 OD010440) por proporcionar cepas de gusanos y OP50 y al Pr. Hinrich Schulenburg (CAU, Kiel, Alemania) por proporcionar todos los aislados microbianos ambientales representados aquí. Este trabajo fue financiado por una subvención UKRI-BBSRC a AB (BB / S017127 / 1). JM está financiado por una beca de doctorado FHM de la Universidad de Lancaster.
Materials
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
References
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood – Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer’s disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson’s disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio – The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer’s disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson’s disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).

