AMEBaS: Automatisk mittlinjeextraktion och bakgrundssubtraktion av ratiometrisk fluorescenstime-lapses av polariserade enskilda celler
Summary
Nuvarande metoder för att analysera den intracellulära dynamiken hos polariserade enskilda celler är ofta manuella och saknar standardisering. Detta manuskript introducerar en ny bildanalyspipeline för att automatisera mittlinjeextraktion av enstaka polariserade celler och kvantifiera spatiotemporalt beteende från time lapses i ett användarvänligt onlinegränssnitt.
Abstract
Cellpolaritet är ett makroskopiskt fenomen som etableras av en samling rumsligt koncentrerade molekyler och strukturer som kulminerar i uppkomsten av specialiserade domäner på subcellulär nivå. Det är förknippat med att utveckla asymmetriska morfologiska strukturer som ligger till grund för viktiga biologiska funktioner som celldelning, tillväxt och migration. Dessutom har störningen av cellpolariteten kopplats till vävnadsrelaterade sjukdomar som cancer och magdysplasi.
Nuvarande metoder för att utvärdera den spatiotemporala dynamiken hos fluorescerande rapportörer i enskilda polariserade celler involverar ofta manuella steg för att spåra en mittlinje längs cellernas huvudaxel, vilket är tidskrävande och benäget för starka biaser. Dessutom, även om ratiometrisk analys kan korrigera den ojämna fördelningen av reportermolekyler med hjälp av två fluorescenskanaler, är bakgrundssubtraktionstekniker ofta godtyckliga och saknar statistiskt stöd.
Detta manuskript introducerar en ny beräkningspipeline för att automatisera och kvantifiera det spatiotemporala beteendet hos enskilda celler med hjälp av en modell av cellpolaritet: pollenrör/rothårstillväxt och cytosolisk jondynamik. En trestegsalgoritm utvecklades för att bearbeta ratiometriska bilder och extrahera en kvantitativ representation av intracellulär dynamik och tillväxt. Det första steget segmenterar cellen från bakgrunden och skapar en binär mask med hjälp av en tröskelteknik i pixelintensitetsutrymmet. Det andra steget spårar en väg genom cellens mittlinje genom en skeletteringsoperation. Slutligen tillhandahåller det tredje steget bearbetade data som en ratiometrisk timelapse och ger en ratiometrisk kymograf (dvs. en 1D-rumslig profil över tid). Data från ratiometriska bilder tagna med genetiskt kodade fluorescerande rapportörer från växande pollenrör användes för att jämföra metoden. Denna pipeline möjliggör snabbare, mindre partisk och mer exakt representation av den spatiotemporala dynamiken längs mittlinjen av polariserade celler, vilket främjar den kvantitativa verktygslådan som finns tillgänglig för att undersöka cellpolaritet. AMEBaS Python-källkoden finns på: https://github.com/badain/amebas.git
Introduction
Cellpolaritet är en grundläggande biologisk process där den samordnade verkan av en samling rumsligt koncentrerade molekyler och strukturer kulminerar i etableringen av specialiserade morfologiska subcellulära domäner1. Celldelning, tillväxt och migration är beroende av sådana polaritetsplatser, medan dess förlust har associerats med cancer i epitelvävnadsrelaterade sjukdomar2.
Apiskt växande celler är ett dramatiskt exempel på polaritet, där polaritetsstället vid spetsen vanligtvis omorienteras till extracellulära signaler3. Dessa inkluderar utvecklande neuriter, svamphyfer, rothår och pollenrör, där flera cellulära processer visar uttalade skillnader från cellens spets mot skaftet. I pollenrör, i synnerhet, är aktinpolymerisation, vesikeltrafik och jonkoncentrationer markant polariserade, vilket visar spetsfokuserade gradienter4. Pollenrör är de manliga gametofyterna av blommande växter och är ansvariga för att leverera spermierna till ägget genom att växa uteslutande vid toppen av cellen med en av de snabbaste tillväxthastigheterna som är kända för en enda cell. De spetsfokuserade gradienterna av joner som kalcium 5 (Ca2+) och protoner 6 (H+) spelar en viktig roll för att upprätthålla pollenrörstillväxten, vilket är avgörande för att uppnå dess huvudsakliga biologiska funktion som kulminerar i en dubbelbefruktning 5,6. Kvantitativa metoder för att analysera den spatiotemporala dynamiken längs mittlinjen av apikalt växande celler är därför viktiga för att undersöka de cellulära och molekylära mekanismerna bakom polariserad tillväxt 7,8,9. Forskare använder ofta kymografer, dvs en matris som representerar pixelintensiteterna i cellens mittlinje (t.ex. kolumner) genom tiden (t.ex. rader), vilket gör det möjligt att visualisera celltillväxt och migration i diagonalen (figur 1). Trots sin användbarhet extraheras kymografer ofta genom att manuellt spåra mittlinjen, vilket är benäget för fördomar och mänskliga fel samtidigt som de är ganska mödosamma. Detta kräver en automatiserad metod för mittlinjeextraktion som är den första funktionen i pipelinen som introduceras häri, kallad AMEBaS: Automatic Midline Extraction and Background Subtraction of ratiometric fluorescence time lapses of polarized single cells.
När det gäller experimentella procedurer kan kvantitativ avbildning av joner/molekyler/arter av intresse i enskilda celler uppnås med genetiskt kodade fluorescerande sonder10. Bland de ständigt växande valen är ratiometriska sonder en av de mest exakta eftersom de avger olika fluorescensvåglängder när de är bundna/obundna till molekylerna av intresse11. Detta möjliggör korrigering av den rumsliga heterogeniteten i den intracellulära koncentrationen av sonden genom att använda förhållandet mellan två kanaler med deras kanalspecifika bakgrund subtraherad. Att uppskatta bakgrundströskeln för varje kanal och tidpunkt kan dock vara en komplex uppgift eftersom den ofta varierar i rymden på grund av effekter som skuggning, där bildens hörn har luminositetsvariation i förhållande till mitten, och i tid på grund av blekning av fluoroforen (fotoblekning)12. Även om det finns flera möjliga metoder, föreslår detta manuskript att bakgrundsintensiteten bestäms automatiskt med hjälp av segmenteringströskeln som erhålls med Isodata-algoritmen13, som sedan jämnas ut över bildrutor genom polynomregression som standard. Rumsliga komponenter som härrör från fluorescensheterogenitet som inte är relaterade till målcellen som avlägsnades i12 ignorerades dock med denna metod. Automatisk trösklar kan utföras med flera metoder, men Isodata-algoritmen gav de bästa resultaten empiriskt. Således är automatisk subtraktion av bakgrundsvärden och kvotmetrisk beräkning den andra huvudfunktionen hos AMEBaS (figur 1), som tillsammans tar emot en stapel av tvåkanaliga fluorescensmikroskopibilder, uppskattar cellens mittlinje och den kanalspecifika bakgrunden och matar ut kymografer för båda kanalerna och deras förhållande (huvudutgång #1) efter bakgrundssubtraktion, utjämning och avlägsnande av extremvärden, tillsammans med en stapel med ratiometriska bilder (huvudutgång #2).
AMEBaS testades med fluorescenstidsintervall av växande Arabidopsis pollenrör erhållna under ett mikroskop, antingen med Ca2+ (CaMeleon)8 eller pH (pHluorin)6 ratiometriska sensorer uttryckta under den pollenspecifika LAT52-promotorn. Bilder från varje kanal togs var 4:e sekund kopplad till ett inverterat mikroskop, en frontbelyst kamera (2560 pixlar × 2160 pixlar, pixelstorlek 6,45 μm), en fluorescensbelysare och en objektivlins för nedsänkning i vatten 63x, 1,2NA. Filterinställningar som användes för CaMeleon var: excitation 426-450 nm (CFP) och 505-515 nm (YFP), emission 458-487 nm (CFP) och 520-550 nm (YFP), medan för pHluorin, excitation 318-390 nm (DAPI) och 428-475 nm (FITC), emission 435-448 nm (DAPI) och 523-536 nm (FITC). En komplett datauppsättning lades till för testning vid Zenodo (DOI: 10.5281/zenodo.7975350)14.
Dessutom testades pipelinen med rothårsdata, där avbildning utfördes med ett light sheet-mikroskop (SPIM) som tidigare beskrivits 15,16 med Arabidopsis rothår som uttryckte den genetiskt kodade Ca2+-reportern NES-YC3.6 under kontroll av UBQ10-promotorn17. Den hemmagjorda LabView-programvaran som styrde kamerainsamlingen, provöversättningen och slutaren av ljusarksmikroskopet gjorde det möjligt att observera de två cpVenus- och CFP-kanalerna, men också att visualisera deras förhållande i realtid. Varje bild av time-lapse representerade en maximal intensitetsprojektion (MIP) mellan de fluorescerande kanalerna cpVenus och CFP bilder erhållna från 15 skivor av provet med 3 μm mellanrum. Time-lapse cpVenus/CFP-kvoten för MIP sparades och användes direkt för AMEBaS-analysen.
Även om denna pipeline kan fungera med flera typer av växande och migrerande celler, var den speciellt utformad för att analysera växande celler som växer uteslutande i spetsen, såsom pollenrör, rothår och svamphyfer, där det finns en korrespondens mellan de icke-växande cytoplasmatiska regionerna mellan ramarna. När en sådan korrespondens inte finns bör användaren välja alternativet complete_skeletonization i steg 1.3.1.1 (se avsnittet Diskussion för mer information).
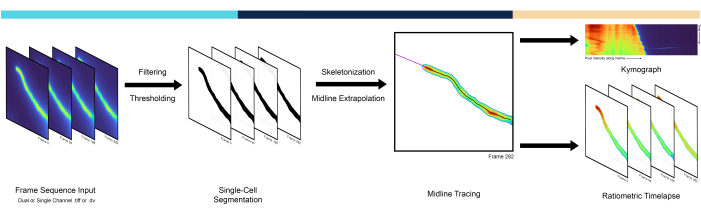
Bild 1: En översikt över pipelinearbetsflödet. AMEBaS-pipelinen analyserar och bearbetar mikroskopiska tidsförlopp i tre huvudsteg: Single-Cell Segmentation, Midline Trace och Kymograph Generation. Klicka här för att se en större version av denna figur.
Protocol
Representative Results
Discussion
Den nya metoden som presenteras här är ett kraftfullt verktyg för att effektivisera och automatisera analysen av fluorescensmikroskopibilder av polariserade celler. Nuvarande metoder som beskrivs i litteraturen, såsom ImageJ Kymograph-plugins, kräver manuell spårning av mittlinjen i den polariserade cellen av intresse, en uppgift som inte bara är tidskrävande utan också utsatt för mänskliga fel. Eftersom definitionen av mittlinjen i denna pipeline stöds av en numerisk metod18,19 som ut…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Författarna är tacksamma för FAPESP-anslagen 2015/22308-2, 2019/23343-7, 2019/26129-6, 2020/06744-5, 2021/05363-0, CNPq, NIH R01 grant GM131043 och NSF-bidragen MCB1714993, MCB1930165 för ekonomiskt stöd. Rothårsdata producerades med infrastrukturen och under överinseende av Prof. Andrea Bassi och Prof. Alex Costa.
Materials
| Github | Github | https://github.com/badain/amebas | |
| Google Colab | https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb |
References
- Drubin, D. G., Nelson, W. J. Origins of cell polarity. Cell. 84 (3), 335-344 (1996).
- Wodarz, A., Näthke, I. Cell polarity in development and cancer. Nature Cell Biology. 9 (9), 1016-1024 (2007).
- Palanivelu, R., Preuss, D. Pollen tube targeting and axon guidance: parallels in tip growth mechanisms. Trends in Cell Biology. 10 (12), 517-524 (2000).
- Portes, M. T., et al. . The Pollen Tube Oscillator: Integrating Biophysics and Biochemistry into Cellular Growth and Morphogenesis. Rhythms in Plants: Dynamic Responses in a Dynamic Environment. , (2015).
- Wudick, M. M., et al. CORNICHON sorting and regulation of GLR channels underlie pollen tube Ca2+ homeostasis. Science. 360 (6388), 533-536 (2018).
- Hoffmann, R. D., et al. Plasma membrane H+-ATPases sustain pollen tube growth and fertilization. Nature Communications. 11 (1), 1-15 (2020).
- Michard, E., et al. Glutamate receptor-like genes form Ca2+ channels in pollen tubes and are regulated by pistil D-serine. Science. 332 (6028), 434-437 (2011).
- Damineli, D. S., Portes, M. T., Feijó, J. A. Oscillatory signatures underlie growth regimes in Arabidopsis pollen tubes: computational methods to estimate tip location, periodicity, and synchronization in growing cells. Journal of Experimental Botany. 68 (12), 3267-3281 (2017).
- Li, K., et al. An optimized genetically encoded dual reporter for simultaneous ratio imaging of Ca2+ and H+ reveals new insights into ion signaling in plants. New Phytologist. 230 (6), 2292-2310 (2021).
- Sadoine, M., et al. Designs, applications, and limitations of genetically encoded fluorescent sensors to explore plant biology. Plant Physiology. 187 (2), 485-503 (2021).
- Grenzi, M., et al. Illuminating the hidden world of calcium ions in plants with a universe of indicators. Plant Physiology. 187 (2), 550-571 (2021).
- Munglani, G., Vogler, H., Grossniklaus, U. Fast and flexible processing of large FRET image stacks using the FRET-IBRA toolkit. PLoS Computational Biology. 18 (4), 1009242 (2022).
- Ridler, T., Calvard, S. Picture thresholding using an iterative selection method. IEEE Transactions on Systems, Man, and Cybernetics. 8 (8), 630-632 (1978).
- Portes, M. T., Feijó, J. . Growing Arabidopsis pollen tubes expressing genetically encoded reporters for calcium and pH. , (2023).
- Candeo, A., Doccula, F. G., Valentini, G., Bassi, A., Costa, A. Light sheet fluorescence microscopy quantifies calcium oscillations in root hairs of Arabidopsis thaliana. Plant & Cell Physiology. 58 (7), 1161-1172 (2017).
- Romano Armada, N., et al. In vivo light sheet fluorescence microscopy of calcium oscillations in Arabidopsis thaliana. Methods in Molecular Biology. 1925, 87-101 (2019).
- Krebs, M., et al. FRET-based genetically encoded sensors allow high-resolution live cell imaging of Ca2+ dynamics. The Plant Journal. 69 (1), 181-192 (2012).
- Lee, T. -. C., Kashyap, R. L., Chu, C. -. N. Building skeleton models via 3-D medial surface axis thinning algorithms. CVGIP: Graphical Models and Image Processing. 56 (6), 462-478 (1994).
- Nunez-Iglesias, J., Blanch, A. J., Looker, O., Dixon, M. W., Tilley, L. A new Python library to analyse skeleton images confirms malaria parasite remodelling of the red blood cell membrane skeleton. PeerJ. 6, 4312 (2018).

