Isolamento, caratterizzazione ed estrazione totale del DNA per l'identificazione di funghi endofiti in piante micoeterotrofe
Summary
Il presente articolo si propone di fornire protocolli dettagliati e adeguati per l’isolamento di funghi endofiti associati alle piante, la conservazione a lungo termine degli isolati, la caratterizzazione morfologica e l’estrazione del DNA totale per la successiva identificazione molecolare e analisi metagenomica.
Abstract
Le piante micoeterotrofe presentano una delle forme più estreme di dipendenza micorrizica, avendo perso totalmente la loro capacità autotrofica. Essenziali come qualsiasi altra risorsa vitale, i funghi a cui queste piante si associano intimamente sono essenziali per loro. Pertanto, alcune delle tecniche più rilevanti nello studio delle specie micoeterotrofe sono quelle che consentono di studiare i funghi associati, in particolare quelli che abitano le radici e gli organi sotterranei. In questo contesto, vengono comunemente applicate tecniche per l’identificazione di funghi endofiti coltura-dipendenti e coltura-indipendenti. L’isolamento degli endofiti fungini fornisce un mezzo per identificarli morfologicamente, analizzare la loro diversità e mantenere inoculi per applicazioni nella germinazione simbiotica dei semi di orchidea. Tuttavia, è noto che esiste una grande varietà di funghi non coltivabili che abitano i tessuti vegetali. Pertanto, le tecniche di identificazione molecolare indipendenti dalla coltura offrono una copertura più ampia della diversità e dell’abbondanza delle specie. Questo articolo si propone di fornire il supporto metodologico necessario per l’avvio di due procedure di indagine: una cultura-dipendente e una indipendente. Per quanto riguarda il protocollo coltura-dipendente, vengono descritti in dettaglio i processi di raccolta e mantenimento dei campioni vegetali dai siti di raccolta alle strutture di laboratorio, insieme all’isolamento dei funghi filamentosi dagli organi sotterranei e aerei delle piante micoeterotrofe, al mantenimento di una collezione di isolati, alla caratterizzazione morfologica delle ife mediante la metodologia di coltura a vetrino e all’identificazione molecolare dei funghi mediante estrazione totale del DNA. Comprendendo metodologie indipendenti dalla coltura, le procedure dettagliate includono la raccolta di campioni di piante per le analisi metagenomiche e l’estrazione del DNA totale dagli organi vegetali aclorofilliani utilizzando un kit commerciale. Infine, per le analisi vengono suggeriti anche protocolli di continuità (ad esempio, reazione a catena della polimerasi [PCR], sequenziamento) e vengono qui presentate tecniche.
Introduction
I funghi endofiti sono, per definizione, quelli che abitano l’interno degli organi e dei tessuti vegetali in caso di infezioni poco appariscenti (cioè senza causare danni al loro ospite)1,2. Questi funghi possono interagire in modo neutro o benefico con le piante ospiti, possono conferire resistenza agli agenti patogeni e alle condizioni ambientali sfavorevoli e possono contribuire alla sintesi di composti benefici per la pianta (ad esempio, fattori di crescita e altri fitormoni)1,3. Gli endofiti micorrizici sono funghi che stabiliscono associazioni micorriziche con la pianta, prendendo parte al trasferimento dei nutrienti4. Nelle Orchidaceae, l’interazione con gli endofiti micorrizici è fondamentale per la germinazione dei semi nella stragrande maggioranza delle specie, e per l’insediamento delle piantine in tutte le piante della famiglia5. In tali contesti, le orchidee micoeterotrofe rappresentano un caso di totale dipendenza nei confronti dei loro partner micorrizici, in quanto dipendono dal trasferimento di nutrienti minerali e composti carboniosi da parte di questi funghi durante il loro intero ciclo vitale6. Pertanto, l’isolamento e l’identificazione dei funghi associati è una base fondamentale quando si studiano le strategie di vita micoeterotrofe. Inoltre, poco si sa sul ruolo degli endofiti fungini nelle piante micoeterotrofe o anche sulla reale diversità di questi funghi 7,8.
Lo studio dei funghi endofiti può essere condotto attraverso diverse tecniche, tradizionalmente descritte come coltura-indipendenti o -dipendenti, ad esempio: (a) osservazione diretta, (b) isolamento fungino e identificazione morfologica e/o molecolare, e (c) estrazione totale del DNA dei tessuti vegetali e identificazione molecolare9. Nell’osservazione diretta (a), i funghi endofiti possono essere studiati mentre si trovano ancora all’interno di cellule e tessuti vegetali mediante microscopia ottica o elettronica9, poiché diversi protocolli di microscopia sono dettagliati da Pena-Passos et al.10. Con i metodi di isolamento (b), gli endofiti fungini possono essere caratterizzati in base alle loro colonie, ife e morfologia della struttura riproduttiva o di resistenza. Inoltre, attraverso tecniche di isolamento, è possibile condurre l’identificazione molecolare degli isolati attraverso l’estrazione del DNA, l’amplificazione delle sequenze di identificazione molecolare (codici a barre o impronte digitali) e il sequenziamento11. Quest’ultima tecnica (c) consente l’identificazione molecolare di funghi endofiti mediante estrazione di DNA all’interno dei tessuti vegetali (metabarcoding), seguita dalla preparazione e dal sequenziamento della libreria12.
Inoltre, gli isolati fungini possono essere applicati in prove di germinazione simbiotica, utilizzando semi di orchidee autotrofe o micoeterotrofe. Un esempio di tale applicazione è l’indagine condotta da Sisti et al.13, che descrive la germinazione e le fasi iniziali dello sviluppo del protocormo in Pogoniopsis schenckii, un’orchidea micoeterotrofica, in associazione con alcuni dei suoi isolati, comprendenti funghi endofiti non micorrizici. Il protocollo di germinazione simbiotica applicato è dettagliato e presentato in un video di Pena-Passos et al.10. L’isolamento dei funghi in associazione con diversi organi della pianta consente di concentrarsi su diversi obiettivi di indagine riguardanti la natura delle interazioni pianta-fungo (ad esempio, per comprendere gli aspetti ecologici o fisiologici dell’associazione, nonché le indagini sul trasferimento di nutrienti dai funghi alla pianta)9.
Le metodologie presentate nella sezione 1 si basano su una raccolta di campioni di organi sotterranei, in quanto questi organi presentano le maggiori difficoltà di raccolta, e sono di grande interesse poiché gli endofiti micorrizici li colonizzano. Tuttavia, entrambi i protocolli inclusi (passaggi 1.1 e 1.2) possono essere applicati ad altri organi micoeterotrofi della pianta (ad esempio, rizomi, steli floreali e frutti). La metodologia di raccolta descritta nella fase 1.1 è designata per l’isolamento di funghi endofiti (sezione 2) per la caratterizzazione morfologica (sezioni 4 e 5) e/o l’estrazione di DNA totale per l’identificazione dell’isolato (sezione 6). D’altra parte, la metodologia di raccolta descritta nella fase 1.2 è assegnata esclusivamente all’estrazione totale del DNA di tessuti vegetali per tecniche di metabarcoding (sezione 7). Nella sezione 3 vengono presentati quattro metodi per la conservazione e la conservazione dei funghi filamentosi, due per la conservazione a breve termine (3-6 mesi) e gli altri due adeguati per la conservazione a lungo termine (>1 anno). La caratterizzazione morfologica (sezioni 4 e 5) può essere associata all’identificazione molecolare per rafforzarla e fornire importanti informazioni sulla macro- e micromorfologia fungina. La figura 1 riassume le metodologie collettive descritte in seguito.
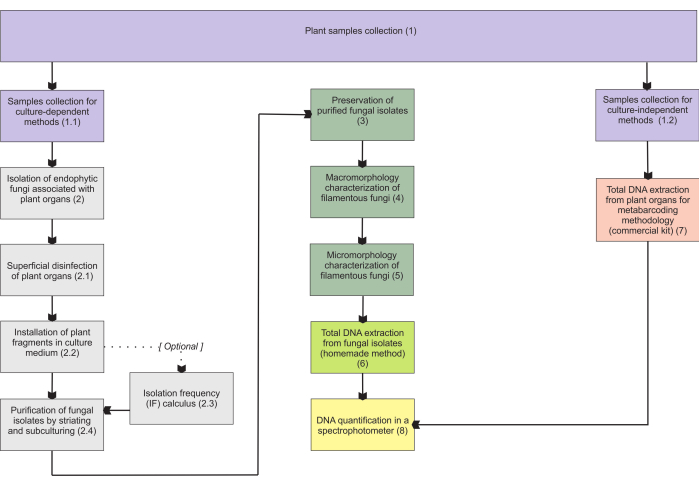
Figura 1: Sintesi schematica dei metodi presentati. Raccolta delle piante e isolamento, conservazione e identificazione molecolare dei funghi mediante metodologie coltura-dipendenti e indipendenti dalla coltura. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
La disinfestazione superficiale dei campioni vegetali è una delle fasi più critiche del protocollo presentato. È altamente auspicabile che non si contaminino le stoviglie PDA con gocce dell’ultimo lavaggio. I batteri sono spesso osservati come contaminanti nelle piastre di isolamento, di solito più dei funghi sporulanti presenti nell’aria, considerando che i batteri endofiti sono comuni anche all’interno dei tessuti vegetali 3,11. Pertanto, l’aggiunta di anti…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Si ringraziano i finanziamenti di FAPESP (2015/26479-6) e CNPq (447453/2014-9). JLSM ringrazia CNPq per le sovvenzioni alla produttività (303664/2020-7). MPP ringrazia Capes (borsa di studio per laurea magistrale, processo 88887.600591/2021-00) e CNPq.
Materials
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used – check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB – hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB – hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer – Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder – Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation – check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
References
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone’s Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).

