Isolering, karakterisering och total DNA-extraktion för att identifiera endofytiska svampar i mykoheterotrofa växter
Summary
Den aktuella artikeln syftar till att tillhandahålla detaljerade och adekvata protokoll för isolering av växtassocierade endofytiska svampar, långsiktigt bevarande av isolat, morfologisk karakterisering och total DNA-extraktion för efterföljande molekylär identifiering och metagenomiska analyser.
Abstract
Mykoheterotrofa växter utgör en av de mest extrema formerna av mykorrhizaberoende, eftersom de helt har förlorat sin autotrofa kapacitet. Lika viktiga som alla andra viktiga resurser, är svamparna som dessa växter är intimt förknippade med viktiga för dem. Därför är några av de mest relevanta teknikerna för att studera mykoheterotrofa arter de som möjliggör undersökning av associerade svampar, särskilt de som lever i rötter och underjordiska organ. I detta sammanhang används ofta tekniker för att identifiera odlingsberoende och odlingsoberoende endofytiska svampar. Att isolera svampendofyter ger ett sätt att morfologiskt identifiera dem, analysera deras mångfald och upprätthålla inokulering för tillämpningar i symbiotisk groning av orkidéfrön. Det är dock känt att det finns en stor variation av icke-odlingsbara svampar som lever i växtvävnader. Odlingsoberoende molekylära identifieringstekniker erbjuder således en bredare täckning av arternas mångfald och abundans. Syftet med denna artikel är att ge det metodstöd som krävs för att inleda två utredningsförfaranden: ett kulturberoende och ett oberoende. När det gäller det odlingsberoende protokollet beskrivs processerna för att samla in och underhålla växtprover från insamlingsplatser till laboratorieanläggningar, tillsammans med isolering av filamentösa svampar från underjordiska och luftorgan hos mykoheterotrofa växter, upprätthållande av en samling isolat, morfologiskt karakterisering av hyfer genom objektglasodlingsmetodik och molekylär identifiering av svampar genom total DNA-extraktion. De detaljerade procedurerna omfattar odlingsoberoende metoder och inkluderar insamling av växtprover för metagenomiska analyser och total DNA-extraktion från aklorofylllösa växtorgan med hjälp av ett kommersiellt kit. Slutligen föreslås även kontinuitetsprotokoll (t.ex. polymeraskedjereaktion [PCR], sekvensering) för analyser, och tekniker presenteras här.
Introduction
Endofytiska svampar är per definition de som lever i det inre av växtorgan och vävnader i oansenliga infektioner (dvs. utan att skada sin värd)1,2. Dessa svampar kan interagera neutralt eller fördelaktigt med värdväxter, kan ge resistens mot patogener och ogynnsamma miljöförhållanden och kan bidra till syntesen av nyttiga föreningar för växten (t.ex. tillväxtfaktorer och andra fytohormoner)1,3. Mykorrhizaendofyter är svampar som etablerar mykorrhizaföreningar med växten och deltar i näringsöverföring4. Hos Orchidaceae är interaktionen med mykorrhizaendofyter grundläggande för frögroning hos de allra flesta arter och plantetablering hos alla växter i familjen5. I sådana sammanhang representerar mykoheterotrofa orkidéer ett fall av totalt beroende av sina mykorrhizapartners, eftersom de är beroende av överföring av mineralnäringsämnen och kolföreningar från dessa svampar under hela deras livscykel6. Därför är isolering och identifiering av associerande svampar en grundläggande bas när man undersöker mykoheterotrofa livsstrategier. Dessutom är lite känt om svampendofyternas roller i mykoheterotrofa växter eller ens den verkliga mångfalden av dessa svampar 7,8.
Undersökningen av endofytiska svampar kan utföras med hjälp av olika tekniker, som traditionellt beskrivs som odlingsoberoende eller -beroende, till exempel: (a) direkt observation, (b) svampisolering och morfologisk och/eller molekylär identifiering, och (c) total DNA-extraktion av växtvävnader och molekylär identifiering9. I direkt observation (a) kan endofytiska svampar undersökas medan de fortfarande befinner sig i det inre av växtceller och vävnader med ljus- eller elektronmikroskopi9, eftersom olika mikroskopiprotokoll beskrivs i detalj av Pena-Passos et al.10. Genom isoleringsmetoder (b) kan svampendofyter karakteriseras enligt deras kolonier, hyfer och morfologi för reproduktions- eller resistensstruktur. Via isoleringstekniker är det också möjligt att utföra molekylär identifiering av isolat genom DNA-extraktion, amplifiering av molekylära identifieringssekvenser (streckkoder eller fingeravtryck) och sekvensering11. Den senare tekniken (c) möjliggör molekylär identifiering av endofytiska svampar per DNA-extraktion i det inre av växtvävnader (metabarcoding), följt av biblioteksförberedelse och sekvensering12.
Dessutom kan svampisolat användas i symbiotiska groningsförsök, med hjälp av frön från autotrofa eller mykoheterotrofa orkidéer. Ett exempel på en sådan tillämpning är den undersökning som utförts av Sisti et al.13, som beskriver groning och de inledande stadierna av protokormutveckling hos Pogoniopsis schenckii, en mykoheterotrofisk orkidé, tillsammans med några av dess isolat, som består av icke-mykorrhizaendofytiska svampar. Det tillämpade symbiotiska groningsprotokollet beskrivs i detalj och presenteras i en video av Pena-Passos et al.10. Att isolera svampar i samband med olika växtorgan möjliggör olika undersökningar av arten av växt-svampinteraktioner (t.ex. för att förstå antingen ekologiska eller fysiologiska aspekter av sambandet, samt undersökningar av näringsöverföringen från svampar till växten)9.
Metoderna som presenteras i avsnitt 1 är baserade på en insamling av underjordiska organprover, eftersom dessa organ utgör de svåraste att samla in, och de är av stort intresse eftersom mykorrhizaendofyter koloniserar dem. Båda de inkluderade protokollen (steg 1.1 och 1.2) kan dock tillämpas på andra mykoheterotrofa växtorgan (t.ex. rhizomer, blomstjälkar och frukter). Den insamlingsmetod som beskrivs i steg 1.1 är avsedd för isolering av endofytiska svampar (avsnitt 2) för morfologisk karakterisering (avsnitt 4 och 5) och/eller total DNA-extraktion för identifiering av isolat (avsnitt 6). Å andra sidan är den insamlingsmetod som beskrivs i steg 1.2 uteslutande avsedd för total DNA-extraktion av växtvävnader för metastreckkodningstekniker (avsnitt 7). I avsnitt 3 presenteras fyra metoder för lagring och konservering av filamentösa svampar, två för korttidslagring (3-6 månader) och de andra två lämpliga för långtidslagring (>1 år). Den morfologiska karakteriseringen (avsnitt 4 och 5) kan associeras med molekylär identifiering för att förstärka den och ge viktig information om svampars makro- och mikromorfologi. Figur 1 sammanfattar de samlade metoder som beskrivs nedan.
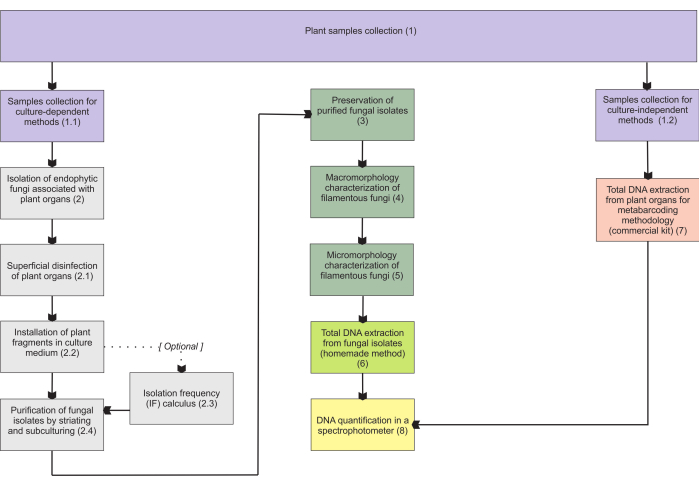
Figur 1: Schematisk sammanfattning av de presenterade metoderna. Växtinsamling och svampisolering, konservering och molekylär identifiering med odlingsberoende och -oberoende metoder. Klicka här för att se en större version av denna figur.
Protocol
Representative Results
Discussion
Den ytliga desinfektionen av växtprover är ett av de mest kritiska stegen i det presenterade protokollet. Ingen kontaminering i PDA-disken med droppar från den senaste tvätten är mycket önskvärd. Bakterier observeras ofta som föroreningar i isoleringsskålarna, vanligtvis mer än luftburna sporbildande svampar, med tanke på att endofytiska bakterier också är vanliga i växtvävnader 3,11. Därför är det viktigt att tillsätta antibiotika i odlingsmed…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Vi tackar för finansiering från FAPESP (2015/26479-6) och CNPq (447453/2014-9). JLSM tackar CNPq för produktivitetsbidrag (303664/2020-7). MPP tackar Capes (masterexamensstipendium, process 88887.600591/2021-00) och CNPq.
Materials
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used – check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB – hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB – hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer – Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder – Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation – check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
References
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone’s Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).

