ヒト幹細胞由来中脳ドーパミン作動性ニューロンの表現型プロファイリング
Summary
このプロトコルは免疫学の汚損および得られた顕微鏡のハイコンテント画像からのニューロンの表現型のプロフィールの生成に先行している人間の中脳のdopaminergicニューロンの細胞の培養を、記述し、遺伝か化学調節による表現型の変化の同一証明を可能にする。
Abstract
パーキンソン病(PD)は、中脳ドーパミン作動性(mDA)ニューロンの喪失を引き起こすさまざまな細胞生物学的プロセスに関連しています。現在の 多くのin vitro PD細胞モデルは複雑さに欠けており、複数の表現型を考慮していません。ヒト人工多能性幹細胞(iPSC)由来のmDAニューロンの表現型プロファイリングは、PD関連細胞タイプのニューロン表現型の範囲を同時に並行して測定することで、これらの欠点に対処することができます。ここでは、市販のヒトmDAニューロンから表現型プロファイルを取得して解析するためのプロトコルについて説明します。ニューロン特異的蛍光染色パネルを使用して、核、α-シヌクレイン、チロシンヒドロキシラーゼ(TH)、および微小管関連タンパク質2(MAP2)関連の表現型を可視化します。記載された表現型プロファイリングプロトコルは、384ウェルプレート、自動リキッドハンドリング、ハイスループット顕微鏡を使用するため、スケーラブルです。プロトコルの有用性は、健康なドナーmDAニューロンおよびロイシンリッチリピートキナーゼ2(LRRK2)遺伝子のPD結合G2019S変異を有するmDAニューロンを用いて例証される。両細胞株をLRRK2キナーゼ阻害剤PFE-360で処理し、表現型の変化を測定した。さらに、クラスタリングまたは機械学習主導の教師あり分類手法を使用して、多次元表現型プロファイルを分析する方法を示します。記載されたプロトコルは、神経疾患のモデリングに取り組んでいる研究者や、ヒトニューロンにおける化合物効果の研究に特に興味を引くでしょう。
Introduction
パーキンソン病(PD)では、さまざまな細胞生物学的プロセスが妨げられています。例えば、ミトコンドリアの機能不全、酸化ストレス、タンパク質分解の欠陥、小胞輸送の破壊、およびエンドリソソーム機能は、中脳ドーパミン作動性(mDA)ニューロンの喪失と関連しており、PD1で一般的に観察されます。したがって、PDには、互いに相互作用して悪化する可能性のある複数の疾患メカニズムが関与しているようです。この機構の相互作用を調査する有用な方法の1つは、中脳ドーパミン作動性(mDA)ニューロンの包括的な表現型フィンガープリントまたはプロファイルの作成です。
表現型プロファイリングは、測定可能な特性のコレクションに基づいてサンプルのプロファイルを作成するアプローチであり、次に、このプロファイルに基づいてサンプルに関する予測を行うことが含まれます2,3。プロファイリングの目的は、多様な特徴を捉えることであり、その一部はこれまで疾患や治療と関連付けられていなかった可能性がある3。その結果、プロファイリングにより、予期しない生物学的プロセスを明らかにすることができます。表現型プロファイリングは、通常、蛍光染色された細胞に依存しており、表現型プロファイルを作成するために、セルペインティングなどの標準化されたアッセイが開発されています4。最近では、表現型プロファイリングは、例えば、低分子の特性評価や、患者由来の線維芽細胞のみに基づくPDサブタイプの正確な予測に適用されています5,6。これらの進歩にもかかわらず、表現型プロファイリングは、LRRK2 G2019SなどのPD関連変異を発現するヒト人工多能性幹細胞(iPSC)由来のmDAニューロンなど、有糸分裂後の分化細胞に適用されることはめったにありません。iPS細胞由来モデルの重要な課題には、分化バッチまたは遺伝子型にわたって微妙または可変の病理学的特徴が存在すること、および単離されたPD表現型が疾患の複雑さを完全に捉えていないという事実が含まれます。さらに、iPS細胞の神経モデルは生理学的に関連性があるが、技術的な複雑さが懸念されるため、PD創薬プロセスで使用されることはほとんどない7,8。
私たちは以前、遺伝的および化合物による表現型の変化に敏感なヒトmDAニューロンにおける複数のPD関連の病態生理学的表現型を測定するための堅牢な方法論を開発しました9。本稿では、mDAニューロンから表現型プロファイルを作成するために、この方法論をさらに最適化したバージョンについて詳しく説明します(図1)。このプロトコルには、高品質のmDAニューロンの使用や技術的な再現性など、前述の表現型プロファイリングアプローチに比べていくつかの利点があります。このプロトコルは、化学的摂動後の生理学的に関連する有糸分裂後mDAニューロンの表現型プロファイリングを、初めて拡張可能な方法で可能にします。完全に分化して凍結保存されたmDAニューロンが市販されており、バッチ間の分化のばらつきが大幅に減少しています。第二に、明確に定義された実験計画(培養期間やエッジウェルの回避など)、自動リキッドハンドリング、自動顕微鏡を使用することで、技術的なばらつきをさらに減らすことができます。さらに、教師なしクラスタリングまたは教師あり分類アプローチを使用した表現型プロファイル分析の最初のステップをここで概説し、表現型プロファイリングデータを分析する方法を示します。このプロトコルは、遺伝的または化学的摂動によって引き起こされるmDAニューロンの表現型の変化に関心のある研究者にとって、特に、スクリーニングキャンペーン中など、非常にスケーラブルな研究セットアップが必要な場合、または毒性作用を決定するために少数の化合物の効果を研究する場合などに使用されます。要約すると、ヒトニューロンの表現型プロファイリングの応用は、複雑な疾患関連の表現型を研究し、薬剤候補の細胞効果を特徴付けるための貴重な技術であると予想されます。
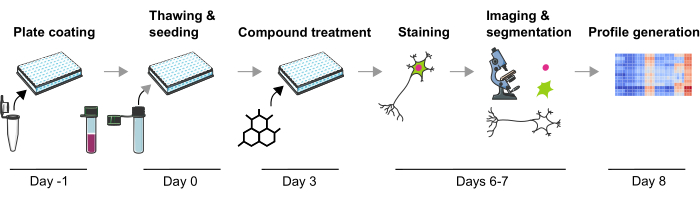
図1:ヒトiPS細胞由来のmDAニューロンから画像ベースの表現型プロファイルを生成するための実験プロトコルの概略図。 この図の拡大版をご覧になるには、ここをクリックしてください。
Protocol
Representative Results
Discussion
表現型プロファイリングは、蛍光染色、顕微鏡、画像解析を適用することにより、細胞内の多数の表現型を測定する技術です3。表現型プロファイルを取得し、細胞株やその他の実験条件で比較することで、単一の読み出しでは見過ごされがちな細胞生物学の複雑な変化を理解することができます。ここでは、PD細胞生物学のモデル化によく使用される細胞型であるヒトiPS細?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
著者らは、提示されたプロトコルの設計につながる貴重な支援と議論をしてくれたKsilinkのすべての同僚に感謝します。
Materials
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse – Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit – Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
References
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson’s disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson’s disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson’s Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson’s Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson’s disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson’s disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson’s disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson’s disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).

