Simülasyon Çalışmalarında Kompleks Lipid Karışımları Kullanılarak Gerçekçi Membran Modellemesi
Summary
Yapı ve bileşimdeki membran lipid çeşitliliği, hücresel süreçlere önemli bir katkıda bulunur ve bir hastalık belirteci olabilir. Moleküler dinamik simülasyonları, membranları ve bunların biyomoleküllerle etkileşimlerini atomistik çözünürlükte incelememizi sağlar. Burada, karmaşık membran sistemlerini oluşturmak, çalıştırmak ve analiz etmek için bir protokol sağlıyoruz.
Abstract
Lipitler, hücre zarlarının yapısal yapı taşlarıdır; Lipid türleri, hücre organelleri ve organizmalar arasında farklılık gösterir. Bu çeşitlilik, membranda, bu arayüzde meydana gelen molekülleri ve süreçleri doğrudan etkileyen farklı mekanik ve yapısal özelliklerle sonuçlanır. Lipid bileşimi dinamiktir ve hücre sinyalleme süreçlerini modüle etmeye hizmet edebilir. Hesaplamalı yaklaşımlar, biyomoleküller arasındaki etkileşimleri tahmin etmek ve deneysel gözlemlenebilirlere moleküler içgörüler sağlamak için giderek daha fazla kullanılmaktadır. Moleküler dinamik (MD), atomların hareketini, üzerlerine etki eden kuvvetlere göre tahmin eden istatistiksel mekaniğe dayalı bir tekniktir. MD simülasyonları, biyomoleküllerin etkileşimini karakterize etmek için kullanılabilir. Burada, tekniği kısaca tanıtıyoruz, lipid çift katmanlarını simüle etmekle ilgilenen yeni başlayanlar için pratik adımları özetliyoruz, yeni başlayanlar için uygun yazılımla protokolü gösteriyoruz ve alternatifleri, zorlukları ve sürecin önemli hususlarını tartışıyoruz. Özellikle, simülasyonda uygun hidrofobik ve mekanik ortamları yakalamak için ilgilenilen bir hücre zarını modellemek için karmaşık lipid karışımlarının kullanılmasının önemini vurguluyoruz. Ayrıca, membran bileşiminin ve özelliklerinin çift katmanların diğer biyomoleküllerle etkileşimlerini modüle ettiği bazı örnekleri de tartışıyoruz.
Introduction
Lipitler, hücreler için sınırlar sağlayan ve hücre içi bölümlendirmeyisağlayan zarların ana bileşenleridir 1,2,3. Lipitler, bir polar baş grubu ve iki hidrofobik yağ asidi kuyruğu ile amfifiliktir; Bunlar, hidrofobik zincirlerin suile temasını en aza indirmek için çift tabaka halinde kendiliğinden birleşir 3,4. Hidrofilik baş gruplarının ve hidrofobik kuyrukların çeşitli kombinasyonları, biyolojik membranlarda gliserofosfolipidler, sfingolipidler ve steroller gibi farklı lipit sınıflarına neden olur (Şekil 1)1,5,6. Gliserofosfolipidler, gliserofosfat, uzun zincirli yağ asitleri ve düşük moleküler ağırlıklı baş gruplarındanoluşan ökaryotik hücre zarlarının birincil yapı taşlarıdır 7. Lipid isimlendirmesi, kafa gruplarındaki farklılıklara dayanır; örnekler arasında fosfatidil-kolin (PC), fosfatidil-etanolamin (PE), fosfatidil-serin (PS), fosfatidil-gliserol (PG), fosfatidil-inositol (PI) veya modifiye edilmemiş fosfatidik asit (PA)5,6. Hidrofobik kuyruklara gelince, omurga yapısı ile birlikte doygunluğun uzunluğu ve derecesi değişir. Olası kombinasyonlar çoktur ve memeli hücrelerinde binlerce lipit türü ile sonuçlanır6. Membran lipid bileşimindeki değişiklikler, hem integral membran proteinlerinin hem de periferik proteinlerinaktivitesini etkileyen farklı mekanik ve yapısal membran özelliklerine yol açar 2,6.
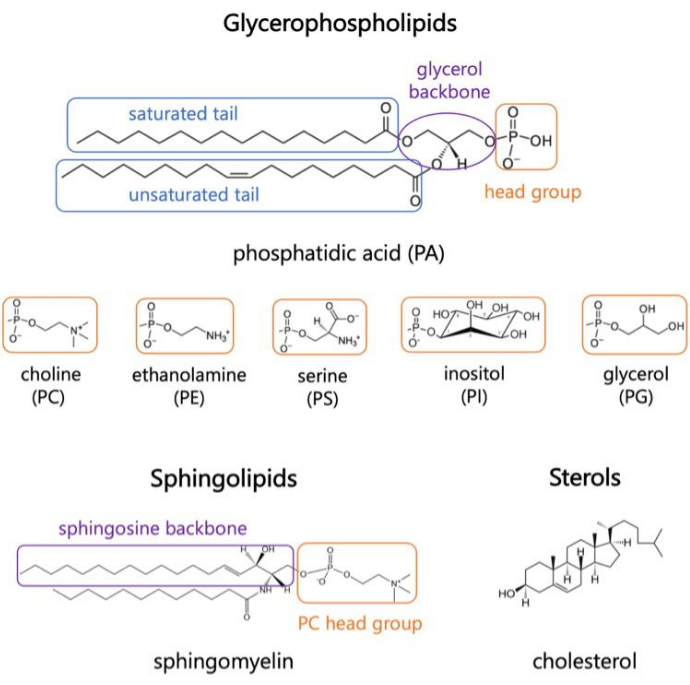
Şekil 1. Temsili lipit yapıları. Yağ asidi kuyrukları mavi kutularda, yaygın lipid baş grupları turuncu renkte ve örnek omurgalar mor renkte gösterilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Lipitler hücresel süreçlerde, sinyal kaskadlarında protein aktivasyonunda ve sağlıklı hücre homeostazında aktif oyunculardır 8,9. Değişen lipid dinamikleri enfeksiyonun sonucudur veyahastalığın patogenezinin belirteçleri olabilir 10,11,12,13,14,15. Hücre için engeller olarak, membran lipidlerinin incelenmesi ve bunların küçük moleküllerin nüfuz etmesindeki rolü, ilaç dağıtım sistemleri ve membran bozulma mekanizmalarıile ilgilidir 16,17. Organeller, dokular ve organizmalar arasındaki kimyasal çeşitlilik ve lipid türlerinin farklı oranları, karmaşık zar dinamiğine yol açar2. Bu nedenle, özellikle bir çalışmanın amacı diğer biyomoleküllerin zar ile etkileşimlerini incelemek olduğunda, lipid çift katmanlarının modelleme çalışmalarında bu özelliklerin korunması önemlidir. Bir modelde dikkate alınacak lipit türleri, ilgilenilen organizmaya ve hücresel bölmeye bağlıdır. Örneğin, PG lipidleri fotosentetik bateria18’de elektron transferi için önemlidir, fosforile inositol lipidleri (PIP’ler) ise plazma zarı (PM) dinamiklerinde ve memeli hücrelerinde sinyal kaskadlarında önemli oyunculardır 19,20. Hücrenin içinde PM, endoplazmik retikulum (ER), Golgi ve mitokondriyal zarlar, işlevlerini etkileyen benzersiz lipid bollukları içerir. Örneğin, ER, lipid biyogenezinin merkezidir ve kolesterolü PM ve Golgi’ye taşır; bol miktarda PC ve PE ile yüksek bir lipit çeşitliliği içerir, ancak membran akışkanlığınıdestekleyen düşük sterol içeriği 21,22,23,24 içerir. Buna karşılık, PM, organizmaya bağlı olarak yüzlerce ve hatta binlerce lipid türünü içerir25, hücre 24’teki diğer zarlara kıyasla karakteristik bir sertlik veren yüksek seviyelerde sfingolipidler ve kolesterol içerir. Yaprakçık asimetrisi, sfingomyelin, PC ve kolesterol açısından zengin bir dış yaprakçık ve basamakları24 sinyallemek için önemli olan PE, PI ve PS açısından zengin bir iç yaprakçık içeren PM gibi membranlar için dikkate alınmalıdır. Son olarak, lipid çeşitliliği, lipid salları olarak bilinen, paketleme ve iç düzende farklılık gösteren mikro alanların oluşumunu da tetikler24,26; Bunlar yanal asimetri sergiler, hücresel sinyalleşmede önemli roller oynadığı varsayılmaktadır26 ve geçici yapıları nedeniyle incelenmesi zordur.
Biyomoleküllerin membranlarla etkileşimlerini araştırmak için floroskopi, spektroskopi gibi deneysel teknikler ve dev unilameller veziküller (GUV’ler) gibi model membran sistemleri kullanılmıştır. Bununla birlikte, ilgili bileşenlerin karmaşık ve dinamik doğasını yalnızca deneysel yöntemlerle yakalamak zordur. Örneğin, proteinlerin transmembran alanlarının görüntülenmesi, bu tür çalışmalarda kullanılan membranların karmaşıklığı ve ilgilenilen işlem sırasında ara veya geçici durumların tanımlanması konusunda sınırlamalar vardır27,28,29. 1980’lerde lipid tek tabakalarının ve çift tabakalarının moleküler simülasyonunun ortaya çıkmasından buyana29, lipit-protein sistemleri ve etkileşimleri artık moleküler düzeyde ölçülebilir. Moleküler dinamik (MD) simülasyonu, moleküller arası kuvvetlerine dayalı olarak parçacıkların hareketini tahmin eden yaygın bir hesaplama tekniğidir. Bir katkı etkileşim potansiyeli, sisteminparçacıkları arasındaki bağlı ve bağlı olmayan etkileşimleri tanımlar 30. Bu etkileşimleri modellemek için kullanılan parametreler kümesi, simülasyon kuvvet alanı (FF) olarak adlandırılır. Bu parametreler, ab initio hesaplamalarından, yarı ampirik ve kuantum mekaniksel hesaplamalardan elde edilir ve diğer yöntemlerin yanı sıra X-ışını ve elektron kırınım deneyleri, NMR, kızılötesi, Raman ve nötron spektroskopisinden elde edilen çoğaltılan verilere optimize edilir31.
MD simülasyonları,32,33,34 çözünürlüğün çeşitli seviyelerindeki sistemleri incelemek için kullanılabilir. Spesifik biyomoleküler etkileşimleri, hidrojen bağlarını ve diğer yüksek çözünürlüklü ayrıntıları karakterize etmeyi amaçlayan sistemler, tüm atom (AA) simülasyonları ile incelenir. Buna karşılık, kaba taneli (CG) simülasyonlar, hesaplama maliyetini azaltmak ve daha büyük ölçekli dinamikleri incelemek için atomları daha büyük fonksiyonel gruplara ayırır33. Bu ikisi arasında, hesaplamayı hızlandırmak için hidrojen atomlarının ilgili ağır atomlarıyla birleştirildiği birleşik atom (UA) simülasyonlarıbulunur 33,35. MD simülasyonları, lipid membranlarının dinamiklerini ve bunların diğer moleküllerle etkileşimlerini araştırmak için güçlü bir araçtır ve membran arayüzünde ilgilenilen süreçler için moleküler düzeyde mekanizmalar sağlamaya hizmet edebilir. Ek olarak, MD simülasyonları, deneysel hedefleri daraltmaya ve mikroskobik etkileşimlere dayalı olarak belirli bir sistemin makromoleküler özelliklerini tahmin etmeye hizmet edebilir.
Kısaca, bir dizi başlangıç koordinatı, hız ve sabit sıcaklık ve basınç gibi bir dizi koşul verildiğinde, her parçacığın konumları ve hızları, etkileşim potansiyelinin ve Newton’un Hareket Yasasının sayısal entegrasyonu yoluyla hesaplanır. Bu, yinelemeli olarak tekrarlanır, böylece bir simülasyon yörüngesi30 oluşturulur. Bu hesaplamalar bir MD motoru ile gerçekleştirilir; Birkaç açık kaynaklı paket arasında, GROMACS36 en yaygın kullanılan motorlardan biridir ve burada açıkladığımız motordur. Ayrıca, simüle edilecek sistemlerin ilk koordinatlarının analizi ve oluşturulması için araçlar içerir37. Diğer MD motorları arasında NAMD38; CHARMM39 ve AMBER40, kullanıcının belirli bir sistemin hesaplama performansına dayalı olarak kendi takdirine bağlı olarak seçebileceği. Simülasyon sırasında yörüngeleri görselleştirmenin yanı sıra sonuçların analizi ve yorumlanması için de kritik öneme sahiptir. Çeşitli araçlar mevcuttur; burada, geniş çizim ve renklendirme yöntemleriyle üç boyutlu (3-D) görselleştirme, hacimsel veri görselleştirme, MD simülasyon sistemlerinin yörüngelerini oluşturma, hazırlama ve analiz etme ve bellek mevcutsa sistem boyutunda herhangi bir sınırlama olmaksızın yörünge filmi yapımı dahil olmak üzere çok çeşitli özellikler sunan görsel moleküler dinamikleri (VMD) tartışıyoruz41,42,43.
Sistem bileşenleri arasında tahmin edilen dinamiklerin doğruluğu, yörüngenin yayılması için seçilen FF’den doğrudan etkilenir. Ampirik FF parametrelendirme çabaları az sayıda araştırma grubu tarafından sürdürülmektedir. MD için en köklü ve yaygın FF CHARMM39, AMBER 40, Martini44, OPLS 45 ve SIRAH 46’dır. Tüm atom katkı maddesi CHARMM36 (C36) kuvvet alanı47, deneysel yapısal verileri doğru bir şekilde yeniden ürettiği için membran sistemlerinin AA MD’si için yaygın olarak kullanılmaktadır. Başlangıçta CHARMM topluluğu tarafından geliştirilmiştir ve GROMACS ve NAMD gibi birden fazla MD motoruyla uyumludur. Yaygın FF’lerdeki gelişmelere rağmen, belirli çalışma sistemlerine olan ilgiler tarafından yönlendirilen, deneysel gözlemlenebilirleri yakından yeniden üreten tahminlere izin vermek için parametre setlerini geliştirmek için sürekli bir çaba vardır48,49.
Lipid membranları simüle ederken karşılaşılan bir zorluk, simülasyon yörüngesinin uzunluğunu belirlemektir. Bu, büyük ölçüde analiz edilecek metriklere ve karakterize etmeyi amaçlayan sürece bağlıdır. Tipik olarak, karmaşık lipid karışımlarının dengeye ulaşması için daha uzun zaman gerekir, çünkü daha fazla türün zar düzleminde yayılması ve kararlı bir yanal organizasyona ulaşması için yeterli zamana sahip olması gerekir. İlgilenilen özellik bir platoya ulaştığında ve sabit bir değer etrafında dalgalandığında bir simülasyonun dengede olduğu söylenir. İlgilenilen özellikler ve etkileşimler üzerinde uygun istatistiksel analiz yapmak için en az 100-200 ns dengeli yörünge elde etmek yaygın bir uygulamadır. Lipid karışımının karmaşıklığına ve araştırma sorusuna bağlı olarak 200-500 ns arasında yalnızca membran simülasyonları çalıştırmak yaygındır. Protein-lipid etkileşimleri tipik olarak 500-2000 ns arasında daha uzun simülasyon süreleri gerektirir. Membran sistemleriyle örneklemeyi ve gözlemlenebilir dinamikleri hızlandırmak için bazı yaklaşımlar şunlardır: (i) örneklemeyi hızlandırmak için membrandaki lipitlerin uç karbonlarını organik çözücü ile değiştiren oldukça hareketli membran mimetik (HMMM) modeli50; ve (ii) daha büyük bir simülasyon zaman adımı51’in kullanılmasına izin vermek için bir sistem içindeki ağır atomların kütlelerinin bir kısmını hidrojen atomlarınınkilerle birleştiren hidrojen kütle yeniden bölümleme (HMR).
Aşağıdaki protokol, AA MD kullanarak gerçekçi membran modelleri oluşturmak, çalıştırmak ve analiz etmek için yeni başlayanlar için uygun bir yaklaşımı ele almaktadır. MD simülasyonlarının doğası göz önüne alındığında, tekrarlanabilirliği ve sonuçların uygun istatistiksel analizini hesaba katmak için birden fazla yörünge çalıştırılmalıdır. İlgilenilen sistem başına en az üç çoğaltma çalıştırmak geçerli bir uygulamadır. İlgilenilen organizma ve süreç için lipit türleri seçildikten sonra, yalnızca zardan oluşan bir sistemin simülasyon yörüngesini oluşturmak, çalıştırmak ve analiz etmek için temel adımlar Şekil 2’de özetlenir ve özetlenir.
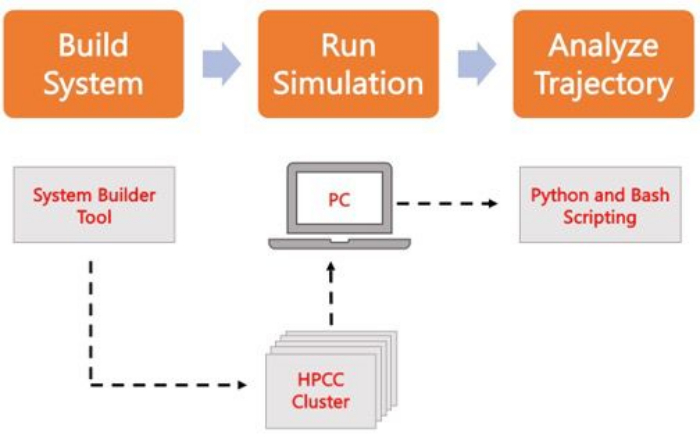
Şekil 2. MD simülasyonlarını çalıştırmak için şematik. Turuncu kutular, protokolde açıklanan üç ana adıma karşılık gelir. Aşağıda simülasyon sürecinin iş akışı yer almaktadır. Sistem kurulumu sırasında, çözülmüş bir membran sisteminin başlangıç koordinatlarını içeren sistem, CHARMM-GUI Membrane Builder gibi bir sistem giriş üreteci ile oluşturulur. Giriş dosyalarını yüksek performanslı bir bilgi işlem kümesine aktardıktan sonra, simülasyon yörüngesi GROMACS gibi bir MD motoru kullanılarak yayılır. Yörünge analizi, görselleştirme ile birlikte bilgisayar kümesinde veya yerel bir çalışma istasyonunda yapılabilir. Analiz daha sonra GROMACS ve VMD gibi yerleşik analiz koduna sahip paketler kullanılarak veya Bash komut dosyaları veya çeşitli Python kitaplıkları kullanılarak gerçekleştirilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Deneysel teknikler, kriyo-elektron mikroskobu (kriyo-EM)58, floresan teknikleri ve atomik kuvvet mikroskobu (AFM)59 kullanarak biyomolekülleri yüksek çözünürlükte görselleştirebilir. Bununla birlikte, biyolojik yolların, hastalık patogenezinin ve atomik veya amino asit düzeyinde terapötik dağıtımın altında yatan moleküler etkileşimlerin etkileşimini ve dinamiklerini yakalamak zordur. Burada, MD simülasyonlarının lipid membranları inceleme yetenekler…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Yazarlar, bu makalenin yazımı sırasındaki simülasyon yörüngeleri ve tartışmaları için Jinhui Li ve Ricardo X. Ramirez’e teşekkür eder. O.C., Buffalo Üniversitesi Başkanlık Bursu ve Ulusal Sağlık Enstitüsü’nün Öğrenci Gelişimini En Üst Düzeye Çıkarma Girişimi tarafından desteklenmiştir Eğitim Hibesi 1T32GM144920-01, Margarita L. Dubocovich’e (PI) verildi.
Materials
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
References
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M., Edward, H. .. . E. g. e. l. m. a. n. . Comprehensive Biophysics. , 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis, , Martin Raff, , Keith Roberts, , Peter Walter, . Molecular Biology of the Cell. , (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, &. #. 2. 2. 0. ;., Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Douglas G, H. a. y. e. s., Daniel, K. Y., Solaiman, D., Richard, . Biobased Surfactants (Second Edition) eds. , 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41 (2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264 (2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. Biochimie. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5 (2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897 (2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167 (2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441 (2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S., Scott Shell, M. . Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. , 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational ‘microscopy’ of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901 (2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B., Otto Geiger, . Biogenesis of Fatty Acids, Lipids and Membranes. , 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068 (2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130 (2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. . Using VMD. , (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. . CCR Facility Description. , (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841 (2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38 (2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- . . Hepatitis C. , (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957 (2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. Biochimie. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. o. b. e. r. t. . T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621 (2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).

