Analyserer Protein Dynamics hjælp af brint Exchange massespektrometri
Summary
Protein kropsbygning og dynamik er nøglen til at forstå forholdet mellem protein struktur og funktion. Hydrogen udveksling kombineret med høj opløsning massespektrometri er en alsidig metode til at studere de konformationelle dynamik proteiner samt karakterisering af protein-ligand-og protein-protein-interaktioner, herunder kontaktflader og allosteriske effekter.
Abstract
Alle cellulære processer, afhænger af funktionaliteten af proteiner. Selvom funktionaliteten af et givet protein er den direkte konsekvens af sin unikke aminosyresekvens, er den kun realiseres ved foldningen af polypeptidkæden i en enkelt veldefineret tredimensionel arrangement eller mere almindeligt i et ensemble af interconverting konformationer. Undersøgelse af forbindelsen mellem protein kropsbygning og dets funktion er derfor afgørende for en fuldstændig forståelse af, hvordan proteiner er i stand til at opfylde deres store udvalg af opgaver. En mulighed for at studere konformationsændringer et protein gennemgår mens forløber gennem dets funktionelle cyklus er hydrogen-1 H / 2 H-udveksling i kombination med høj opløsning massespektrometri (HX-MS). HX-MS er en alsidig og robust metode, der tilføjer en ny dimension til strukturel information fås ved f.eks crystallography. Det bruges til at studere proteinfoldning og udfoldning binding af små molecule ligander, protein-protein interaktioner, konformationsændringer tilknytning til enzymkatalyse og allostery. Desuden er HX-MS anvendes ofte, når mængden af protein er meget begrænset eller krystallisation af proteinet ikke er mulig. Her giver vi en generel protokol for at studere protein dynamik med HX-MS og beskriver som et eksempel på, hvordan at afsløre samspillet grænseflade to proteiner i en kompleks.
Introduction
Antallet af krystalstrukturer af proteiner og protein komplekser steget kraftigt i de seneste år. De præsenterer uvurderlige snapshots af den strukturelle organisering af disse proteiner og skabe et grundlag for struktur-funktion analyse. Men dynamikken i proteiner og de konformationsændringer, som er afgørende for deres funktioner, er sjældent afsløret ved røntgenkrystallografi. Cryo-elektronmikroskopi på den anden side er i stand til at fange protein og protein-komplekser i forskellige konformationer, men generelt kan ikke løse konformationelle ændringer ned til sekundær struktur niveau 1. Konformationelle dynamik af proteiner i opløsning ved atomare detaljer kan kun løses ved NMR, men denne metode er stadig begrænset til proteiner af relativt små størrelser (generelt ≤ 30 kDa) og har brug for høje koncentrationer af proteiner (≥ 100 mM), som hæmmer eksperimenter med oligomeriserende eller sammenlægning tilbøjelige proteiner 2. En metode, derer i stand til at bygge bro mellem høj opløsning røntgenkrystallografi og cryo-elektronmikroskopi, og som ikke er begrænset af protein størrelse eller koncentration er amid hydrogen-1 H / 2 H-exchange (HX) i kombination med massespektrometri (MS). I de senere år har denne metode udviklet til et værdifuldt analytisk værktøj til analyse af protein-dynamik, proteinfoldning, proteinstabilitet og konformationsændringer 3-5. Den molekylære basis for denne metode er den labile karakter af backbone amide hydrogener i proteiner, som vil veksle med deuteriumatomer når proteinet er anbragt i en D2O løsning. Den efterfølgende stigning i protein masse over tid måles med høj opløsning MS.
I korte ustrukturerede peptider HX afhænger kun af temperatur katalysator fusion (OH -, H 3 O + dvs. pH, se figur 3) og kæder af tilstødende rester aminosyre side grundet induktiv, katalytic og steriske effekter. Disse virkninger på den iboende kemiske vekselkurs k lm er elegant kvantificeret ved Bai et al. 6 og et program er til rådighed (udlånt Z. Zhang), som beregner k lm for hver aminosyre i et polypeptid, afhængig af pH og temperatur. Ved neutral pH og omgivelsestemperaturer k ch er i størrelsesordenen af 10 1 -10 3 sek -1. I foldede proteiner kan HX være 2-9 størrelsesordener langsommere hovedsageligt på grund af hydrogenbinding i den sekundære struktur og i mindre grad på grund af begrænset adgang hydratiserede OH – ioner til det indre af en tæt foldet protein. HX i native proteiner implicerer derfor delvis eller global udfoldning, kemisk udveksling og refoldning til den oprindelige tilstand i henhold til ligning (1), og de observerede valutakurser k obs afhænger åbningen rate k op, balancedagens kurs k cl og den iboende kemisk udveksling rate k ch ifølge ligning (2).
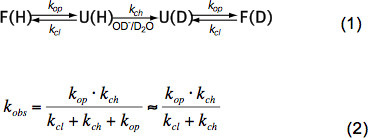
Under native state betingelser k op er meget mindre end k lm og kan ignoreres i nævneren. Der er to ekstreme valutakurser regimer kaldet EX1 og EX2. Hvis k cl er meget mindre end k lm (EX1) den observerede hastighed er praktisk taget lig med åbningen sats og HX giver umiddelbar iagttagelse af udfoldning af et konstruktionselement. En sådan udveksling regime, hvor alle amid protoner udveksling på en gang ved åbning af den strukturelle element, er umiddelbart kan opgøres i MS med en bimodal fordeling af isotoptoppe 7. Hvis k cl er meget større end k lm (EX2) den observerede rente er proportional med k ch hvorved proportionalitetskonstanten er lig med folde-udfoldning balancer konstant K u = k op </sub> / K cl. Under disse betingelser, er mange åbning og lukning begivenheder er nødvendige, før alle amid protoner bytte for deuteroner, hvilket fører til en gradvis stigning i den gennemsnitlige masse, mens den isotopiske fordeling forbliver nogenlunde det samme. Den EX2 regime tillader bestemmelse af fri energi udfolde ΔG u og dermed stabiliteten på et strukturelt element. Under oprindelige tilstand betingelse EX2 regime er mest almindelig. Forøgelse af pH og tilsætning af chaotrope midler kan flytte udvekslingen mekanisme til at EX1. Derfor kan HX-MS bruges til at udforske termodynamisk samt kinetiske parametre for proteinfoldning og konformationelle ændringer.
Som nævnt ovenfor HX er uløseligt pH og temperatur afhængig og udveksling halveringstid af en helt opløsningsmiddel udsat proton rygraden amidgruppen er mellem 5-400 msek ved fysiologisk pH (pH 7,6) og 30 ° C, men 10 min til> 15 timer med et gennemsnit på> 2 timer ved pH 2,9 og 0 °C (bortset fra proton første rygraden amidbinding af et polypeptid, som udveksler med en halveringstid på ca. 1-2 min.) Under sådanne langsom udveksle forhold er det muligt at fordøje prøven ved hjælp af proteaser (f.eks pepsin), som er aktiv under disse betingelser, med ud at miste alle de oplysninger, der findes i de inkorporerede deuteroner. Siden indførelsen af peptisk fordøjelse under langsomme udveksle forhold, kan ikke kun de samlede HX kinetik af proteiner i fuld længde analyseres men HX kan lokaliseres til bestemte regioner 8,9. Rumlig opløsning øjeblikket er begrænset til størrelsen af de peptiske fragmenter, hvilket er generelt mellem 10-30 rester. Overlappende fragmenter skabt på grund af den ikke-specifikke karakter af spaltning med pepsin, kan imidlertid føre til en stigning i rumlig opløsning. Desuden blev flere andre proteaser fundet at være aktive under quench betingelser dog langt mindre effektive end pepsin 10. Yderligere increaSE for rumlig opløsning kan nås ved fragmentering af peptider i gasfasen ved metoder, der bevarede deutereringen mønster såsom indfangning af elektroner dissociation (ECD), elektron transfer dissociation (EBD) og infrarød multiphoton dissociation (IRMPD) 11-13. Disse teknikker forhindre tab af rumlig opløsning på grund af intramolekylær proton migration ("scrambling"), som er observeret ved kollision-induceret dissociation (CID) den mest almindeligt anvendte fragmentering teknik. Men disse metoder kræver optimering for hvert enkelt peptid og er således stadig ganske udfordrende.
HX-MS er blevet anvendt til analyse af protein-ligand-og protein-protein-interaktioner, herunder virale capsid samlingen 14-17. Proteinudfoldning og genfoldning såvel som temperatur inducerede konformationelle ændringer blev undersøgt 7,18,19. Phosphorylering og enkelt aminosyremutation-relaterede konformationsændringer 16,20 og nucleotide-inducerede ændringer blev analyseret 21,22. Derfor er denne metode synes ideelt egnet til at analysere montage og dynamik molekylære maskiner. En attraktiv kandidat, hvis mekanisme er af stor almen interesse, er Hsp90 anstandsdame kompleks.
Protocol
Representative Results
Discussion
Binding af et samspil partner til et protein uundgåeligt forårsager ændringer i tilgængelighed opløsningsmiddel på bindingssted. Derudover er der mange proteiner undergår dynamiske konformationsændringer ved binding, som påvirker andre regioner end den faktiske bindende interface. HX-MS er en robust metode til at overvåge disse ændringer og er endda i stand til at afsløre konformationsændringer i proteiner på tidsskalaer der andre metoder ikke kan dække.
For med held at udfør…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Vi takker M. Boysen for kommentarer til manuskriptet. Dette projekt blev finansieret af Deutsche Forschungsgemeinschaft (SFB638 og MA 1278/4-1 til MPM, og Cluster of Excellence: CellNetworks EXC 81/1). MPM er investigator i Cluster of Excellence: CellNetworks.
Materials
| Reagent/Equipment | Company | Catalogue number | Comments (optional) |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
Riferimenti
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5′-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O’Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).

