ДНК-аффинно очищенного Чип (DAP-чип) Метод определения целей гена для бактериальных двухкомпонентный регуляторных систем
Summary
Это видео статье описывается микрочипов на основе метода в пробирке для определения целевых генов и сайты связывания для двухкомпонентных регуляторов срабатывания системы.
Abstract
В методах естественных условиях, таких как чип-чип являются хорошо зарекомендовавшие себя методы, используемые для определения глобальных целей гена для транскрипционных факторов. Тем не менее, они имеют ограниченное применение в изучении бактериальных двухкомпонентных систем регулирования с неохарактеризованных условиях активации. Такие системы регулировать транскрипцию только при активации в присутствии уникальных сигналов. Так как эти сигналы часто неизвестно, пробирке микрочипов на основе метода в описано в этой статье видео может быть использован для определения генов цели и сайты связывания для регуляторов реагирования. Этот метод ДНК-аффинно очищенного-чип может быть использован для любого очищенного регулятора в любом организме с виртуализированного генома. Протокол включает позволяя очищают отмеченных белка для связывания с стриженой геномной ДНК, а затем очистки сродство ДНК связанной с белком, а затем флуоресцентного мечения ДНК и гибридизации в массив пользовательских плитки. Предыдущие шаги, которые могут быть использованы для оптимизации анализа для спецификациирегуляторы грн, также описаны. Пики, полученные в результате анализа данных массива используются для прогнозирования обязательные мотивы сайта, которые затем экспериментально проверенные. Предсказания мотив может быть дополнительно использован для определения генов целей ортологичных регуляторов реагирования в близкородственных видов. Мы продемонстрировать применимость этого метода путем определения генов цели и связывающие мотивы сайта и таким образом предсказывать функцию для sigma54 зависит от регулятора ответ DVU3023 в экологической бактерии Desulfovibrio обыкновенная Hildenborough.
Introduction
Способность бактерий, чтобы выжить и процветать в решающей степени зависит от того, как хорошо они способны воспринимать и отвечать возмущений в их среде, а это, в свою очередь, зависит от их систем передачи сигнала. Количество сигнальных систем бактерия кодирует вектор был назван его "микробная IQ" и может быть признаком как изменчивости его среды и ее способность чувствовать несколько сигналов и тонкой настройки его отклика 1. Два компонента системы передачи сигнала (TCS) являются наиболее распространенными системами сигнализации, используемые бактериями, и они состоят из гистидинкиназа (HK), что воспринимает внешний сигнал и передает через фосфорилирования к регулятору отклика эффектор (RR) 2. ОР могут иметь различные выходных доменов и, следовательно, различные режимы эффекторные, но наиболее распространенным ответом является регуляция транскрипции с помощью ДНК-связывающего домена 1. Сигналы зондирования и соответствующие функции протокат Большинство ТТС остаются неизвестными.
Хотя естественных условиях методы в таких, как чип-чип обычно используются для определения геномных сайтов связывания транскрипционных факторов 3, они могут быть использованы только для бактериальных RR, двухкомпонентная система, если условия активирующие или сигналы известны. Часто экологические сигналы, которые активируют TCS труднее определить, чем их генов мишеней. Пробирке микрочипов в основе анализа, описанного здесь, могут использоваться для эффективного и быстрого определения генов цели и прогнозировать функции ТТС. Этот анализ имеет преимущество в том, что РП может быть фосфорилирован и, таким образом активированного в пробирке с использованием малых молекул доноров, таких как ацетил фосфата 4.
В этом методе, названный DAP-чип для ДНК-аффинной очистке чипа (рис. 1), ген RR интересующий клонированный с His-меткой в Е. палочка, и отмеченных белок очищен разрешается биго по стриженой геномной ДНК. ДНК белком затем обогащается аффинной очистки, обогащенный и вход ДНК усиливаются, флуоресцентно меченных, объединяли и гибридизации с массивом плитки, которая на заказ для организма интереса (рис. 1). Microarray эксперименты подлежат артефактов и, следовательно, дополнительные шаги используются для оптимизации анализа. Одним из таких шагов является попытка определить одну цель для РР изучается с помощью электрофореза сдвига мобильность анализов (EMSA) (см. рабочий процесс на рисунке 2). Затем, после связывания с геномной ДНК и DAP шагов, ДНК белком и вход рассматриваются кПЦР чтобы увидеть, если положительный целевой обогащается во фракции белком относительно входного фракции, подтвердив тем самым оптимальное обязательные условия для RR (рис. 2). После массива гибридизации, данные анализируются, чтобы найти пиков высокой интенсивности сигнала, указывающие геномной локусов, где белок чобъявление связаны. Функции могут быть предсказаны на основе РР целевых генов, полученных. Целевые геномные локусы используются для прогнозирования обязательные мотивы сайта, которые затем экспериментально проверены с использованием EMSAs (рис. 2). Функциональные прогнозы и генные мишени для РР может быть продлен до близкородственных видов, которые кодируют ортологичные RRs путем сканирования эти геномы для аналогичных обязательных мотивов (рис. 2). Метод доступа DAP-чип может обеспечить большой объем информации для TCS, где ранее не было ни одного. Способ также может быть использован для любого транскрипционного регулятора, если белок может быть очищен и условиях связывания ДНК может быть определена, и для любого интересующего организма с последовательностью генома доступны.
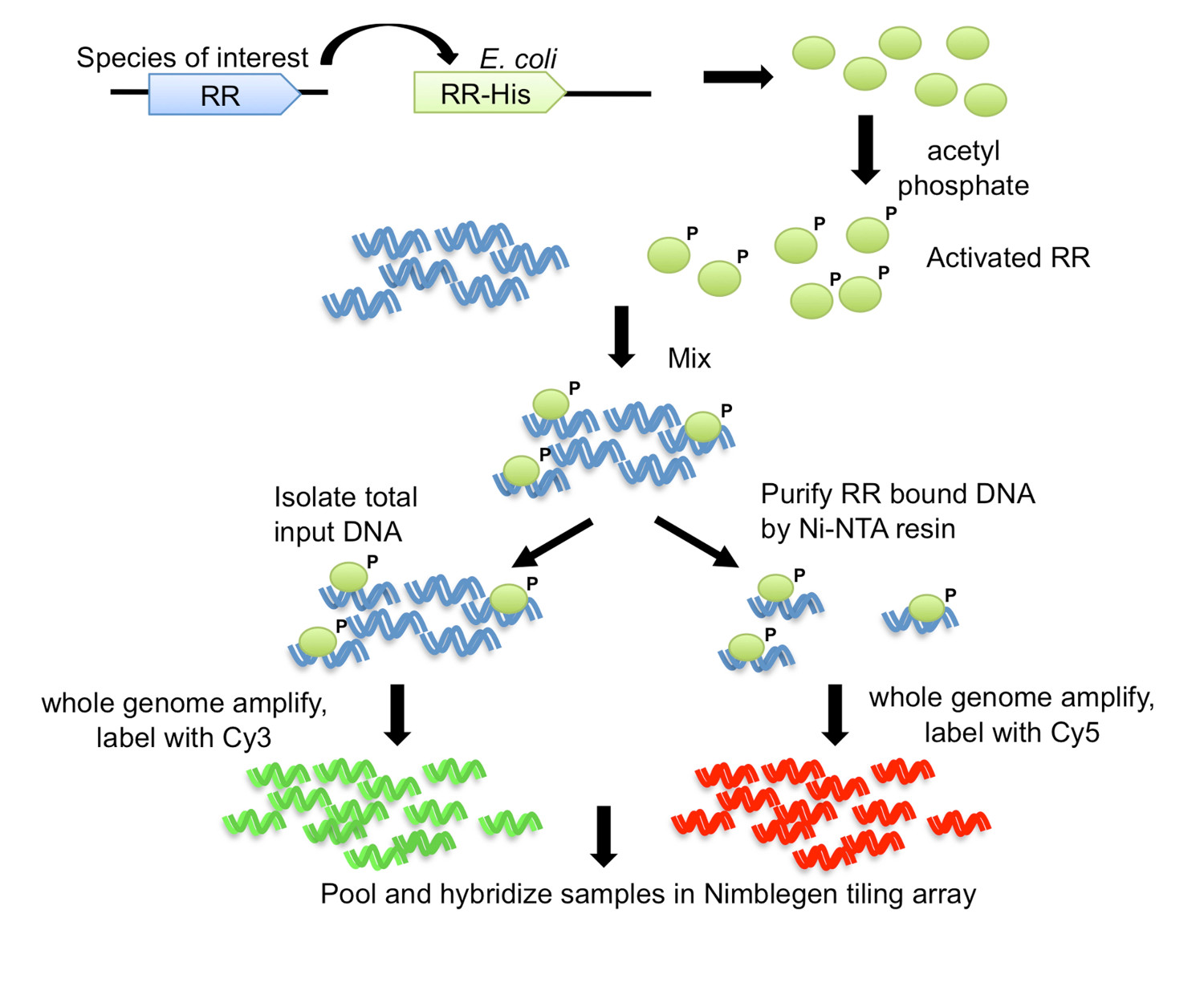
Рисунок 1. ДНК-аффинно очищенного-чип (DAP-чип) Стратегия 7. Ген RR от интересующего организма, клонируют с карбокси-концевой His-меткой в Е. Штамм выражение. Очищенный белок His-меченого активируется путем фосфорилирования с ацетил фосфата и смешивают с обрезной геномной ДНК. Аликвоту реакции связывания сохраняется в качестве входного ДНК, а остальные подвергают аффинной очистки с использованием Ni-NTA смолы. Входной и RR-связаны ДНК амплифицируют весь геном, и помечены с Cy3 и Cy5, соответственно. Меченый ДНК объединяли и гибридизовали в массив плитки, которую затем анализируют для определения генов мишеней. Рисунок изменен и перепечатана с помощью творческую лицензию общин от 7.
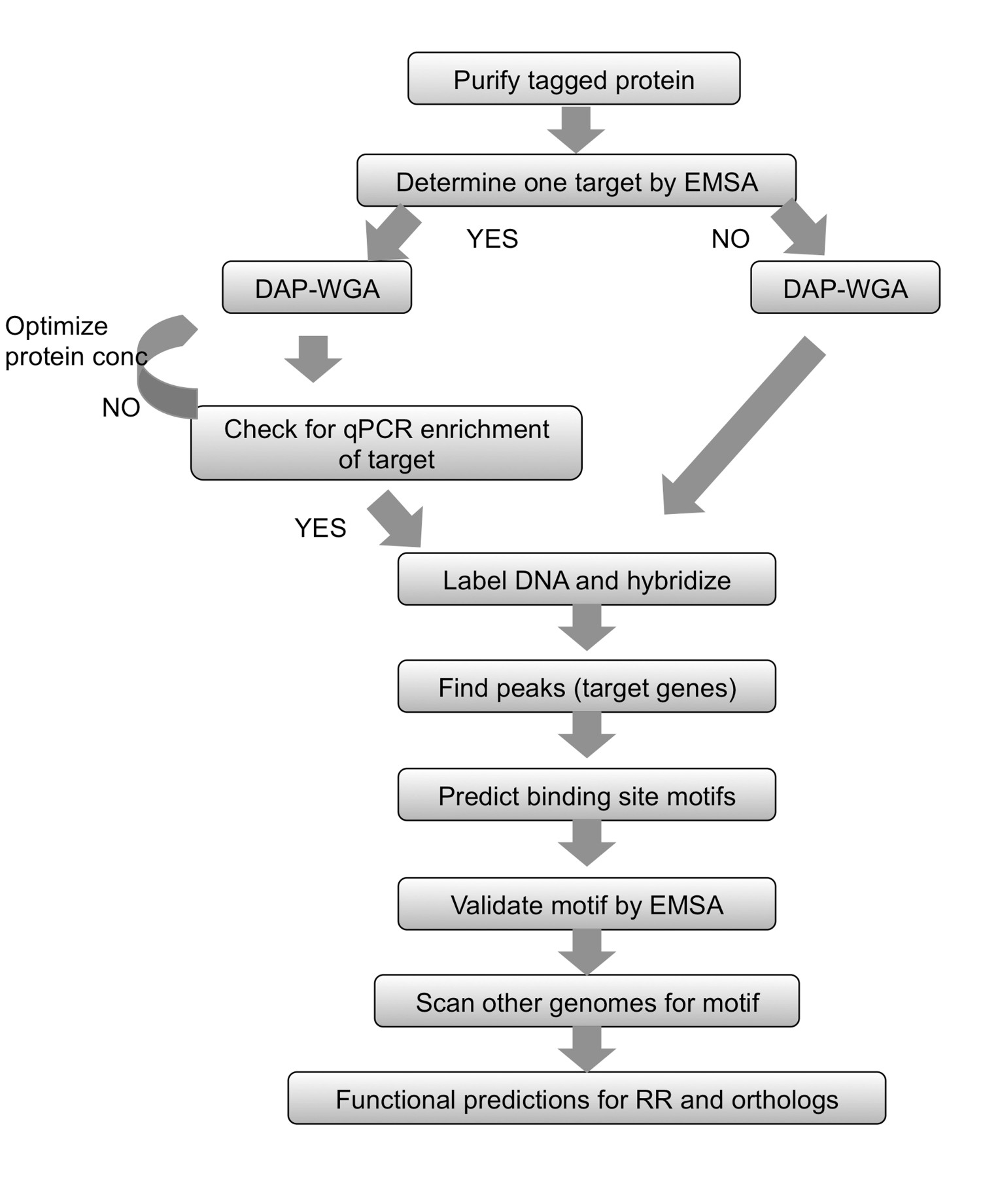
Рисунок 2. Резюме процесса. Для любого очищенного меченого Proteiп, начать с определения цели использования Emsa. Разрешить белок связывать геномной ДНК, а затем ДНК-сродства-Purify (DAP) и весь геном усиления (WGA) обогатили и вход ДНК. Если ген-мишень, как известно, использовать КПЦР чтобы гарантировать, что известно целевой обогащен фракции белком. Если цель не могла быть определена, перейдем непосредственно к маркировке ДНК и массива гибридизации. Если обогащение кПЦР не могло быть обнаружено, а затем повторите белок-гДНК связывания и DAP-WGA шаги, используя различные количества белка. Используйте анализ массива найти пики и сопоставить их с генов-мишеней. Используйте вверх по течению регионы генов-мишеней для прогнозирования обязательные мотивы сайта. Подтвердить мотивы экспериментально с помощью EMSAs. Используйте мотив для сканирования геномы родственных видов, кодирующих Ортологи РР в стадии изучения, и предсказать гены целевых в тех видах, а также. На основе целей гена, полученных, физиологическая функция РР и его ортологов может быть предсказано. Рисунок изменен и перепечатана с помощью творческого грommons лицензировать с 7.
Protocol
Representative Results
Discussion
Метод доступа DAP-чип описано здесь был успешно использован для определения генов цели в течение нескольких записей ресурсов в Desulfovibrio обыкновенная Hildenborough 7, один из которых показан здесь как представитель результате. Для RR DVU3023, выбирая цель гена-кандидата был прямым. DVU3025 рас…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Мы благодарим Эми Чен за помощь в подготовке к съемках клипа и для демонстрации техники. Эта работа проводится ENIGMA: Экосистемы и сети Интегрированный с генами и молекулярных ансамблей (http://enigma.lbl.gov), Программа научно Зона фокусировки на Национальной лаборатории Лоренса Беркли, была поддержана Управлением науки, Управления биологического и экологических исследований, Департамента энергетики США по договору № DE-AC02-05CH11231.
Materials
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
Riferimenti
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).

