DNA-afinidade-purificada Método Chip (DAP-chip) para determinar genes alvos para bacteriana Dois Sistemas Regulatórios componentes
Summary
Este artigo vídeo descreve um método de microarray baseado in vitro para determinar os genes alvos e sítios de ligação para dois reguladores de resposta do sistema de componentes.
Abstract
Os métodos in vivo, tais como Chip-chip são técnicas bem estabelecidas usados para determinar genes alvos globais para fatores de transcrição. No entanto, eles são de uso limitado na exploração de dois sistemas de regulação componente bacteriano com condições de ativação descaracterizados. Tais sistemas de regular a transcrição apenas quando activada, na presença de sinais únicos. Uma vez que estes sinais são muitas vezes desconhecidos, o método de microarray com base in vitro descritos neste artigo de vídeo podem ser utilizados para determinar alvos de genes e sítios de ligação para reguladores da resposta. Este método de ADN purificado por afinidade-chip pode ser utilizado para qualquer regulador purificada em qualquer organismo com um genoma sequenciado. O protocolo envolve permitindo que a proteína purificada marcado para se ligar ao DNA genómico cortado e, em seguida, a purificação por afinidade de ADN ligado à proteína, seguido de marcação fluorescente do ADN e hibridação com uma matriz de ladrilhos costume. Precedendo passos que podem ser utilizados para optimizar o ensaio para a especificidadereguladores c também são descritos. Os picos gerados pela análise dos dados de matriz são utilizados para prever motivos sítios de ligação, que são, em seguida, validada experimentalmente. As previsões motivo pode ser ainda utilizado para determinar genes alvos dos reguladores de resposta ortólogos em espécies estreitamente relacionadas. Nós demonstramos a aplicabilidade deste método através da determinação dos genes alvos e motivos do site vinculativo e prevendo, assim, a função de um regulador de resposta DVU3023-dependente sigma54 na bactéria Desulfovibrio vulgaris Hildenborough ambiental.
Introduction
A capacidade das bactérias para sobreviver e prosperar é criticamente dependente da forma como eles são capazes de perceber e responder a perturbações em seus ambientes, e este por sua vez é dependente de seus sistemas de transdução de sinal. O número de sistemas de sinalização uma bactéria codifica tem sido chamado o seu "QI microbiana" e pode ser uma indicação de ambos variabilidade do seu ambiente e da sua capacidade de sentir múltiplos sinais e afinar a sua resposta 1. Dois sistemas de componentes de transdução de sinal (TCS) são os sistemas de sinalização mais prevalentes utilizados por bactérias, e que consistem de uma histidina-quinase (HK), que detecta o sinal externo e transmite através da fosforilação de um regulador de resposta efectora (RR) 2. RRS pode ter uma variedade de domínios de saída e, portanto, diferentes modos efetoras, mas a resposta mais comum é a regulação da transcrição por meio de um DNA domínio 1 vinculativo. Os sinais detectados e as funções correspondentes dos vast maioria dos TCSs permanecem desconhecidos.
Embora métodos in vivo, tais como Chip-chip são habitualmente usadas para a determinação de locais de ligação de factores de transcrição do genoma 3, que apenas pode ser utilizado para RRS sistema de dois componente bacteriano, se são conhecidas as condições de activação ou sinalização. Muitas vezes, os sinais ambientais que ativam a TCS são mais difíceis de determinar que os seus genes alvos. O microarray ensaio in vitro com base aqui descrita pode ser utilizada para determinar rápida e eficazmente os genes alvos e prever as funções de TCS. Este ensaio tira partido do facto de RRS pode ser fosforilada e assim activado in vitro utilizando pequenos doadores molécula como fosfato acetil 4.
Neste método, chamado DAP-chip para ADN de afinidade-purificada de chip (Figura 1), o gene da RR de interesse é clonado com uma cauda de His no E. coli, e uma proteína marcada subsequentemente purificado é permitido biª a cortado DNA genômico. O DNA ligado à proteína é então enriquecida por afinidade-purificação, o ADN enriquecido e entrada são amplificados, marcado por fluorescência, reunidas e hibridado com um conjunto azulejos que é feito para o organismo de interesse (Figura 1). Microarray experiências estão sujeitos a artefactos e, por conseguinte, são empregues passos adicionais para optimizar o ensaio. Um tal passo é a tentativa de determinar um destino para o RR em estudo utilizando ensaios de desvio de mobilidade electroforética (EMSA) (ver fluxo de trabalho na Figura 2). Em seguida, após a ligação ao ADN genómico e as etapas de DAP, o DNA ligado à proteína e de entrada são examinados por qPCR para ver se o alvo positivo é enriquecido na fracção ligada à proteína em relação à fracção de entrada, confirmando, assim, as condições óptimas de ligação para o RR (Figura 2). Após a matriz de hibridação, os dados são analisados para encontrar os picos de intensidade de sinal mais elevado, indicando locus genómico onde a proteína hanúncio vinculada. As funções podem ser previstos para o RR com base nos genes alvos obtidos. Os loci alvo genómicas são usadas para prever motivos sítios de ligação, que são, em seguida, experimentalmente validados usando EMSAs (Figura 2). As previsões funcionais e genes alvos para a RR pode então ser estendido para as espécies que codificam RRS ortólogos por inventariar os genomas de motivos de ligação semelhantes (Figura 2) intimamente relacionados. O método DAP-chip pode fornecer uma riqueza de informações para a TCS onde antes não havia nenhuma. O método também pode ser utilizado para qualquer regulador de transcrição se a proteína pode ser purificada e as condições de ligação de ADN pode ser determinada, e para todo o organismo de interesse com uma sequência de genoma disponível.
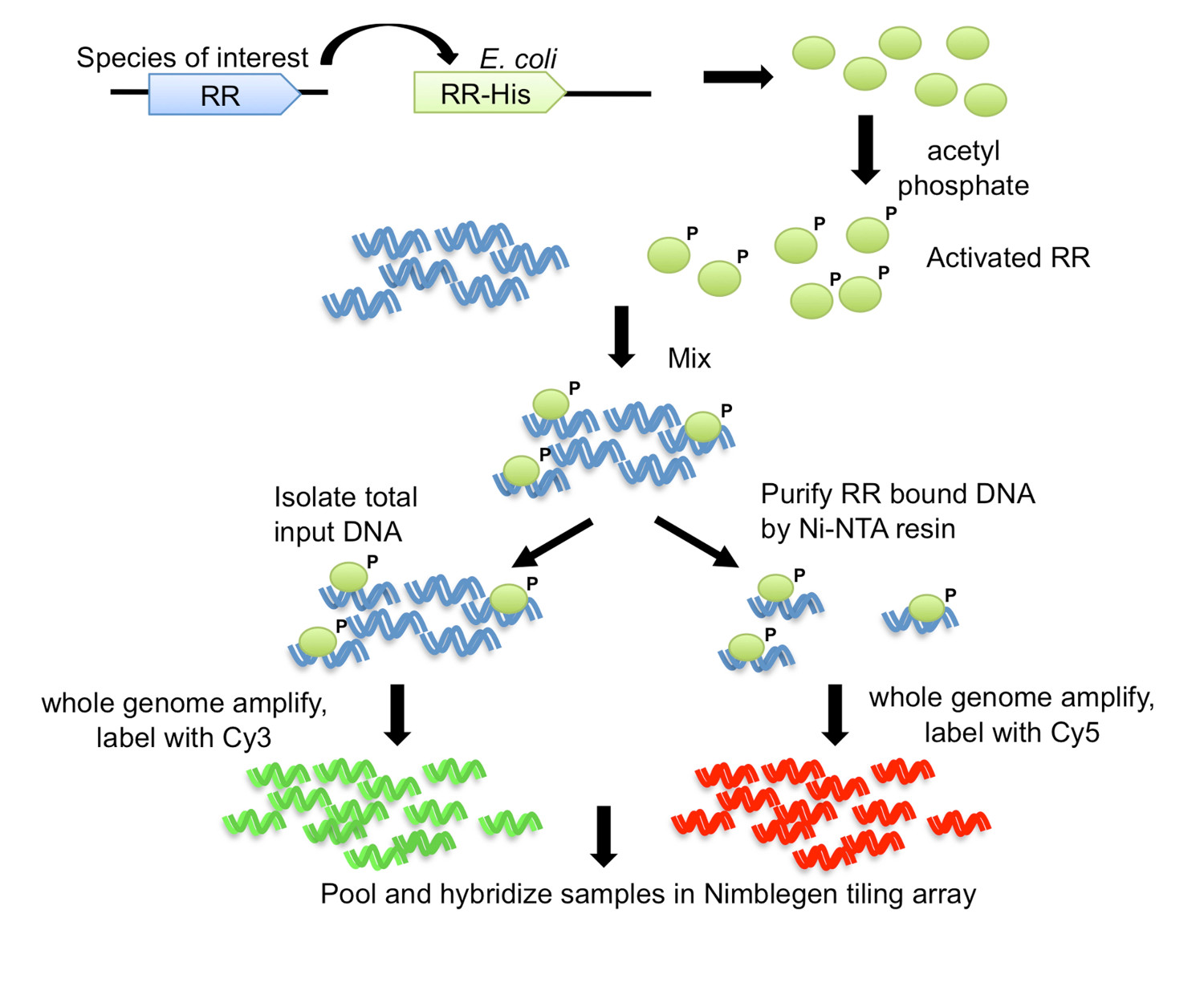
Figura 1. O chip de ADN purificado por afinidade (DAP-estratégia chip) 7. O gene RR a partir do organismo de interesse é clonado com um carboxi-terminal His-tag numa E. tensão expressão coli. Purificado proteína marcada com His é activada por fosforilação com fosfato de acetilo, e misturados com o DNA genómico cortado. Uma aliquota da reacção de ligação é guardado como o ADN de entrada, enquanto o resto é submetido a purificação por afinidade usando uma resina Ni-NTA. A entrada e o DNA ligado a RR são todo o genoma amplificado e marcado com Cy3 e Cy5, respectivamente. O ADN marcado é reunidas e hibridado com uma matriz de ladrilhos, que é então analisado para determinar os alvos do gene. Figura modificada e reimpresso com a licença creative commons de 7.
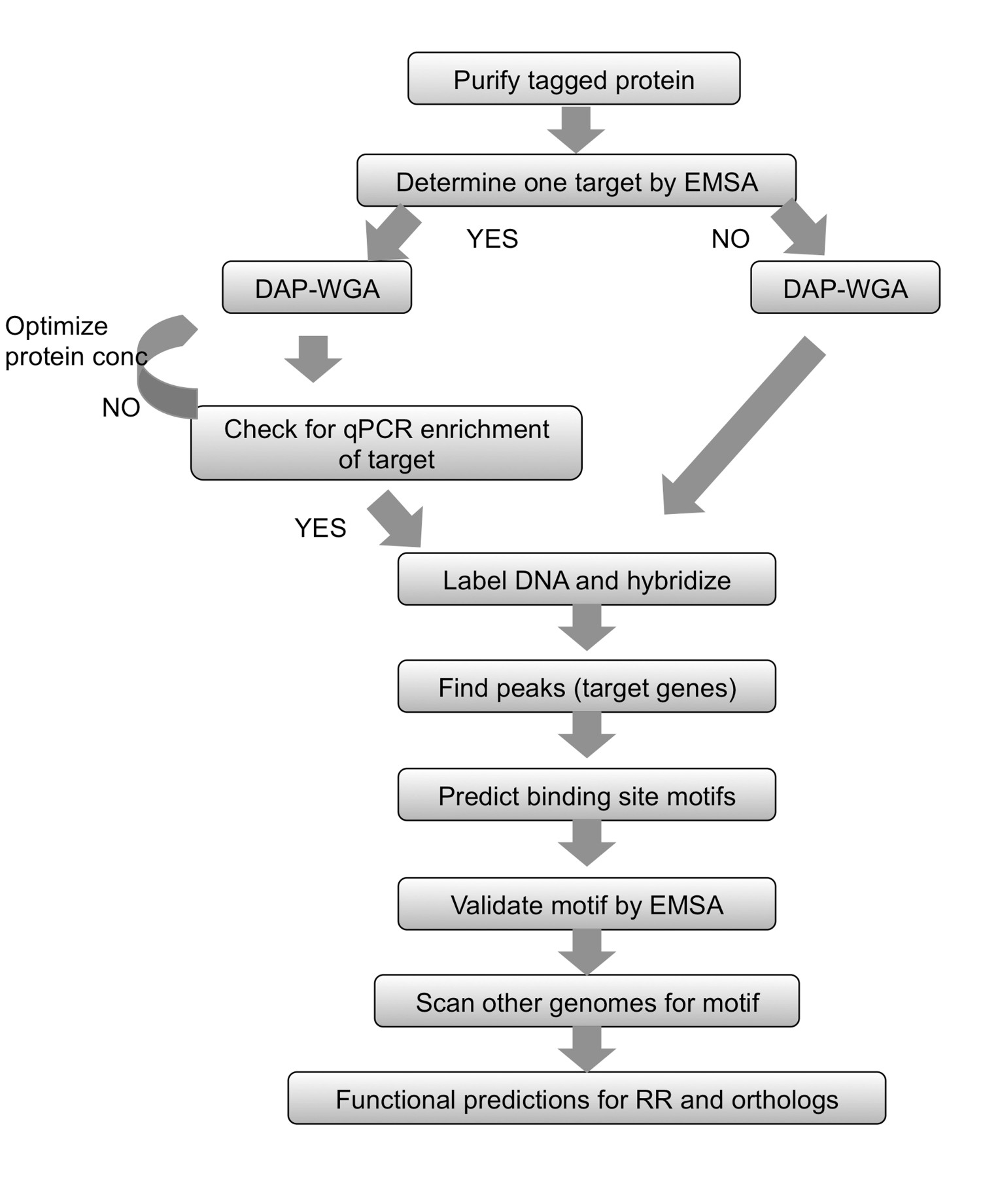
Figura 2. Resumo do fluxo de trabalho. Para qualquer Protei marcado purificadon, começar por determinar um alvo usando EMSA. Permitir proteína para ligar DNA genômico e DNA-afinidade-Purify (DAP) e todo o genoma amplificar (WGA) e do DNA de entrada enriquecido. Se um gene alvo é conhecido, usar qPCR para garantir que o alvo conhecido é enriquecido na fracção ligada a proteína. Se nenhum destino poderia ser determinado, prossiga diretamente para rotulagem de DNA e variedade de hibridização. Se o enriquecimento por qPCR não pôde ser observado, em seguida, repita a ligação proteína-gDNA e passos DAP-WGA usando diferentes quantidades de proteína. Use a análise de matriz para encontrar picos e mapeá-los para atingir genes. Use as regiões a montante de genes alvo de prever motivos local de ligação. Validar os motivos experimentalmente usando EMSAs. Use o motivo para digitalizar os genomas de espécies relacionadas que codificam ortólogos da RR em estudo, e prever genes alvo nas espécies também. Com base nos genes alvos obtidos, a função fisiológica da RR e seus ortólogos pode ser previsto. Figura modificada e reimpresso usando o c criativoommons licença de 7.
Protocol
Representative Results
Discussion
O método DAP-chip aqui descrito foi utilizado com sucesso para determinar os genes alvos para vários RRS em Desulfovibrio vulgaris Hildenborough 7 dos quais um é mostrado aqui como um resultado representativo. Para RR DVU3023, escolhendo um alvo gene candidato foi simples. DVU3025 está localizado imediatamente a jusante do gene RR, e os genes-alvo e RR são conservadas em diversas espécies Desulfovibrio, e adicionalmente DVU3025 tem um promotor dependente de sigma54 previsto. O EMSA for…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Agradecemos Amy Chen por sua ajuda na preparação para a filmagem do vídeo e para demonstrar a técnica. Este trabalho realizado pelo ENIGMA: Ecossistemas e redes integradas com os genes e Assembléias Molecular (http://enigma.lbl.gov), um programa científico da área de foco no Lawrence Berkeley National Laboratory, foi apoiado pelo Escritório de Ciência, Instituto de Ciências Biológicas e Pesquisa Ambiental, do Departamento de Energia dos EUA sob Contrato n º DE-AC02-05CH11231.
Materials
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
Riferimenti
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).

