DNA的亲和纯化的芯片(DAP芯片)方法来确定目标基因的细菌双组分调控系统
Summary
这个视频文章介绍了一种体外基于微阵列方法来确定两个组件系统响应监管机构的靶基因和结合位点。
Abstract
在体内方法,如芯片芯片是用于确定转录因子基因总体目标行之有效的方法。然而,它们的用途有限的探索细菌双组分调控系统具有未知功能的激活条件。这种系统只调节转录时的独特信号的存在被激活。由于这些信号往往是未知的,这个视频文章中描述的体外基于微阵列方法可用于确定基因目标和响应监管机构的结合位点。在一个测序基因组的任何生物体可以用于任何纯化的调节器将该DNA-亲和纯化的芯片的方法。该协议涉及允许纯化标记的蛋白结合到剪切基因组DNA,然后亲和纯化的蛋白结合的DNA,然后将DNA与杂交到自定义平铺阵列的荧光标记。前述可用于优化测定法SPECIFI步骤Ç监管部门也有所说明。由阵列数据分析所产生的峰被用来预测的结合位点基序,其然后通过实验验证。动机的预测可以进一步用于确定密切相关的物种的直系同源响应调节剂的靶基因。我们证明了该方法的适用性通过确定目标基因和结合位点基序,从而预测功能,用于在环境细菌脱硫寻常 Hildenborough一个sigma54依赖性反应调节DVU3023。
Introduction
细菌的生存和发展的能力严重依赖于他们如何能够感知并在其环境中的扰动做出响应,而这又取决于它们的信号转导系统。信令系统的细菌进行编码的数量被称为它的“微生物IQ”,可以是其环境的两个变异和其检测多个信号的能力并微调其反应1的指示。二组分信号转导系统(TCS)是用于由细菌最普遍的信令系统,并且它们包括感测外部信号,并通过磷酸化的效应反应器(RR)2发送一个组氨酸激酶(HK)的。反射器可以具有多种输出域,因此不同的效应器模式,但最常见的反应是通过DNA结合结构域1的转录调控。感测到的信号和输精管的相应功能吨多数个TCS的仍下落不明。
虽然体内方法,如芯片芯片通常用于测定的转录因子3基因组结合位点,它们只能用于细菌的双组分体系的RR,如果活化条件或信号是已知的。通常情况下,环境因素,激活一个TCS是难以确定比他们的靶基因。 在体外微阵列此处描述的基础的检测,可用于有效和快速地确定该基因的目标和预测的TCS的功能。该测定法利用了一个事实,即反射器能够被磷酸化,从而使用小分子献血者像乙酰磷酸4 体外活化的优势。
在该方法中,命名为DAP芯片用于DNA-亲和纯化的芯片( 图1)中,感兴趣的RR基因的克隆与在大肠杆菌中的His-标签大肠杆菌和纯化后的蛋白标记允许双向第二,以剪切基因组DNA。该蛋白结合的DNA,然后通过亲和纯化富集,富集和输入DNA被扩增,荧光标记,汇集在一起,并杂交到被定制到感兴趣的有机体( 图1)由一个平铺阵列。微阵列实验如有文物,因此额外的步骤来优化检测。一个这样的步骤是试图确定一个目标为RR正在研究利用电泳迁移率变动分析(EMSA)(见工作流在图2中)。然后,下面的结合到基因组DNA和DAP的步骤中,蛋白结合和输入DNA是通过定量PCR检查,以查看是否阳性靶富集的蛋白结合部分相对于输入级分,从而确认为最佳的结合条件RR( 图2)。阵列杂交后,对数据进行分析,发现较高的强度信号的指示的基因位点的峰,其中蛋白h广告的约束。功能可预测的基础上获得的基因目标的路由反射器。靶基因位点被用于预测的结合位点基序,然后将其实验使用EMSAs( 图2)进行验证。功能预测和靶基因的RR然后可以延伸到密切相关,通过扫描这些基因对于相似的结合基序( 图2)进行编码的直向同源物种的RR。民主行动党芯片法可以为TCS那里以前有没有提供了丰富的信息。该方法也可用于任何转录调节子,如果蛋白质可被纯化和DNA结合条件可以被确定,并且针对感兴趣与基因组序列的任何有机体提供。
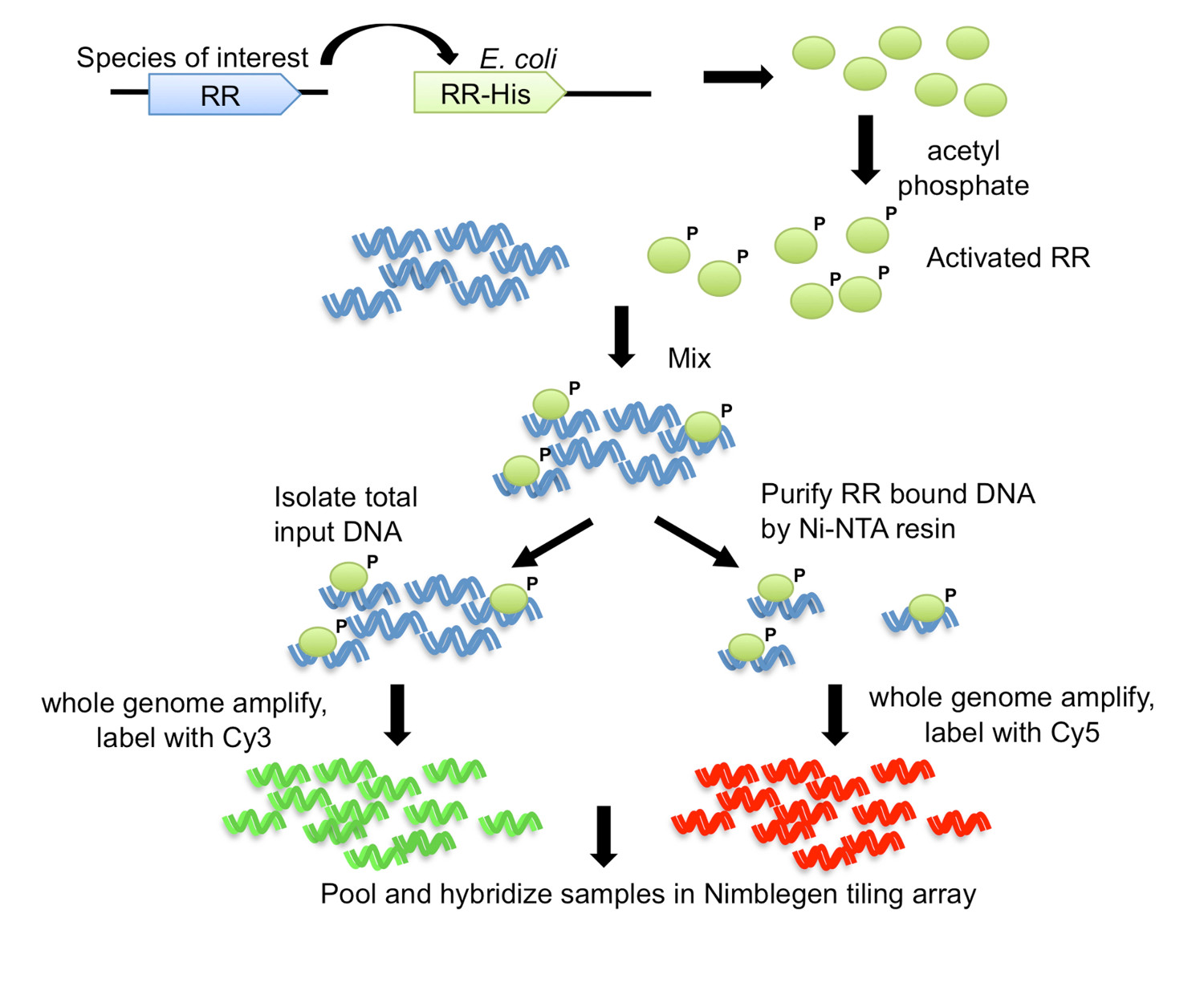
图1的DNA-亲和纯化的芯片(DAP-芯片)战略7。从感兴趣的有机体RR基因被克隆与羧基末端His-标签到大肠杆菌大肠杆菌表达菌株。纯化的His-标记的蛋白是由磷酸与乙磷酸激活,并与剪切基因组DNA混合。结合反应的等分试样保存为输入DNA,而其余的是使用Ni-NTA树脂进行亲和纯化。输入和RR结合的DNA是扩增整个基因组中,并标记有Cy3和Cy5分别。标记的DNA被汇集在一起,并杂交到一个平铺阵列中,然后进行分析以确定该基因的目标。图修改并使用Creative Commons授权从7转载。
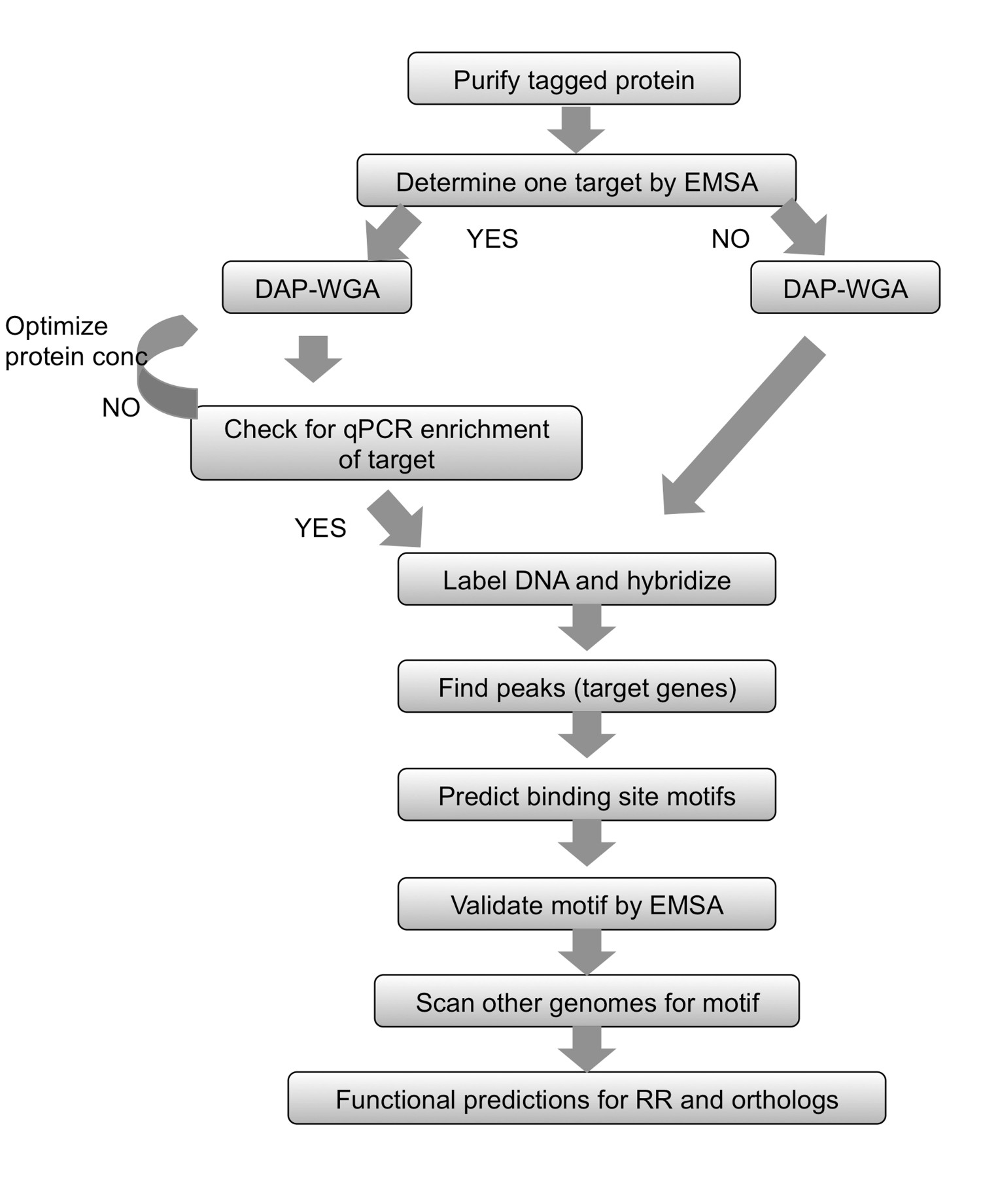
图2总结工作流程。对于任何纯化标签proteiN,利用EMSA确定目标开始。让蛋白结合的基因组DNA,然后DNA的亲和净化(DAP)和全基因组扩增(WGA)的丰富和输入的DNA。如果一个基因靶标是已知的,可使用定量PCR,以确保已知靶标富集的蛋白质结合的级分。如果没有目标可确定,直接进行DNA标记和阵列杂交。如果富集通过qPCR无法观察到的,然后重复蛋白基因组DNA结合,并使用不同的蛋白量DAP-WGA的步骤。利用阵列分析发现峰,并将其映射到目标基因。使用靶基因的上游区域,预测的结合位点基序。实验使用EMSAs验证图案。使用主题来扫描相关品种正在研究编码RR的同源基因的基因组,并预测目标基因在这些物种也是如此。根据所获得的基因目标,RR和其同源基因的生理功能可预测。图修改,并使用广告C转载ommons从7许可。
Protocol
Representative Results
Discussion
这里所描述的DAP芯片方法成功地用于确定多个路由反射器在脱硫寻常 Hildenborough 7其中之一是这里示出为具有代表性的结果的靶基因。对于RR DVU3023,选择一个候选基因的目标是简单的。 DVU3025位于紧接在RR基因的下游,并在RR和靶基因是保守的几个脱硫弧菌种,另外DVU3025具有预测的sigma54依赖型启动子。在EMSA提供了一种简单的方法来快速测试在RR为结合到候选靶基因,并且还允?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
我们感谢陈小姐为她准备的视频拍摄帮助和演示该技术。由ENIGMA进行这项工作:生态系统和网络集成与基因和分子组装(http://enigma.lbl.gov),一个科学的对焦区域计划在劳伦斯伯克利国家实验室,通过科学,生物办公室的办公室,并支持环境研究能源的合同号DE-AC02-05CH11231美系,。
Materials
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
Riferimenti
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).

