COSMIC ile Hücrelerde İlaç-DNA Etkileşimlerinin genom Haritalama (Küçük moleküllerinin çapraz bağlanmasını Kromatin yalıtmak için)
Summary
Genom hedefleme moleküllerinin doğrudan hedefleri belirlenmesi önemli bir sorun olmaya devam etmektedir. DNA bağlama molekülleri genomu meşgul anlamak için, kromatin izole etmek için küçük moleküllerin çapraz bağlama dayanan bir yöntem (COSMIC) geliştirdi.
Abstract
Genom en etkili kemoterapötik bazı hedef, ancak bu ilaçların en doz sınırlayıcı toksisite ve çok sayıda olumsuz yan etkilere neden DNA sekansı özgüllük yoksundur. Diziye özgü küçük moleküller ile genom hedefleme artan terapötik indeksi ve daha az hedef dışı etkileri olan molekülleri sağlayabilir. N-metilpirol / N-metilimidazol poliamidler rasyonel mükemmel hassasiyetle spesifik DNA dizileri hedeflemek için tasarlanmış olabilir moleküllerdir. Ve en doğal transkripsiyon faktörleri farklı olarak, polyamidler afinite kaybı olmaksızın metil ve chromatinized DNA'ya bağlanabilir. Poliamidlerin dizisi özgüllüğü yoğun aynı kökenli yerinin tanımlanması (CSI) ile in vitro olarak, geleneksel biyokimyasal ve biyofiziksel yaklaşımlarla ele alınmıştır, ancak hücrelerin genomik hedeflere bağlanabilen poliamidin çalışması zor olmaya devam etmektedir. Bir yöntemi rapor Burada, küçük moleküllerin çapraz bağlama için isolate kromatin (KOZMIK), genom çapında polyamid bağlanma yeri tanımlar. KOZMIK kromatin immunoprecipitation (chip) benzer olmakla birlikte, iki önemli noktada ayrılır: olarak bir foto çapraz seçici zamansal kontrollü polyamid bağlanma olgularının yakalama ve (2) biotin afinite kolu poliamid saflaştırmak için kullanılır sağlamak için kullanılır (1) Yarı denatüre edici koşullar altında a-DNA konjügatları kovalent olmayan bir şekilde bağlı olan DNA azaltmak için kullanılır. KOZMIK poliamitler ve diğer genom hedefleme kemoterapötik ajanların genom bağlayıcı olayları ortaya çıkarmak için kullanılan bir genel bir stratejidir.
Introduction
Bilgi, insan vücudunda her hücre DNA'sında kodlanmış yapmak. Bu bilginin seçici olarak kullanımı, bir hücrenin kaderi yönetir. Transkripsiyon faktörleri (TF) genomunda genlerin özel bir alt ifade özel DNA dizilerine bağlanan proteinler, ve TFS arıza gelişimsel bozukluklar, kanser, ve diyabet gibi hastalıkların geniş bir dizinin başlangıcından bağlantılıdır . 1,2 Biz seçici genom bağlanan ve gen düzenleyici ağlar modüle moleküller geliştirmek isteyen olmuştur.
Poliamidler N-metilpirol oluşan ve N, rakip doğal transkripsiyon faktörleri. 3-6 Bu moleküller DNA minör oyuk içinde oluşan özel dizilerine bağlanan özgüllükleri ve afiniteleri olan hedef DNA molekülleri olabilir metilimidazol rasyonel tasarlanmıştır. 4,5,7 -11 Polyamidler hem bastırmak için kullanılan ve spesifik g ifadesini aktive edilmişenes. 4,12-19 Ayrıca ilginç 20-24 anti-viral ve anti-kanser 12,13,25-30 özelliklere sahiptir. Poliamidlerin bir çekici özelliği 31,32 metillenmiş ve histon proteinleri 9,10,33 sarılı DNA dizilerini ulaşmak için yetenekleridir.
DNA bağlama moleküllerinin kapsamlı bir birleşme spesifisitelerine ölçmek için, laboratuar, 34-39, in vitro özgüllükleri (genomescapes) dayalı bağlanma yerlerinin tahmini oluşum genomu üzerinde görüntülenebilir. Kognat Alanı tanımlayıcısı (CSI) yöntemini yarattığı için in vitro bağlanma şiddetleri dernek sabitleri ile doğru orantılıdır (K a). 34,35,37 Bu genomescapes genom boyunca poliamid doluluk içgörü sağlar, ancak canlı hücrelerde bağlanmasını ölçme poliamid bir meydan okuma olmuştur. DNA, sıkı bir şekilde bağlanma sitelerinin erişilebilirliğini etkileyebilir çekirdek içinde paketlenir. Birpoliamidlere bu chromatinized DNA dizilerinin ccessibility bir gizem kalıyor.
Son zamanlarda, birçok yöntem ortaya çıkmıştır küçük moleküller ve nükleik asitler arasındaki etkileşimleri incelemek için. 40-48 kimyasal afinite yakalama ve kitlesel paralel DNA dizileme (kimya-seq) böyle bir tekniktir. Chem seq ligand-hedef etkileşimi yakalamak için ilgi konusu bir genomik hedef ve ilgi konusu bir küçük molekülün, bir biyotinile türevine küçük moleküller çapraz bağlanması için formaldehit kullanır. 48,49
Formaldehit çapraz bağlama hata üretebilirken dolaylı etkileşimleri yol açar. 50 Yeni bir yöntem, küçük moleküllerin çapraz bağlama olarak adlandırılan bu "hayalet" tepe ortadan kaldırmak için bir foto çapraz bağlayıcının ile kromatin (KOZMIK), 51 izole etmek için. 50 başlamak için geliştirilen biz tasarladık ve poliamitlerin üç işlevli türevleri sentezlendi. Bu moleküller, bir DNA-bağlama pol ihtivayamide bir Foto çapraz bağlayıcı (psoralen) ve bir afinite kolu (biotin, Şekil 1). Üç işlevli poliamidler ile, kovalent. 365 nm UV ışıması, DNA hasarı ya da olmayan psoralen-esaslı çapraz bağlanmasını yol açan bir dalga boyuna sahip poliamid-DNA etkileşimlerini yakalayabilir 51 Sonra, katı, yarı altında çekilen DNA genom fragman ve saflaştırmak -denaturing koşulları kovalent olmayan bir şekilde bağlı olan DNA azaltmak için kullanılır. Böylece, kimya-SEQ ilişkin bir yöntem olarak KOZMIK görebilir, ancak DNA 'nın daha doğrudan bir okuma ile hedeflenmesi. Önemli bir şekilde, zayıf (K 10 3 -10 4 M-1) DNA için psoralen afinitesi yapar saptanabilir etki poliamid spesifikliği. 51,52 zenginleştirilmiş DNA fragmanları kantitatif polimeraz zincir reaksiyonu ya da 51 ile analiz edilebilir (kozmik qPCR) ya da yeni nesil dizileme 53 (COSMIC-seq) tarafından. Bu veriler ligandların tarafsız bir genom güdümlü tasarım etkinleştirmek arasıOnların arzu genomik loci ile hareket ve hedef dışı etkilerini en aza indirmek.
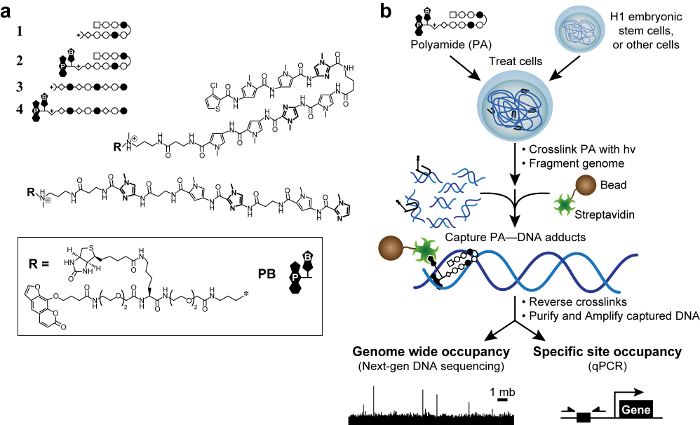
Şekil 1. Biyoaktif poliamidler ve KOZMİK düzeni. (A) Firkete poliamitler 1-2 hedef DNA sekansı 5'-WACGTW-3 '. Lineer poliamidler 3-4 hedef 5'-AAGAAGAAG-3 '. N-metilimidazol Rings netlik için kalın edilmektedir. Açık ve dolu daireler sırasıyla N-metilpirol ve N-metilimidazol temsil etmektedir. Kare 3-klorotiofen temsil eder ve elmas β-alanin temsil etmektedir. Psoralen biotin, sırasıyla, P ve B ile temsil edilir. (B) KOZMİK düzeni. Hücreler Poliamidlerin üç işlevli türevleri ile muamele edilmektedir. 365 nm UV ışınlama ile çapraz sonra, hücreler l vardırysed ve genomik DNA 'kırılır. Streptavidin-kaplı manyetik boncuklar, poliamid-DNA adüktleri yakalamak için ilave edilir. DNA serbest bırakılır ve kantitatif PCR (qPCR) veya yeni nesil dizileme (NGS) tarafından analiz edilebilir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
Protocol
Representative Results
Discussion
One of the primary challenges with conventional ChIP is the identification of suitable antibodies. ChIP depends heavily upon the quality of the antibody, and most commercial antibodies are unacceptable for ChIP. In fact, the Encyclopedia of DNA Elements (ENCODE) consortium found only 20% of commercial antibodies to be suitable for ChIP assays.50 With COSMIC, antibodies are replaced by streptavidin. Because polyamides are functionalized with biotin, streptavidin is used in place of an antibody to capture polyam…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
We thank members of the Ansari lab and Prof. Parameswaran Ramanathan for helpful discussions. This work was supported by NIH grants CA133508 and HL099773, the H. I. Romnes faculty fellowship, and the W. M. Keck Medical Research Award to A.Z.A. G.S.E. was supported by a Peterson Fellowship from the Department of Biochemistry and Molecular Biosciences Training Grant NIH T32 GM07215. A.E. was supported by the Morgridge Graduate Fellowship and the Stem Cell and Regenerative Medicine Center Fellowship, and D.B. was supported by the NSEC grant from NSF.
Materials
| Phenylmethylsulfonyl fluoride (PMSF) | any source | ||
| Benzamidine | any source | ||
| Pepstatin | any source | ||
| Proteinase K | any source | ||
| Dynabeads MyOne Streptavidin C1 | Life Technologies | 65001 | |
| PBS, pH 7.4 | Life Technologies | 10010-023 | Other sources can be used |
| StemPro® Accutase® Cell Dissociation Reagent | Life Technologies | A1110501 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | We have tried other manufacturers of DNA columns with success. |
| TruSeq ChIP Sample Prep Kit | Illumina | IP-202-1012 | This Kit can be used to prepare COSMIC DNA for next-generation sequencing |
| Matrigel Basement Membrane Matrix | BD Biosciences | 356231 | Used to coat plates in order to grow H1 ESCs |
| pH paper | any source | ||
| amber microcentrifuge tubes | any source | ||
| microcentrifuge tubes | any source | ||
| pyrex filter | any source | Pyrex baking dishes are suitable | |
| qPCR master mix | any source | ||
| RNase | any source | ||
| HCl (6 N) | any source | ||
| 10-cm tissue culture dishes | any source | ||
| Serological pipettes | any source | ||
| Pasteur pipettes | any source | ||
| Pipette tips | any source | ||
| 15-mL conical tubes | any source | ||
| centrifuge | any source | ||
| microcentrifuge | any source | ||
| nutator | any source | ||
| Magnetic separation rack | any source | ||
| UV source | CalSun | B001BH0A1A | Other UV sources can be used, but crosslinking time must be optimized empirically |
| Misonix Sonicator | Qsonica | S4000 with 431C1 cup horn | Other sonicators can be used, but sonication conditions must be optimized empirically |
| Humidified CO2 incubator | any source | ||
| Biological safety cabinet with vacuum outlet | any source |
Riferimenti
- Lee, T. I., Young, R. A. Transcriptional Regulation and Its Misregulation in Disease. Cell. 152, 1237-1251 (2013).
- Vaquerizas, J. M., Kummerfeld, S. K., Teichmann, S. A., Luscombe, N. M. A census of human transcription factors: function, expression and evolution. Nat Rev Genet. 10, 252-263 (2009).
- Meier, J. L., Yu, A. S., Korf, I., Segal, D. J., Dervan, P. B. Guiding the Design of Synthetic DNA-Binding Molecules with Massively Parallel Sequencing. J. Am. Chem. Soc. 134, 17814-17822 (2012).
- Dervan, P. B. Molecular recognition of DNA by small molecules. Bioorg. Med. Chem. 9, 2215-2235 (2001).
- Wemmer, D. E., Dervan, P. B. Targeting the minor groove of DNA. Curr. Opin. Struct. Biol. 7, 355-361 (1997).
- Eguchi, A., Lee, G. O., Wan, F., Erwin, G. S., Ansari, A. Z. Controlling gene networks and cell fate with precision-targeted DNA-binding proteins and small-molecule-based genome readers. Biochem. J. 462, 397-413 (2014).
- Mrksich, M., et al. Antiparallel side-by-side dimeric motif for sequence-specific recognition in the minor groove of DNA by the designed peptide 1-methylimidazole-2-carboxamide netropsin. Proc Natl Acad Sci U S A. 89, 7586-7590 (1992).
- Edayathumangalam, R. S., Weyermann, P., Gottesfeld, J. M., Dervan, P. B., Luger, K. Molecular recognition of the nucleosomal “supergroove”. Proc Natl Acad Sci U S A. 101, 6864-6869 (2004).
- Suto, R. K., et al. Crystal structures of nucleosome core particles in complex with minor groove DNA-binding ligands. J. Mol. Biol. 326, 371-380 (2003).
- Gottesfeld, J. M., et al. Sequence-specific Recognition of DNA in the Nucleosome by Pyrrole-Imidazole Polyamides. J. Mol. Biol. 309, 615-629 (2001).
- Chenoweth, D. M., Dervan, P. B. Structural Basis for Cyclic Py-Im Polyamide Allosteric Inhibition of Nuclear Receptor Binding. J. Am. Chem. Soc. 132, 14521-14529 (2010).
- Raskatov, J. A., et al. Modulation of NF-κB-dependent gene transcription using programmable DNA minor groove binders. Proc. Natl. Acad. Sci. U.S.A. 109, 1023-1028 (2012).
- Yang, F., et al. Antitumor activity of a pyrrole-imidazole polyamide. Proc. Natl. Acad. Sci. U.S.A. 110, 1863-1868 (2013).
- Mapp, A. K., Ansari, A. Z., Ptashne, M., Dervan, P. B. Activation of gene expression by small molecule transcription factors. Proc. Natl. Acad. Sci. U.S.A. 97, 3930-3935 (2000).
- Ansari, A. Z., Mapp, A. K., Nguyen, D. H., Dervan, P. B., Ptashne, M. Towards a minimal motif for artificial transcriptional activators. Chem. Biol. 8, 583-592 (2001).
- Arora, P. S., Ansari, A. Z., Best, T. P., Ptashne, M., Dervan, P. B. Design of artificial transcriptional activators with rigid poly-L-proline linkers. J. Am. Chem. Soc. 124, 13067-13071 (2002).
- Nickols, N. G., Jacobs, C. S., Farkas, M. E., Dervan, P. B. Modulating Hypoxia-Inducible Transcription by Disrupting the HIF-1–DNA Interface. ACS Chemical Biology. 2, 561-571 (2007).
- Pandian, G. N., et al. A synthetic small molecule for rapid induction of multiple pluripotency genes in mouse embryonic fibroblasts. Sci. Rep. 2, 544 (2012).
- Pandian, G. N., et al. Synthetic Small Molecules for Epigenetic Activation of Pluripotency Genes in Mouse Embryonic Fibroblasts. Chem Bio Chem. 12, 2822-2828 (2011).
- He, G., et al. Binding studies of a large antiviral polyamide to a natural HPV sequence. Biochimie. 102, 83-91 (2014).
- Edwards, T. G., Vidmar, T. J., Koeller, K., Bashkin, J. K., Fisher, C. DNA Damage Repair Genes Controlling Human Papillomavirus (HPV) Episome Levels under Conditions of Stability and Extreme Instability. PLoS ONE. 8, e75406 (2013).
- Edwards, T. G., Helmus, M. J., Koeller, K., Bashkin, J. K., Fisher, C. HPV Episome Stability is Reduced by Aphidicolin and Controlled by DNA Damage Response Pathways. Journal of Virology. , (2013).
- Edwards, T. G., et al. HPV episome levels are potently decreased by pyrrole-imidazole polyamides. Antiviral Res. 91, 177-186 (2011).
- Dickinson, L. A., et al. Inhibition of RNA polymerase II transcription in human cells by synthetic DNA-binding ligands. Proc. Natl. Acad. Sci. U.S.A. 95, 12890-12895 (1998).
- Dickinson, L. A., et al. Arresting Cancer Proliferation by Small-Molecule Gene Regulation. Chem. Biol. 11, 1583-1594 (2004).
- Nickols, N. G., et al. Activity of a Py–Im Polyamide Targeted to the Estrogen Response Element. Molecular Cancer Therapeutics. 12, 675-684 (2013).
- Raskatov, J. A., Puckett, J. W., Dervan, P. B. A C-14 labeled Py–Im polyamide localizes to a subcutaneous prostate cancer tumor. Bioorg. Med. Chem. 22, 4371-4375 (2014).
- Jespersen, C., et al. Chromatin structure determines accessibility of a hairpin polyamide–chlorambucil conjugate at histone H4 genes in pancreatic cancer cells. Bioorg. Med. Chem. Lett. 22, 4068-4071 (2012).
- Chou, C. J., et al. Small molecules targeting histone H4 as potential therapeutics for chronic myelogenous leukemia. Molecular Cancer Therapeutics. 7, 769-778 (2008).
- Nickols, N. G., Dervan, P. B. Suppression of androgen receptor-mediated gene expression by a sequence-specific DNA-binding polyamide. Proc. Natl. Acad. Sci. U.S.A. 104, 10418-10423 (2007).
- Minoshima, M., Bando, T., Sasaki, S., Fujimoto, J., Sugiyama, H. Pyrrole-imidazole hairpin polyamides with high affinity at 5CGCG3 DNA sequence; influence of cytosine methylation on binding. Nucleic Acids Res. 36, 2889-2894 (2008).
- Warren, C. L., et al. Fabrication of duplex DNA microarrays incorporating methyl-5-cytosine. Lab on a Chip. 12, 376-380 (2012).
- Dudouet, B., et al. Accessibility of nuclear chromatin by DNA binding polyamides. Chem. Biol. 10, 859-867 (2003).
- Carlson, C. D., et al. Specificity landscapes of DNA binding molecules elucidate biological function. Proc. Natl. Acad. Sci. U.S.A. 107, 4544-4549 (2010).
- Warren, C. L., et al. Defining the sequence-recognition profile of DNA-binding molecules. Proc. Natl. Acad. Sci. U.S.A. 103, 867-872 (2006).
- Tietjen, J. R., Donato, L. J., Bhimisaria, D., Ansari, A. Z., Voigt, C. Chapter One – Sequence-Specificity and Energy Landscapes of DNA-Binding Molecules. Methods Enzymol. 497, 3-30 (2011).
- Puckett, J. W., et al. Quantitative microarray profiling of DNA-binding molecules. J. Am. Chem. Soc. 129, 12310-12319 (2007).
- Keles, S., Warren, C. L., Carlson, C. D., Ansari, A. Z. CSI-Tree: a regression tree approach for modeling binding properties of DNA-binding molecules based on cognate site identification (CSI) data. Nucleic Acids Res. 36, 3171-3184 (2008).
- Hauschild, K. E., Stover, J. S., Boger, D. L., Ansari, A. Z. CSI-FID: High throughput label-free detection of DNA binding molecules. Bioorg. Med. Chem. Lett. 19, 3779-3782 (2009).
- Lee, M., Roldan, M. C., Haskell, M. K., McAdam, S. R., Hartley, J. A. . In vitro Photoinduced Cytotoxicity and DNA Binding Properties of Psoralen and Coumarin Conjugates of Netropsin Analogs: DNA Sequence-Directed Alkylation and Cross-Link. 37, 1208-1213 (1994).
- Wurtz, N. R., Dervan, P. B. Sequence specific alkylation of DNA by hairpin pyrrole–imidazole polyamide conjugates. Chem. Biol. 7, 153-161 (2000).
- Tung, S. -. Y., Hong, J. -. Y., Walz, T., Moazed, D., Liou, G. -. G. Chromatin affinity-precipitation using a small metabolic molecule: its application to analysis of O-acetyl-ADP-ribose. Cell. Mol. Life Sci. 69, 641-650 (2012).
- Rodriguez, R., Miller, K. M. Unravelling the genomic targets of small molecules using high-throughput sequencing. Nat Rev Genet. 15, 783-796 (2014).
- Guan, L., Disney, M. D. Covalent Small-Molecule–RNA Complex Formation Enables Cellular Profiling of Small-Molecule–RNA Interactions. Angew. Chem. Int. Ed. 52, 10010-10013 (2013).
- White, J. D., et al. Picazoplatin, an Azide-Containing Platinum(II) Derivative for Target Analysis by Click Chemistry. J. Am. Chem. Soc. 135, 11680-11683 (2013).
- Rodriguez, R., et al. Small-molecule–induced DNA damage identifies alternative DNA structures in human genes. Nat Chem Biol. 8, 301-310 (2012).
- Bando, T., Sugiyama, H. Synthesis and Biological Properties of Sequence-Specific DNA-Alkylating Pyrrole−Imidazole Polyamides. Acc. Chem. Res. 39, 935-944 (2006).
- Anders, L., et al. Genome-wide localization of small molecules. Nat. Biotechnol. 32, 92-96 (2014).
- Jin, C., et al. Chem-seq permits identification of genomic targets of drugs against androgen receptor regulation selected by functional phenotypic screens. Proc. Natl. Acad. Sci. U.S.A. 111, 9235-9240 (2014).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
- Erwin, G. S., Bhimsaria, D., Eguchi, A., Ansari, A. Z. Mapping Polyamide–DNA Interactions in Human Cells Reveals a New Design Strategy for Effective Targeting of Genomic Sites. Angew. Chem. Int. Ed. 53, 10124-10128 (2014).
- Hyde, J. E., Hearst, J. E. Binding of psoralen derivatives to DNA and chromatin: influence of the ionic environment on dark binding and photoreactivity. Biochimica. 17, 1251-1257 (1978).
- Erwin, G. S., Bhimsaria, D., Rodríguez-Martínez, J. A., Grieshop, M. P., Ansari, A. Z. Genome-wide localization of polyamide-based genome readers reveals sequence-based binding to repressive heterochromatin. In preparation. , (2015).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Meth. 8, 424-429 (2011).
- Deliard, S., Zhao, J., Xia, Q., Grant, S. F. A. Generation of High Quality Chromatin Immunoprecipitation DNA Template for High-throughput Sequencing (ChIP-seq). J Vis Exp. (74), e50286 (2013).
- Shi, Y. B., Spielmann, H. P., Hearst, J. E. Base-catalyzed reversal of a psoralen-DNA cross-link. Biochimica. 27, 5174-5178 (1988).
- Kumaresan, K. R., Hang, B., Lambert, M. W. Human Endonucleolytic Incision of DNA 3′ and 5′ to a Site-directed Psoralen Monoadduct and Interstrand. J. Biol. Chem. 270, 30709-30716 (1995).
- Cimino, G. D., Shi, Y. B., Hearst, J. E. Wavelength dependence for the photoreversal of a psoralen-DNA crosslink. Biochimica. 25, 3013-3020 (1986).
- Heinz, S., et al. Simple Combinations of Lineage-Determining Transcription Factors Prime cis-Regulatory Elements Required for Macrophage and B Cell Identities. Mol. Cell. 38, 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9, R137 (2008).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nat Biotech. 26, 1351-1359 (2008).
- Diamandis, E. P., Christopoulos, T. K. The biotin-(strept)avidin system: principles and applications in biotechnology. Clin. Chem. 37, 625-636 (1991).
- Martinson, H. G., True, R. J. On the mechanism of nucleosome unfolding. Biochimica. 18, 1089-1094 (1979).
- Gloss, L. M., Placek, B. J. The Effect of Salts on the Stability of the H2A−H2B Histone Dimer. Biochimica. 41, 14951-14959 (2002).
- Jackson, V. Formaldehyde Cross-Linking for Studying Nucleosomal Dynamics. Methods. 17, 125-139 (1999).
- Kasinathan, S., Orsi, G. A., Zentner, G. E., Ahmad, K., Henikoff, S. High-resolution mapping of transcription factor binding sites on native chromatin. Nat Meth. 11, 203-209 (2014).
- Teytelman, L., Thurtle, D. M., Rine, J., van Oudenaarden, A. Highly expressed loci are vulnerable to misleading ChIP localization of multiple unrelated proteins. Proc. Natl. Acad. Sci. U.S.A. 110, 18602-18607 (2013).
- . Phantompeakqualtools home page Available from: https://www.encodeproject.org/software/phantompeakqualtools/ (2010)
- Wang, D., Lippard, S. J. Cellular processing of platinum anticancer drugs. Nature Reviews Drug Discovery. 4, 307-320 (2005).
- Hurley, L. H. DNA and its associated processes as targets for cancer therapy. Nat. Rev. Cancer. 2, 188-200 (2002).

