Generering CRISPR / Cas9 mediert Monoallelic Slett å studere Enhancer funksjon i mus embryonale stamceller
Summary
Experimental validation of enhancer activity is best approached by loss-of-function analysis. Presented here is an efficient protocol that uses CRISPR/Cas9 mediated deletion to study allele-specific regulation of gene transcription in F1 ES cells which contain a hybrid genome (Mus musculus129 x Mus castaneus).
Abstract
Enhancers control cell identity by regulating tissue-specific gene expression in a position and orientation independent manner. These enhancers are often located distally from the regulated gene in intergenic regions or even within the body of another gene. The position independent nature of enhancer activity makes it difficult to match enhancers with the genes they regulate. Deletion of an enhancer region provides direct evidence for enhancer activity and is the gold standard to reveal an enhancer’s role in endogenous gene transcription. Conventional homologous recombination based deletion methods have been surpassed by recent advances in genome editing technology which enable rapid and precisely located changes to the genomes of numerous model organisms. CRISPR/Cas9 mediated genome editing can be used to manipulate the genome in many cell types and organisms rapidly and cost effectively, due to the ease with which Cas9 can be targeted to the genome by a guide RNA from a bespoke expression plasmid. Homozygous deletion of essential gene regulatory elements might lead to lethality or alter cellular phenotype whereas monoallelic deletion of transcriptional enhancers allows for the study of cis-regulation of gene expression without this confounding issue. Presented here is a protocol for CRISPR/Cas9 mediated deletion in F1 mouse embryonic stem (ES) cells (Mus musculus129 x Mus castaneus). Monoallelic deletion, screening and expression analysis is facilitated by single nucleotide polymorphisms (SNP) between the two alleles which occur on average every 125 bp in these cells.
Introduction
Transkripsjonsregulerende elementer er avgjørende for rom-tid-finjustering av genuttrykk under utvikling en og modifikasjon av disse elementene kan føre til sykdom på grunn av avvikende genuttrykk to. Mange sykdomsassosierte regioner identifisert av genom brede assosiasjonsstudier er i ikke-kodende regioner og har funksjoner av transkripsjonsforsterkere 3-4. Identifisere Forsterker og matche dem med genene de regulerer er komplisert som de ofte ligger flere kilobaser bort fra genene de regulerer og kan aktiveres i en vev-spesifikk måte 5-6. Enhancer spådommer er ofte basert på histone modifikasjon merker, mediator-cohesin komplekser og binding av celletype-spesifikke transkripsjonsfaktorer 7-10. Validering av forutsagte enhancers gjøres oftest gjennom en vektor basert analyse hvor forsterkeren aktiverer ekspresjon av et reportergen 11-12. Disse dataene gir valuable informasjon om regulatoriske potensialet antatte forsterkersekvenser, men ikke avsløre sin funksjon i deres endogene genomisk kontekst eller identifisere genene de regulerer. Genome redigering fungerer som et kraftig verktøy for å studere funksjonen til transkripsjonsregulerende elementer i deres endogene sammenheng med tap-av-funksjon analyse.
Nylige fremskritt innen genom redigering, nemlig CRISPR / Cas9 genom redigering systemet, lette etterforskningen av genomet funksjon. Den CRISPR / Cas9 systemet er enkelt å bruke og kan tilpasses for mange biologiske systemer. Den Cas9 protein er rettet mot et bestemt sted i genomet av en guide RNA (gRNA) 13. Den SpCas9 / gRNA komplekse skanner genomet for sin target genomisk sekvens som må være 5 'til en protospacer tilstøtende motiv (PAM) sekvens, NGG 14-15. Base sammenkobling av gRNA til sitt mål, en 20 nukleotid (nt) sekvens komplementær til gRNA, aktiverer SpCas9 nukleaseaktiviteten resulterer i en dobbee tråd pause (DSB) 3 bp oppstrøms for PAM-sekvensen. Spesifisitet oppnås ved fullstendig baseparing i den gRNA frø regionen, 6-12 nt tilstøtende til PAM; omvendt, ikke samsvarer med 5 'av frøet blir vanligvis tolereres 16-17. Den introduserte DSB kan repareres enten ved ikke-homolog ende bli (NHEJ) DNA reparasjon eller homologi rettet reparasjon (HDR) mechanisms.NHEJ DNA-reparasjon ofte skaper innsetting / sletting (indels) av noen bp på målstedet som kan forstyrre den åpne leseramme (ORF) av et gen. For å generere større slettinger i genomet to gRNAs, som flankerer regionen av interesse, kan brukes 18-19. Denne fremgangsmåten er spesielt nyttig for studiet av transkripsjonsforsterkere som smyger seg inn i locus kontrollregioner eller super-forsterkere som er større enn konvensjonelle forsterkere 9,18,20-22.
Monoallelic slettinger er en verdifull modell for å studere cis -regulation av transkripsjon. Den observerte change i transkripsjon nivå etter monoallelic sletting av en forsterker korrelerer til rollen som forsterker i genregulering uten konfunderende effekter som kan oppstå når transkripsjon av begge alleler påvirkes potensielt påvirke celle fitness. Vurderer redusert uttrykk er vanskelig men uten evne til å skille slettet fra villtype-allelet. Videre genotyping slettinger i hvert allel uten evne til å skille de to alleler er krevende, særlig for store delesjoner av> 10 kb til en Mb 23 i hvilken det er vanskelig å forsterke hele villtype-regionen med PCR. Bruken av F1 ES-celler som genereres av krysset Mus musculus 129 med Mus castaneus gjør at to alleler lett å skille allel-spesifikk PCR 18,24. Den hybride genomet i disse cellene letter allel-spesifikk delesjon og ekspresjon screening-analyse. I gjennomsnitt er det en SNP hver 125 bp mellom disse to genomer, Som gir fleksibilitet i primer design for uttrykk og genotyping analyser. Tilstedeværelsen av en SNP kan påvirke tennsatsen smeltetemperaturen (T m) og rettet mot spesifisitet i sanntid kvantitativ PCR (qPCR) amplifikasjon slik at for diskriminering av de to allelene 25. Videre er en mistilpasning i 3'-enden av primeren har stor innvirkning på evnen av DNA-polymerase for å strekke seg fra primeren å forhindre forsterkning av den uønskede allel målet 26. Beskrevet i følgende protokoll er bruk av F1 ES-celler for allel spesifikk forsterkningsslettinger som er større enn 1 kb og påfølgende uttrykk analyse ved hjelp av CRISPR / Cas9 genom redigering systemet (figur 1).
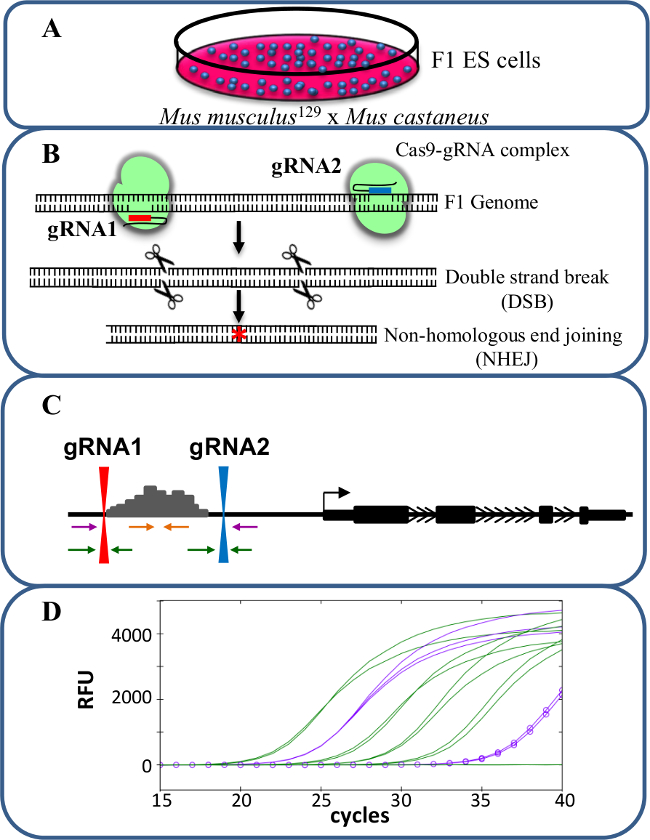
Figur 1. Enhancer sletting bruker CRISPR / Cas9 å studere cis -reggler av genuttrykk. (A) F1 ES-celler som genereres av en krysning mellom Mus musculus 129 og Mus castaneus brukes til å tillate for allel bestemt sletting. (B) To førings RNA (gRNA) blir brukt for å indusere en stor Cas9-mediert sletting av forøker regionen. (C) Primer sett brukes for å identifisere store mono- og bi-allele delesjoner. De oransje primere er inne primere, lilla primere er utenfor primere og de grønne primere blir gRNA flankerer primere. (D) Endringer i genuttrykk overvåkes ved hjelp av allel-spesifikk qPCR. RFU betegner relative fluorescens enheter. Klikk her for å se en større versjon av dette tallet.
Protocol
Representative Results
Discussion
CRISPR / Cas9 formidlet genom redigering teknologien gir en enkel, rask og billig metode for genom modifikasjon. Metoden beskrevet her for å generere og analysere monoallelic Enhancer sletting for funksjonell Enhancer karakterisering dro fordel av SNPs i F1 museceller. Fordelene med denne typen metode er: 1) monoallelic enhancer delesjoner ikke frembringer samtidig andre virkninger som oppstår når et kritisk enhancer er slettet fra begge allelene, det vil si, en stor reduksjon i protein nivåene av det regul…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
We would like to thank all the members of the Mitchell lab for helpful discussions. This work was supported by the Canadian Institutes of Health Research, the Canada Foundation for Innovation and the Ontario Ministry of Research and Innovation (operating and infrastructure grants held by JAM).
Materials
| Phusion High-Fidelity DNA Polymerase | NEB | M0530S | high fidelity DNA polymerase used in gRNA assembly |
| Gibson Assembly Master Mix | NEB | E2611L | |
| gRNA_Cloning Vector | Addgene | 41824 | A target sequence is cloned into this vector to create the gRNA plasmid |
| pCas9_GFP | Addgene | 44719 | Codon-optimized SpCas9 and EGFP co-expression plasmid |
| AflII | NEB | R0520S | |
| EcoRI | NEB | R3101S | |
| Neon Transfection System 100 µL Kit | Life Technologies | MPK10096 | Microporator transfection technology |
| prepGEM | ZyGEM | PT10500 | genomic DNA extraction reagent |
| Nucleo Spin Gel & PCR Clean-up | Macherey-Nagel | 740609.5 | |
| High-Speed Plasmid Mini Kit | Geneaid | PD300 | |
| Maxi Plasmid Kit Endotoxin Free | Geneaid | PME25 | |
| SYBR select mix for CFX | Life Technologies | 4472942 | qPCR reagent |
| iScript cDNA synthesis kit | Bio-rad | 170-8891 | Reverse transcription reagent |
| 0.25% Trypsin with EDTA | Life Technologies | 25200072 | |
| PBS without Ca/Mg2+ | Sigma | D8537 | |
| 0.5M EDTA | Bioshop | EDT111.500 | |
| HBSS | Life Technologies | 14175095 | |
| 1M HEPES | Life Technologies | 13630080 | |
| BSA fraction V (7.5%) | Life Technologies | 15260037 | |
| Max Efficiency DH5α competent cells | Invitrogen | 18258012 | |
| FBS | ES cell qualified | FBS is subjected to a prior testing in mouse ES cells for pluripotency | |
| DMSO | Sigma | D2650 | |
| Glutamax | Invitrogen | 35050 | |
| DMEM | Life Technologies | 11960069 | |
| Pencillin/Streptomycin | Invitrogen | 15140 | |
| Sodium pyruvate | Invitrogen | 11360 | |
| Non-essential aminoacid | Invitrogen | 11140 | |
| β-mercaptoethanol | Sigma | M7522 | |
| 96-well plate | Sarstedt | 83.3924 | |
| Sealing tape | Sarstedt | 95.1994 | |
| CoolCell LX | Biocision | BCS-405 | alcohol-free cell freezing container |
| CHIR99021 | Biovision | 1748-5 | Inhibitor for F1 ES cell culture |
| PD0325901 | Invivogen | inh-pd32 | Inhibitor for F1 ES cell culture |
| LIF | Chemicon | ESG1107 | Inhibitor for F1 ES cell culture |
Riferimenti
- Sagai, T., Hosoya, M., Mizushina, Y., Tamura, M., Shiroishi, T. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb. Development. 132 (4), 797-803 (2005).
- Kleinjan, D. A., Lettice, L. A. Long-range gene control and genetic disease. Adv Genet. 61, 339-388 (2008).
- Visel, A., Rubin, E. M., Pennacchio, L. A. Genomic views of distant-acting enhancers. Nature. 461 (7261), 199-205 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Shen, Y., et al. A map of the cis-regulatory sequences in the mouse genome. Nature. 488 (7409), 116-120 (2012).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147 (6), 1408-1419 (2011).
- Whyte, W. A., et al. Master transcription factors and mediator establish super-enhancers at key cell identity genes. Cell. 153 (2), 307-319 (2013).
- Chen, C. Y., Morris, Q., Mitchell, J. A. Enhancer identification in mouse embryonic stem cells using integrative modeling of chromatin and genomic features. BMC Genomics. 13 (1), 152 (2012).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Melnikov, A., et al. Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay. Nat Biotechnol. 30 (3), 271-277 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Cho, S. W., et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24 (1), 132-141 (2014).
- Zhou, H. Y., et al. A Sox2 distal enhancer cluster regulates embryonic stem cell differentiation potential. Genes Dev. 28 (24), 2699-2711 (2014).
- Fujii, W., Kawasaki, K., Sugiura, K., Naito, K. Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease. Nucleic Acids Res. 41 (20), e187 (2013).
- Tuan, D. Y., Solomon, W. B., London, I. M., Lee, D. P. An erythroid-specific, developmental-stage-independent enhancer far upstream of the human ‘beta-like globin’ genes. Proc Natl Acad Sci U S A. 86 (8), 2554-2558 (1989).
- Amano, T., et al. Chromosomal dynamics at the Shh locus: limb bud-specific differential regulation of competence and active transcription. Dev Cell. 16 (1), 47-57 (2009).
- Li, Y., et al. CRISPR reveals a distal super-enhancer required for Sox2 expression in mouse embryonic stem cells. PLoS One. 9 (12), e114485 (2014).
- Canver, M. C., et al. Characterization of genomic deletion efficiency mediated by clustered regularly interspaced palindromic repeats (CRISPR)/Cas9 nuclease system in mammalian cells. J Biol Chem. 289 (31), 21312-21324 (2014).
- Mlynarczyk-Evans, S., et al. X chromosomes alternate between two states prior to random X-inactivation. PLoS Biol. 4 (6), e159 (2006).
- Lefever, S., Pattyn, F., Hellemans, J., Vandesompele, J. Single-nucleotide polymorphisms and other mismatches reduce performance of quantitative PCR assays. Clin Chem. 59 (10), 1470-1480 (2013).
- Huang, M. M., Arnheim, N., Goodman, M. F. Extension of base mispairs by Taq DNA polymerase: implications for single nucleotide discrimination in PCR. Nucleic Acids Res. 20 (17), 4567-4573 (1992).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Yalcin, B., et al. Sequence-based characterization of structural variation in the mouse genome. Nature. 477 (7364), 326-329 (2011).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Gibson, D. G., Smith, H. O., Hutchison, C. A., Venter, J. C., Merryman, C. Chemical synthesis of the mouse mitochondrial genome. Nat Methods. 7 (11), 901-903 (2010).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell. 12 (4), 393-394 (2013).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of specific cell population by fluorescence activated cell sorting (FACS). J Vis Exp. (41), (2010).
- Forlenza, M., Kaiser, T., Savelkoul, H. F., Wiegertjes, G. F. The use of real-time quantitative PCR for the analysis of cytokine mRNA levels. Methods Mol Biol. 820, 7-23 (2012).
- Wu, J. H., Hong, P. Y., Liu, W. T. Quantitative effects of position and type of single mismatch on single base primer extension. J Microbiol Methods. 77 (3), 267-275 (2009).
- Sanyal, A., Lajoie, B. R., Jain, G., Dekker, J. The long-range interaction landscape of gene promoters. Nature. 489 (7414), 109-113 (2012).

