Screening per Funzionale non codificante Varianti genetiche Utilizzando Electrophoretic Mobility Shift Assay (EMSA) e DNA-affinità precipitazioni Assay (DAPA)
Summary
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Abstract
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Introduction
Sequencing e studi di genotipizzazione basato, compresi gli studi Genome-Wide Association (GWAS), studi locus candidato, e profondo-sequenziamento studi, hanno identificato molte varianti genetiche che sono statisticamente associati con una malattia, caratteristica, o fenotipo. Contrariamente alle previsioni iniziali, la maggior parte di queste varianti (85-93%) si trovano in regioni non codificanti e non cambiano la sequenza di amminoacidi delle proteine 1,2. Interpretando la funzione di queste varianti non codificanti e determinare i meccanismi biologici che li collegano alla malattia associata, tratto, o fenotipo è dimostrato impegnativo 3-6. Abbiamo sviluppato una strategia generale di identificare i meccanismi molecolari che collegano le varianti di un importante fenotipo intermedio – l'espressione genica. Questo gasdotto è specificamente progettato per identificare la modulazione di legare da varianti genetiche TF. Questa strategia combina approcci computazionali e tecniche di biologia molecolare finalizzate a prevedereeffetti biologici delle varianti candidati in silico e verificare empiricamente queste previsioni (Figura 1).
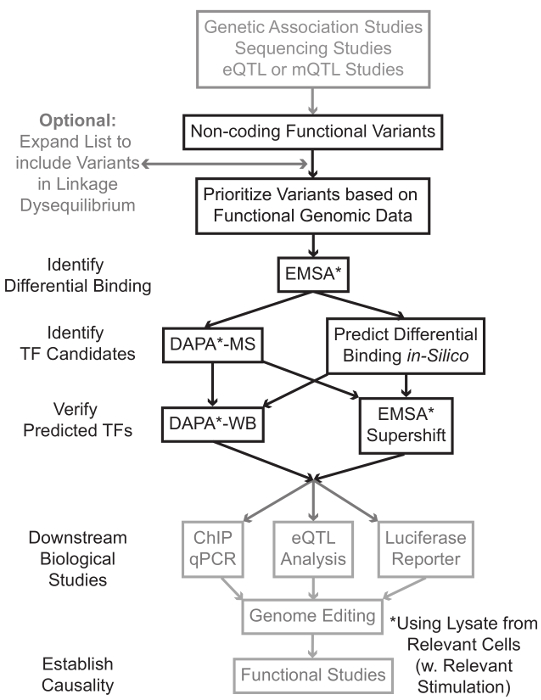
Figura 1:.. Approccio strategico per l'analisi dei passaggi non codificanti varianti genetiche che non sono inclusi nel protocollo dettagliato associato a questo manoscritto sono di colore grigio Clicca qui per vedere una versione più grande di questa figura.
In molti casi, è importante iniziare ampliare l'elenco delle varianti di includere tutti quelli in alta linkage disequilibrium (LD) con ciascuna variante statisticamente associato. LD è una misura di associazione non casuale di alleli in due differenti posizioni cromosomiche, che può essere misurata dal r 2 statistica 7. r 2 è una misura del linKage squilibrio fra due varianti, con una R 2 = 1 denota perfetto collegamento tra due varianti. Alleli in alta LD si trovano a co-separare sul cromosoma attraverso popolazioni ancestrali. array di genotipizzazione attuali non includono tutte le varianti conosciute nel genoma umano. Invece, sfruttano il LD all'interno del genoma umano e comprendono un sottoinsieme delle varianti note che agiscono come proxy per altre varianti di una particolare regione del LD 8. Così, una variante senza alcuna conseguenza biologica può essere associato ad una particolare malattia perché è in LD con causale variant-variante con un effetto biologico significativo. Procedurale, si consiglia di convertire l'ultima versione dei 1.000 genomi Project 9 file di chiamata variante (VCF) in file binari compatibili con PLINK 10,11, uno strumento open-source per l'intera analisi di associazione genoma. In seguito, tutte le altre varianti genetiche con LD r 2> 0.8 con ogni ingresso va geneticariant può essere identificato come candidati. E 'importante usare la popolazione di riferimento appropriato per questo passo- esempio, se una variante è stato identificato nei soggetti di origine europea, i dati provenienti da soggetti di origini simili dovrebbero essere utilizzati per l'espansione LD.
espansione LD si traduce spesso in decine di varianti candidati, ed è probabile che solo una piccola frazione di queste contribuiscono alla malattia meccanismo. Spesso, è praticamente impossibile esaminare sperimentalmente ciascuna di queste varianti singolarmente. E 'quindi utile per sfruttare le migliaia di pubblicamente disponibili i set di dati di genomica funzionale come filtro per dare priorità alle varianti. Ad esempio, il consorzio ENCODE 12 ha eseguito migliaia di esperimenti di ChIP-Seq che descrivono il legame del TF e co-fattori, e segni istoni in una vasta gamma di contesti, insieme ai dati della cromatina di accessibilità da tecnologie come DNasi-ss 13, ATAC -seq 14, e FAIRE-ss 15. databAsi e server web, come il browser UCSC Genome 16, tabella di marcia Epigenomics 17, Blueprint Epigenome 18, Cistrome 19, e Remap 20 fornire libero accesso ai dati prodotti da questi e altre tecniche sperimentali in una vasta gamma di tipi e condizioni di cellule. Quando ci sono troppe varianti per esaminare sperimentalmente, questi dati possono essere utilizzati per dare la priorità quelli situati all'interno probabili regioni regolatorie in materia tipi di cellule e tessuti. Inoltre, nei casi in cui una variante è all'interno di un picco ChIP-seq per una proteina specifica, questi dati possono fornire cavi potenziali per il TF specifico (s) o cofattori cui legame potrebbe essere che interessano.
Successivamente, le varianti risultanti priorità sono proiettati sperimentale per validare proteina genotipo-dipendente predetto legame con EMSA 21,22. EMSA misura la variazione della migrazione del oligo su un gel TBE non riducente. oligo fluorescente è incubato con illisato nucleare e legame di fattori nucleari ritardare il movimento del oligo sul gel. In questo modo, oligo che ha legato più fattori nucleari presenterà come un segnale fluorescente forte alla scansione. In particolare, l'EMSA non richiede previsioni circa le proteine specifiche il cui legame saranno interessati.
Una volta che le varianti sono identificati che si trovano all'interno di regioni regolatorie previsti e sono in grado di fattori nucleari differenziale vincolanti, metodi computazionali sono impiegati per predire il TF specifico (s), il cui legame che potrebbero influenzare. Noi preferiamo usare CIS-BP 23,24, RegulomeDB 25, UNIProbe 26, e JASPAR 27. Una volta che il candidato TF sono identificate, queste previsioni possono essere specificamente testati utilizzando anticorpi contro questi TF (EMSA-supershifts e DAPA-western). Un EMSA-supershift comporta l'aggiunta di un anticorpo specifico TF al lisato nucleare e oligo. Un risultato positivo in un EMSA-supershift è represented come un ulteriore spostamento nella banda EMSA, o una perdita di banda (valutata in riferimento 28). Nel DAPA complementare, un duplex oligo 5'-biotinilato contenente la variante e il 20 paia di basi di accompagnamento nucleotidi sono incubati con lisato nucleare dal tipo di cellula in questione (s) per catturare eventuali fattori nucleari legame specifico gli oligonucleotidi. Il complesso fattore duplex-nucleare oligo è immobilizzato dalla streptavidina microsfere in una colonna magnetica. I fattori nucleari legati vengono raccolti direttamente tramite eluizione 29,48. previsioni vincolanti possono poi essere valutati da un Western blot utilizzando anticorpi specifici per la proteina. Nei casi in cui non ci sono previsioni evidenti, o anche molte previsioni, i eluizioni dalla variante di pull-down degli esperimenti DAPA possono essere inviati a un nucleo di proteomica per identificare TF candidati mediante spettrometria di massa, che possono in seguito essere convalidati utilizzando questi precedentemente descritte metodi.
Nel resto della articlE, viene fornito il protocollo dettagliato per EMSA e DAPA analisi delle varianti genetiche.
Protocol
Representative Results
Discussion
Nonostante i progressi nelle tecnologie di sequenziamento e genotipizzazione hanno notevolmente migliorato la nostra capacità di individuare le varianti genetiche associate alla malattia, la nostra capacità di comprendere i meccanismi funzionali colpite da queste varianti è in ritardo. Una fonte importante del problema è che molte varianti associate alla malattia sono situati in n on-regioni codificanti del genoma, che probabilmente incidere più difficile da prevedere meccanismi che controllano l'espressi…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children’s Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Materials
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1X) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1M) | Sigma | 646563 | Reducing agent |
| PMSF | Thermo Scientific | 36978 | Protease Inhibitor |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10X) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/mL of penicillin, 10,000 µg/mL of streptomycin, and 25 µg/mL of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25mM HEPES |
Riferimenti
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium–understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5′-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).

