Imitieren die Funktion von Signalproteinen: Gegen künstliches Signal Transduction Therapie
Summary
We present guidelines for developing synthetic ‘chemical transducers’ that can induce communication between naturally unrelated proteins. In addition, detailed protocols are presented for synthesizing and testing a specific ‘transducer’ that enables a growth factor to activate a detoxifying enzyme and consequently, to regulate the cleavage of an anticancer prodrug.
Abstract
Signal transduction pathways, which control the response of cells to various environmental signals, are mediated by the function of signaling proteins that interact with each other and activate one other with high specificity. Synthetic agents that mimic the function of these proteins might therefore be used to generate unnatural signal transduction steps and consequently, alter the cell’s function. We present guidelines for designing ‘chemical transducers’ that can induce artificial communication between native proteins. In addition, we present detailed protocols for synthesizing and testing a specific ‘transducer’, which can induce communication between two unrelated proteins: platelet-derived growth-factor (PDGF) and glutathione-S-transferase (GST). The way by which this unnatural PDGF-GST communication could be used to control the cleavage of an anticancer prodrug is also presented, indicating the potential for using such systems in ‘artificial signal transduction therapy’. This work is intended to facilitate developing additional ‘transducers’ of this class, which may be used to mediate intracellular protein-protein communication and consequently, to induce artificial cell signaling pathways.
Introduction
Signaltransduktionswege spielt eine bedeutende Rolle in praktisch allen zellulären Prozess und damit die Zelle rasch auf Umgebungssignale zu reagieren. 1 Diese Wege häufig durch die Bindung eines Signalmoleküls an einen extrazellulären Rezeptor ausgelöst werden, die in der Aktivierung von intrazellulären Enzymen führt. Verstärkung und Ausbreitung dieses Signals innerhalb der Zelle wird durch die Funktion vermittelt Proteine von Signalisierungs, die ein Netzwerk von Protein-Protein-Wechselwirkungen bilden, in denen Enzyme mit hoher Spezifität reversibel aktiviert werden. Da Dysregulation dieser Netze führt häufig zur Entwicklung von Krebs, hat es bei der Einrichtung "Signaltransduktion Therapie von Krebs" viel Interesse gewesen, 2 , wobei Medikamente wurden entwickelt , um bösartige Signalwege zu stören. Vor kurzem haben wir einen alternativen Ansatz zur Signaltransduktion Therapie, die beruht auf der Fähigkeit von Arzneimitteln zu erzeugen unnatürliche Signaltransduktionswege vorgeschlagen. <sup> 3 Insbesondere glauben wir, dass synthetische Wirkstoffe durch die Gestaltung, die die Funktion von Signalproteinen nachahmen, wäre es möglich , die Zelle die Funktion indirekt zu modulieren. Beispielsweise können diese künstlichen Netzwerken Proteinbiomarker ermöglichen Enzyme zu aktivieren, die Prodrugs abspalten. Alternativ könnten diese Signalprotein-Mimetika können unnatürliche Zelle Signalwege zu aktivieren, in therapeutischen Wirkungen zur Folge hat.
Um die Machbarkeit dieses Ansatzes zu demonstrieren, haben wir vor kurzem eine synthetische "chemische Wandler '4 , die durch Aktivierung von Glutathion-s-Transferase (GST), das von Blutplättchen abstammenden Wachstumsfaktor (PDGF) auszulösen , die Spaltung eines Antikrebs – Prodrug ermöglicht ist nicht seine natürlichen Bindungspartner. Die Struktur dieses "Wandler" besteht aus einem anti-PDGF-Aptamer-DNA, die mit einem bivalenten Inhibitor für GST modifiziert wird. Daher gehört diese synthetische Mittel auf eine Familie von Molekülen mit Bindungsstellenverschiedene Proteine, 5-7 wie chemische Induktoren der Dimerisierung (CIDs) 8-10 und auch an die Gruppe von Protein-Bindemittel auf Basis von Oligonukleotid-synthetisches Molekül – Konjugate. 11-21
Die allgemeinen Prinzipien der Konstruktion solcher Systeme zugrundeliegende Aufgabe wird hierin beschrieben und detaillierte Protokolle zum Synthetisieren und Testen der Funktion dieses "Wandler" mit herkömmlichen enzymatischen Assays bereitgestellt. Diese Arbeit soll zusätzlichen "Wandler" dieser Klasse zu erleichtern entwickeln, die verwendet werden können intrazelluläre Protein-Protein-Kommunikation zu vermitteln und folglich künstliche Zellsignalwege induzieren.
1 schematisch beschreibt die Arbeitsweise von synthetischen "chemische Sensoren" , die unnatürliche Protein-Protein – Kommunikation vermitteln kann. In dieser Darstellung eine "chemische Wandler", die für Prot synthetische Bindemittel integrierteins I und II (Bindemittel I und II), ermöglicht Protein II die katalytische Aktivität des Proteins auszulösen I, die nicht ihren natürlichen Bindungspartner ist. In Abwesenheit von Protein II bindet der Wandler die katalytische Stelle des Enzyms (Proteins I) und inhibiert seine Aktivität (Abbildung 1, Zustand II). Die Bindung des "Transducer" zum Protein II, jedoch fördert Wechselwirkungen zwischen Bindemittel I und der Oberfläche des Protein II (Abbildung 1, Zustand III), die als Ergebnis ihrer Affinität zu Protein I. reduziert, die effektive Konzentration des ' frei "Wandler in der Lösung verringert wird , was dazu führt zur Dissoziation des Wandlers-Protein – Komplex I und Reaktivierung von Protein I (Abbildung 1, Zustand iv). Zusammengenommen markieren diese Schritte drei Grundsätze für die Gestaltung effizienter "Wandler" zugrunde liegen: (1) ein "Wandler" sollte für jeden der Protein-Targets ein spezifisches Bindemittel haben, (2) die Interaktion between Bindemittel II und Protein sollte II als die Wechselwirkung zwischen Bindemittel I und Protein I, und (3) Bindemittel stärker I der Lage mit der Oberfläche des Proteins zu interagieren II sein müssen. Das letzte Prinzip erfordert nicht unbedingt, dass Bindemittel Ich hätte allein eine hohe Affinität und Selektivität gegenüber Protein II. Stattdessen wird es auf unserer jüngsten Studien basieren , die zeigten , daß ein Protein , ein synthetisches Molekül in die Nähe bringen wahrscheinlich Wechselwirkungen zwischen diesem Molekül und der Oberfläche des Proteins zu fördern. 19,22,23
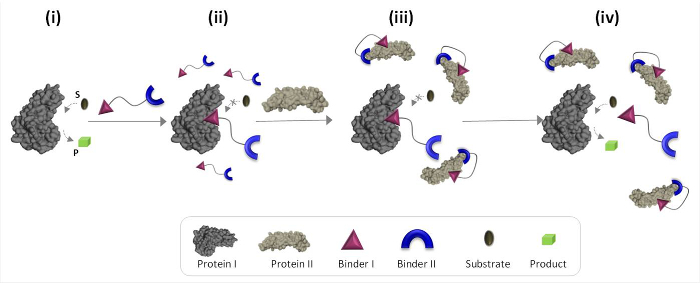
Abb . 1: Funktionsprinzipien von "chemischen Wandler" Wenn die "chemische Wandler" zu einem aktiven Protein hinzugefügt I (Zustand i), bindet es an seine aktive Stelle durch Bindemittel I und hemmt ihre Aktivität (Zustand ii). In Gegenwart von Protein II jedoch die ungebundene "chemische transducer 'in Wechselwirkung mit Protein II durch Bindemittel II, die II Wechselwirkungen zwischen Bindemittel I und der Oberfläche des Proteins fördert. Dieser induzierte Bindemittel I-Protein II Wechselwirkung reduziert die effektive Konzentration des Bindemittels I, die zur Dissoziation des 'transducer'-Protein führt I – Komplex und zur Protein Reaktivierung I (Stand iv). Bitte hier klicken um eine größere Version dieser Figur zu sehen .
Protocol
Representative Results
Discussion
We presented a method for designing and testing of a ‘chemical transducer’ that can induce artificial communication between two naturally unrelated proteins, GST and PDGF, without modifying the native proteins. The unnatural GST-PDGF communications could be detected in real time by using enzymatic assays that follow the changes in the activity of GST in the presence of the ‘chemical transducer’ and increasing the concentrations of PDGF. In addition to detecting the activation of GST by PDGF, these assays were used to fol…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Diese Arbeit wurde von der Minerva-Stiftung, der HFSP-Organisation und einen Europäischen Forschungsrat Grant (Starting Grant 338.265) unterstützt wurde.
Materials
| 1-chloro-2,4-dinitrobenzene | Sigma-Aldrich | 237329 | |
| Acetic acid | Bio Lab | 01070521 | |
| Acetnitrile | J.T.Baker | 9017-03 | |
| Ascorbic acid | Sigma-Aldrich | A4544 | |
| Copper(II) Sulfate pentahydrate | Merck-Millipore | 102790 | |
| Dimethyl sulfoxide | Merck-Millipore | 802912 | |
| Dulbecco's Phosphate Buffered Saline | Biological Industries | 02-023-5A | |
| Ethacrynic acid | Tokyo Chemical Industry Co. Ltd | E0526 | |
| Glutathione-s-transferase M1-1 | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| JS-K | Sigma-Aldrich | J4137 | |
| L-glutathione reduced | Sigma-Aldrich | G4251 | |
| Magnesium Chloride | J.T.Baker | 0162 | |
| nitrate/nitrite colorimetric assay kit | Cayman Chemical | 780001 | |
| Oligonucleotides | W. M. Keck Foundation Biotechnology at Yale University | custom order | |
| PDGF-BB | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| TBTA | Sigma-Aldrich | 678937 | |
| Triethylamine | Sigma-Aldrich | T0886 | |
| Desalting column | GE Healthcare | illustra MicroSpin G-25 Columns | |
| HPLC | Waters | 2695 separation module | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 4.6 mm × 50 mm) | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5μM, 10 mm × 50 mm) | |
| Plate reader | BioTek | synergy H4 hybrid |
Riferimenti
- Hunter, T. Signaling—2000 and Beyond. Cell. 100, 113-127 (2000).
- Levitzki, A., Klein, S. Signal transduction therapy of cancer. Mol Aspects Med. 31, 287-329 (2010).
- Peri-Naor, R., Motiei, L., Margulies, D. Artificial signal transduction therapy: a futuristic approach to disease treatment. Future Med. Chem. 7, 2091-2093 (2015).
- Peri-Naor, R., Ilani, T., Motiei, L., Margulies, D. Protein-Protein Communication and Enzyme Activation Mediated by a Synthetic Chemical Transducer. J. Am. Chem. Soc. 137, 9507-9510 (2015).
- Corson, T. W., Aberle, N., Crews, C. M. Design and Applications of Bifunctional Small Molecules: Why Two Heads Are Better Than One. ACS Chem. Biol. 3, 677-692 (2008).
- Rutkowska, A., Schultz, C. Protein Tango: The Toolbox to Capture Interacting Partners. Angew. Chem. Int. Ed. 51, 8166-8176 (2012).
- Meyer, C., Köhn, M. A Molecular Tête-à-Tête Arranged by a Designed Adaptor Protein. Angew. Chem. Int. Ed. 51, 8160-8162 (2012).
- Klemm, J. D., Schreiber, S. L., Crabtree, G. R. Dimerization as a Regulatory Mechanism in Signal Transduction. Annu. Rev. Immunol. 16, 569-592 (1998).
- DeRose, R., Miyamoto, T., Inoue, T. Manipulating signaling at will: chemically-inducible dimerization (CID) techniques resolve problems in cell biology. Pflugers Arch. 465, 409-417 (2013).
- Gestwicki, J. E., Marinec, P. S. Chemical control over protein-protein interactions: beyond inhibitors. Comb. Chem. High Throughput. Screen. 10, 667-675 (2007).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. 25, 848-862 (2013).
- Diezmann, F., Seitz, O. DNA-guided display of proteins and protein ligands for the interrogation of biology. Chem. Soc. Rev. 40, 5789-5801 (2011).
- Röglin, L., Ahmadian, M. R., Seitz, O. DNA-Controlled Reversible Switching of Peptide Conformation and Bioactivity. Angew. Chem. Int. Ed. 46, 2704-2707 (2007).
- Röglin, L., Altenbrunn, F., Seitz, O. DNA and RNA-Controlled Switching of Protein Kinase Activity. ChemBioChem. 10, 758-765 (2009).
- Harris, D. C., Chu, X., Jayawickramarajah, J. DNA-Small Molecule Chimera with Responsive Protein-Binding Ability. J. Am. Chem. Soc. 130, 14950-14951 (2008).
- Harris, D. C., Saks, B. R., Jayawickramarajah, J. Protein-Binding Molecular Switches via Host-Guest Stabilized DNA Hairpins. J. Am. Chem. Soc. 133, 7676-7679 (2011).
- Kim, Y., Cao, Z., Tan, W. Molecular assembly for high-performance bivalent nucleic acid inhibitor. Proc. Nat. Acad. Sci. U.S.A. 105, 5664-5669 (2008).
- Han, D., et al. A Logical Molecular Circuit for Programmable and Autonomous Regulation of Protein Activity Using DNA Aptamer-Protein Interactions. J. Am. Chem. Soc. 134, 20797-20804 (2012).
- Motiei, L., Pode, Z., Koganitsky, A., Margulies, D. Targeted Protein Surface Sensors as a Tool for Analyzing Small Populations of Proteins in Biological Mixtures. Angew. Chem. Int. Ed. 53, 9289-9293 (2014).
- Ranallo, S., Rossetti, M., Plaxco, K. W., Vallée-Bélisle, A., Ricci, F. A Modular, DNA-Based Beacon for Single-Step Fluorescence Detection of Antibodies and Other Proteins. Angew. Chem. Int. Ed. 54, 13214-13218 (2015).
- Franzini, R. M., et al. Identification of Structure-Activity Relationships from Screening a Structurally Compact DNA-Encoded Chemical Library. Angew. Chem. Int. Ed. 54, 3927-3931 (2015).
- Unger-Angel, L., et al. Protein recognition by bivalent, ‘turn-on’ fluorescent molecular probes. Chem. Sci. 6, 5419-5425 (2015).
- Nissinkorn, Y., et al. Sensing Protein Surfaces with Targeted Fluorescent Receptors. Chem. Eur. J. 21, 15981-15987 (2015).
- Huber, C. G., Oefner, P. J., Bonn, G. K. High-Resolution Liquid Chromatography of Oligonucleotides on Nonporous Alkylated Styrene-Divinylbenzene Copolymers. Anal. Biochem. 212, 351-358 (1993).
- Lyon, R. P., Hill, J. J., Atkins, W. M. Novel class of bivalent glutathione S-transferase inhibitors. Biochimica. 42, 10418-10428 (2003).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. , 1-16 (2013).

