מחק את תפקודם של חלבוני איתות: לקראת תרפים התמר איתותים מלאכותיות
Summary
We present guidelines for developing synthetic ‘chemical transducers’ that can induce communication between naturally unrelated proteins. In addition, detailed protocols are presented for synthesizing and testing a specific ‘transducer’ that enables a growth factor to activate a detoxifying enzyme and consequently, to regulate the cleavage of an anticancer prodrug.
Abstract
Signal transduction pathways, which control the response of cells to various environmental signals, are mediated by the function of signaling proteins that interact with each other and activate one other with high specificity. Synthetic agents that mimic the function of these proteins might therefore be used to generate unnatural signal transduction steps and consequently, alter the cell’s function. We present guidelines for designing ‘chemical transducers’ that can induce artificial communication between native proteins. In addition, we present detailed protocols for synthesizing and testing a specific ‘transducer’, which can induce communication between two unrelated proteins: platelet-derived growth-factor (PDGF) and glutathione-S-transferase (GST). The way by which this unnatural PDGF-GST communication could be used to control the cleavage of an anticancer prodrug is also presented, indicating the potential for using such systems in ‘artificial signal transduction therapy’. This work is intended to facilitate developing additional ‘transducers’ of this class, which may be used to mediate intracellular protein-protein communication and consequently, to induce artificial cell signaling pathways.
Introduction
מסלולי העברת אותות לשחק תפקיד משמעותי כמעט בכל תהליך הסלולר המאפשרים לתא להגיב לאותות סביבתיים במהירות. 1 מסלולים אלה הם בדרך כלל מופעלים על ידי הקשירה של מולקולת איתות קולטן תאי, שתוצאתה הפעלה של אנזימים תאיים. הגברת התפשטות של האיתותים בתוך התא מתווכת על ידי הפונקציה של איתות חלבונים יוצרים רשת של אינטראקציות בין חלבונים שבו אנזימים מופעלים הפיך עם סגוליות גבוהות. בגלל חוסר ויסות של רשתות אלה לעיתים קרובות מוביל להתפתחות סרטן, חלה התעניינות רבה בהקמת 'טיפול הולכת אותות של סרטן', 2 לפיה תרופות נועדו לשבש מסלולי איתות ממאיר. הצענו לאחרונה גישה חלופית לאותת טיפול תמרה המסתמך על היכולת של תרופות כדי ליצור מסלולי העברת אותות טבעיים. <sup> 3 בפרט, אנו מאמינים כי על ידי עיצוב סוכנים סינטטיים המחקים את תפקודם של חלבוני איתות, אפשר יהיה לווסת את הפונקציה של התא בעקיפין. לדוגמא, רשתות המלאכותיות אלה עשויות לאפשר סמנים ביולוגיים חלבון להפעיל אנזימים לבקע prodrugs. לחלופין, mimetics חלבון איתות אלה יוכל להפעיל מסלולי איתות תאי טבעי, וכתוצאה מכך השפעות טיפוליות.
כדי להוכיח את ההיתכנות של גישה זו, יצרנו לאחרונה 'מתמר כימי "סינתטי 4 המאפשר גורם גדילה הנגזרות טסיות (PDGF) כדי לעורר את המחשוף של prodrug נגד סרטן על ידי הפעלת גלוטתיון-s-transferase (GST), אשר הוא לא שותף המחייב טבעי. המבנה של "מתמר" זה מורכב aptamer DNA אנטי PDGF כי הוא שונה עם מעכב הדו-ערכי עבור GST. לפיכך, סוכן סינתטי זה שייך למשפחה של מולקולות עם אתרי הקישור כדיחלבונים שונים, 5-7 כגון מעוררים כימיים של dimerization (מספרי CID) 8-10 וגם לקבוצת חלבון-קלסרים מבוססת על conjugates מולקולה סינטטי oligonucleotide. 11-21
העקרונות הכלליים שבבסיס העיצוב של מערכות כאלה מתוארים במסמך זה ו פרוטוקולים מפורטים סינתזה ובדיקת התפקוד של זה 'מתמר' עם מבחני האנזימטית קונבנציונליים מסופקים. עבודה זו נועדה להקל על פיתוח 'מתמרים' נוסף של מחלקה זו, אשר עשוי לשמש לתווך תקשורת בין חלבונים תאיים וכתוצאה מכך, לגרום מסלולי איתות תאים מלאכותיים.
איור 1 סכמטי המתאר את עקרונות ההפעלה של הסינתטי "מתמר כימי 'שיכול לתווך תקשורת בין חלבונים טבעיים. באיור זה, א 'מתמר כימית', משלבת קלסרים סינטטי protEins I ו- II (קלסרים I ו- II), מאפשרים חלבון השני כדי לעורר את הפעילות הקטליטית של חלבון לי, שהוא לא שותף המחייב טבעי. בהיעדר חלבון II, מתמר נקשר לאתר הקטליטית של האנזים (אני חלבון) ומעכב את פעילותו (איור 1, המדינה השנייה). הכריכה של "מתמר" לחלבון השני, לעומת זאת, מקדם אינטראקציות בין לי קלסר ועל פני השטח של חלבון II (איור 1, מדינת iii), אשר מפחית זיקה כלפי I. חלבון כתוצאה מכך, הוא הריכוז היעיל של ' מתמר חינם 'בפתרון הצטמצם, מה שמוביל דיסוציאציה של החלבון מתמר שאני מורכב וכדי מחדש של חלבון אני (איור 1, מדינת iv). יחדיו, את הפעולות הבאות להדגיש שלושה עקרונות יסוד שביסוד העיצוב של 'מתמר' יעיל: (1) "מתמר" צריך קלסר ספציפי עבור כל אחת מן מטרות החלבון, (2) betwe האינטראקציהen קלסר השני והחלבון שני צריכים להיות חזק יותר את האינטראקציה בין לי קלסר והחלבון לי, ו- (3) קלסר אני חייב להיות מסוגל לקיים אינטראקציה עם פני השטח של חלבון II. העיקרון אחרון זה לא בהכרח דורש קלסר שאני לבד תהיה זיקה גבוהה סלקטיביות לכיוון החלבון השני. במקום זאת, היא מבוססת על המחקרים עדכניים שלנו שהראו כי הבאת מולקולה סינטתית בסמיכות חלבון עשויה לקדם אינטראקציות בין מולקולה זו ועל פני השטח של החלבון. 19,22,23
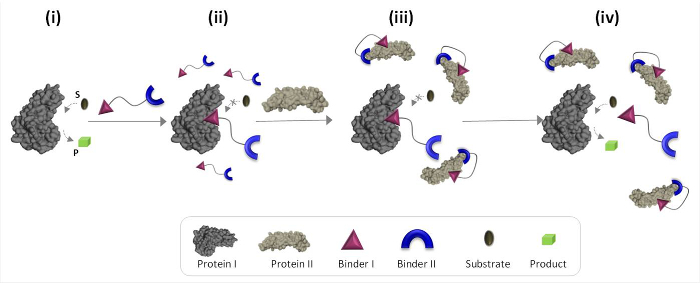
איור 1:. הפעלת עקרונות 'מתמר כימי' כאשר 'מתמר הכימית' מתווסף חלבון פעיל אני (מדינת i), הוא נקשר לאתר הפעיל שלה באמצעות קלסר לי ומעכב את פעילותו (המדינה השנייה). בנוכחות החלבון השני, לעומת זאת, לא כרוך לא כימיתransducer 'אינטראקציה עם חלבון II באמצעות קלסר השנייה, המקדמת אינטראקציות בין לי קלסר ועל פני השטח של חלבון II. קלסר המושרה זה I-חלבון II אינטראקציה מפחית את ריכוז יעיל של קלסר לי, מה שמוביל דיסוציאציה של שנות ה-חלבון transducer' שאני מורכבים לחלבון שאני מחדש (המדינה iv). נא ללחוץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו .
Protocol
Representative Results
Discussion
We presented a method for designing and testing of a ‘chemical transducer’ that can induce artificial communication between two naturally unrelated proteins, GST and PDGF, without modifying the native proteins. The unnatural GST-PDGF communications could be detected in real time by using enzymatic assays that follow the changes in the activity of GST in the presence of the ‘chemical transducer’ and increasing the concentrations of PDGF. In addition to detecting the activation of GST by PDGF, these assays were used to fol…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
מחקר זה מומן על ידי קרן מינרווה, ארגון HFSP, ומענק המועצה האירופית למחקר (החל גרנט 338,265).
Materials
| 1-chloro-2,4-dinitrobenzene | Sigma-Aldrich | 237329 | |
| Acetic acid | Bio Lab | 01070521 | |
| Acetnitrile | J.T.Baker | 9017-03 | |
| Ascorbic acid | Sigma-Aldrich | A4544 | |
| Copper(II) Sulfate pentahydrate | Merck-Millipore | 102790 | |
| Dimethyl sulfoxide | Merck-Millipore | 802912 | |
| Dulbecco's Phosphate Buffered Saline | Biological Industries | 02-023-5A | |
| Ethacrynic acid | Tokyo Chemical Industry Co. Ltd | E0526 | |
| Glutathione-s-transferase M1-1 | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| JS-K | Sigma-Aldrich | J4137 | |
| L-glutathione reduced | Sigma-Aldrich | G4251 | |
| Magnesium Chloride | J.T.Baker | 0162 | |
| nitrate/nitrite colorimetric assay kit | Cayman Chemical | 780001 | |
| Oligonucleotides | W. M. Keck Foundation Biotechnology at Yale University | custom order | |
| PDGF-BB | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| TBTA | Sigma-Aldrich | 678937 | |
| Triethylamine | Sigma-Aldrich | T0886 | |
| Desalting column | GE Healthcare | illustra MicroSpin G-25 Columns | |
| HPLC | Waters | 2695 separation module | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 4.6 mm × 50 mm) | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5μM, 10 mm × 50 mm) | |
| Plate reader | BioTek | synergy H4 hybrid |
Riferimenti
- Hunter, T. Signaling—2000 and Beyond. Cell. 100, 113-127 (2000).
- Levitzki, A., Klein, S. Signal transduction therapy of cancer. Mol Aspects Med. 31, 287-329 (2010).
- Peri-Naor, R., Motiei, L., Margulies, D. Artificial signal transduction therapy: a futuristic approach to disease treatment. Future Med. Chem. 7, 2091-2093 (2015).
- Peri-Naor, R., Ilani, T., Motiei, L., Margulies, D. Protein-Protein Communication and Enzyme Activation Mediated by a Synthetic Chemical Transducer. J. Am. Chem. Soc. 137, 9507-9510 (2015).
- Corson, T. W., Aberle, N., Crews, C. M. Design and Applications of Bifunctional Small Molecules: Why Two Heads Are Better Than One. ACS Chem. Biol. 3, 677-692 (2008).
- Rutkowska, A., Schultz, C. Protein Tango: The Toolbox to Capture Interacting Partners. Angew. Chem. Int. Ed. 51, 8166-8176 (2012).
- Meyer, C., Köhn, M. A Molecular Tête-à-Tête Arranged by a Designed Adaptor Protein. Angew. Chem. Int. Ed. 51, 8160-8162 (2012).
- Klemm, J. D., Schreiber, S. L., Crabtree, G. R. Dimerization as a Regulatory Mechanism in Signal Transduction. Annu. Rev. Immunol. 16, 569-592 (1998).
- DeRose, R., Miyamoto, T., Inoue, T. Manipulating signaling at will: chemically-inducible dimerization (CID) techniques resolve problems in cell biology. Pflugers Arch. 465, 409-417 (2013).
- Gestwicki, J. E., Marinec, P. S. Chemical control over protein-protein interactions: beyond inhibitors. Comb. Chem. High Throughput. Screen. 10, 667-675 (2007).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. 25, 848-862 (2013).
- Diezmann, F., Seitz, O. DNA-guided display of proteins and protein ligands for the interrogation of biology. Chem. Soc. Rev. 40, 5789-5801 (2011).
- Röglin, L., Ahmadian, M. R., Seitz, O. DNA-Controlled Reversible Switching of Peptide Conformation and Bioactivity. Angew. Chem. Int. Ed. 46, 2704-2707 (2007).
- Röglin, L., Altenbrunn, F., Seitz, O. DNA and RNA-Controlled Switching of Protein Kinase Activity. ChemBioChem. 10, 758-765 (2009).
- Harris, D. C., Chu, X., Jayawickramarajah, J. DNA-Small Molecule Chimera with Responsive Protein-Binding Ability. J. Am. Chem. Soc. 130, 14950-14951 (2008).
- Harris, D. C., Saks, B. R., Jayawickramarajah, J. Protein-Binding Molecular Switches via Host-Guest Stabilized DNA Hairpins. J. Am. Chem. Soc. 133, 7676-7679 (2011).
- Kim, Y., Cao, Z., Tan, W. Molecular assembly for high-performance bivalent nucleic acid inhibitor. Proc. Nat. Acad. Sci. U.S.A. 105, 5664-5669 (2008).
- Han, D., et al. A Logical Molecular Circuit for Programmable and Autonomous Regulation of Protein Activity Using DNA Aptamer-Protein Interactions. J. Am. Chem. Soc. 134, 20797-20804 (2012).
- Motiei, L., Pode, Z., Koganitsky, A., Margulies, D. Targeted Protein Surface Sensors as a Tool for Analyzing Small Populations of Proteins in Biological Mixtures. Angew. Chem. Int. Ed. 53, 9289-9293 (2014).
- Ranallo, S., Rossetti, M., Plaxco, K. W., Vallée-Bélisle, A., Ricci, F. A Modular, DNA-Based Beacon for Single-Step Fluorescence Detection of Antibodies and Other Proteins. Angew. Chem. Int. Ed. 54, 13214-13218 (2015).
- Franzini, R. M., et al. Identification of Structure-Activity Relationships from Screening a Structurally Compact DNA-Encoded Chemical Library. Angew. Chem. Int. Ed. 54, 3927-3931 (2015).
- Unger-Angel, L., et al. Protein recognition by bivalent, ‘turn-on’ fluorescent molecular probes. Chem. Sci. 6, 5419-5425 (2015).
- Nissinkorn, Y., et al. Sensing Protein Surfaces with Targeted Fluorescent Receptors. Chem. Eur. J. 21, 15981-15987 (2015).
- Huber, C. G., Oefner, P. J., Bonn, G. K. High-Resolution Liquid Chromatography of Oligonucleotides on Nonporous Alkylated Styrene-Divinylbenzene Copolymers. Anal. Biochem. 212, 351-358 (1993).
- Lyon, R. P., Hill, J. J., Atkins, W. M. Novel class of bivalent glutathione S-transferase inhibitors. Biochimica. 42, 10418-10428 (2003).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. , 1-16 (2013).

