Determinazione della Zeta Potenziale tramite nanoparticelle Traslocazione Le velocità attraverso una Tunable Nanopore: usando le particelle di DNA modificato come esempio
Summary
Qui usiamo un nanoporo sintonizzabile poliuretano integrato in una tecnica di rilevamento di impulsi resistivo caratterizzare nanoparticelle chimica di superficie tramite la misura della velocità di traslocazione delle particelle, che può essere utilizzato per determinare il potenziale zeta delle singole nanoparticelle.
Abstract
tecnologie nanoporo, noti collettivamente come sensori resistivi Pulse (RPS), vengono utilizzati per rilevare, quantificare e caratterizzare le proteine, molecole e nanoparticelle. sensing sintonizzabile impulso resistivo (TRPS) è relativamente recente adattamento di RPS che incorpora un poro sintonizzabile che può essere alterato in tempo reale. Qui, usiamo TRPS di monitorare i tempi di traslocazione di nanoparticelle DNA modificato, e attraversa la membrana pori sintonizzabile in funzione della concentrazione di DNA e struttura (cioè, a singolo filamento di DNA a doppio filamento).
TRPS si basa su due elettrodi Ag / AgCl, separate da una membrana elastomerica pori che stabilisce una corrente ionica stabile su un campo elettrico applicato. A differenza di varie tecnologie di caratterizzazione delle particelle ottiche a base, TRPS possono caratterizzare le singole particelle, tra un campione di popolazione, permettendo per i campioni multimodali da analizzare con facilità. Qui, dimostriamo misurazioni potenziale zetatramite velocità traslocazione delle particelle di standard noti e applicarle per assaggiare i tempi traslocazione analiti, con il risultato di misura del potenziale zeta di questi analiti.
Oltre ad acquisire valori di potenziale zeta medi, i campioni sono tutti misurati utilizzando una prospettiva particelle-by-particella esibendo ulteriori informazioni su un determinato campione attraverso distribuzioni di popolazione campione, per esempio. Di conseguenza, il metodo illustrato potenziale all'interno applicazioni di rilevamento per entrambi i campi medico e ambientale.
Introduction
nanoparticelle funzionalizzate stanno diventando sempre più popolare come biosensori in entrambi i campi medico e ambientale. La capacità di alterare la chimica di superficie di una nanoparticella, con il DNA, per esempio, si sta rivelando utile per i sistemi di somministrazione di farmaci mirati 1 e monitoraggio interazioni DNA-proteina 2-4. Una proprietà di nanoparticelle sempre più comune di essere utilizzato nel test biologici e nella consegna di terapie è superparamagnetismo 5. particelle superparamagnetiche (SPP) sono estremamente utili per identificare e rimuovere analiti specifici da miscele complesse e possono farlo con il semplice uso di un unico magnete. Una volta rimosse, le particelle di analiti-bound possono essere caratterizzate e analizzati adatto allo scopo.
I metodi precedenti utilizzati per la rilevazione e la caratterizzazione di nanoparticelle comprendono tecniche ottiche quali light scattering dinamico (DLS), altrimenti noti come la spettroscopia di correlazione di fotoni. Anche se un hitecnica di throughput gh, DLS si limita ad essere una tecnica basata compensazione e nell'analisi di campioni multimodali senza l'aggiunta di software specializzato, le particelle più grandi produrranno un segnale molto più dominante, lasciando alcune delle particelle più piccole completamente inosservato 6,7. Particle-by-particella tecniche di caratterizzazione sono quindi molto più favorevole per analizzare i sistemi di nanoparticelle funzionalizzati nanoparticelle e.
tecnologie basate RPS sono basati su applicazione di un campo elettrico per un campione e monitorare il meccanismo di trasporto delle particelle attraverso un nanoporo sintetico o biologico. Un relativamente recente tecnica di rilevazione e la caratterizzazione di nanoparticelle sulla base di RPS è sintonizzabile resistivo impulso di rilevamento (TRPS) 8-16. TRPS è un sistema a due elettrodi separati da una membrana elastomerica pori sintonizzabile. Metodo pori sintonizzabile permette di analiti di una serie di forma 17 e la dimensione deve essere misurata tramite il loro transmeccanismi di porta attraverso il poro. Pori sintonizzabili sono stati precedentemente utilizzati per la rilevazione di piccole particelle (70-95 nm di diametro) producendo risultati paragonabili a altre tecniche come la spettroscopia elettronica a trasmissione (TEM) 10. Quando un campo elettrico viene applicato, una corrente ionica viene osservata e come particelle / molecole passano attraverso il poro, essi bloccare temporaneamente il poro, provocando una riduzione della corrente che può essere definita come un 'evento blocco'. Ogni evento blocco è rappresentativo di una singola particella in modo che ogni particella di un campione può essere caratterizzato individualmente in base alla grandezza blocco, Δ 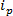 E larghezza a metà massimo FWHM, così come altre proprietà del blocco. Analizzando le singole particelle che passano attraverso un nanoporo è vantaggioso per i campioni multimodali come TRPS grado di distinguere correttamente ed efficacemente una gamma di dimensioni delle particelle amonGST un singolo campione. Sintonizzabile rilevamento impulso resistivo completa dimensione 10, potenziale zeta 12,18 e la concentrazione di 15 misurazioni simultaneamente in un unico passaggio e quindi può ancora distinguere campioni di simile, se non la stessa dimensione con la loro carica di superficie 19; un vantaggio rispetto alle tecniche dimensionali alternative.
E larghezza a metà massimo FWHM, così come altre proprietà del blocco. Analizzando le singole particelle che passano attraverso un nanoporo è vantaggioso per i campioni multimodali come TRPS grado di distinguere correttamente ed efficacemente una gamma di dimensioni delle particelle amonGST un singolo campione. Sintonizzabile rilevamento impulso resistivo completa dimensione 10, potenziale zeta 12,18 e la concentrazione di 15 misurazioni simultaneamente in un unico passaggio e quindi può ancora distinguere campioni di simile, se non la stessa dimensione con la loro carica di superficie 19; un vantaggio rispetto alle tecniche dimensionali alternative.
Potenziale Zeta è definito come il potenziale elettrostatico in corrispondenza del piano di taglio 20, e viene calcolato le velocità delle particelle, e attraversa un poro 19. Zeta potenziali misure delle singole particelle dà quindi informazioni sui meccanismi di traslocazione e il comportamento dei sistemi di nanoparticelle in soluzione, informazioni preziose per il futuro dei disegni di analisi nanoparticelle per una gamma di applicazioni. l'analisi delle particelle-by-particella di natura tale permette anche per la diffusione e la distribuzione dei valori potenziale zeta tra un campione di popolazione da esplorare, consentendo per ulteriori informazioni ocinetica di reazione n (single-stranded di DNA a doppia elica, per esempio) e stabilità di particelle in soluzione da raggiungere.
Qui, descriviamo una tecnica che individua e caratterizza entrambe le superfici modificate e DNA modificato spp. Il protocollo qui descritto è applicabile ad una gamma di nanoparticelle inorganiche e biologiche, ma dimostrano la procedura utilizzando superfici DNA modificato per la loro vasta gamma di applicazioni. La tecnica consente all'utente di distinguere tra obiettivi a filamento singolo e doppio filamento di DNA su una superficie di nanoparticelle, sulla base delle velocità di traslocazione particelle attraverso un sistema di pori e quindi i loro potenziali zeta.
Protocol
Representative Results
Discussion
Il calcolo per il potenziale zeta ha utilizzato un metodo basato calibrazione relativi al lavoro in Arjmandi et al. 21. La durata della traslocazione di particelle, e attraversa un nanoporo viene misurata in funzione della tensione applicata, utilizzando un campo e particella media velocità elettrici sulla totalità di un poro conica regolare. La mobilità elettroforetica è la derivata di 1 / T (dove T è la durata blocco) rispetto alla tensione, moltiplicata per il quadrato della lunghezza d…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Gli autori ringraziano Izon Science Ltd per il loro sostegno. Il lavoro è stato sostenuto dalla Commissione Europea per la Ricerca (PCIG11-GA-2012-321.836 Nano4Bio).
Materials
| Phosphate buffered Saline (PBS) | Sigma Aldrich, UK | P4417 | 1 tablet dissolved in 200 mL deionised water to make buffer solution. |
| Tween-20 | Sigma Aldrich, UK | P1379 | 0.05% (v/v) in PBS buffer as a surfactant |
| Carboxyl polystyrene nanoparticles | Bangs Laboratories, US | CPC200 | Nominal diamter of 220 nm, raw concentration of 1E12 particles/mL, specific surface charge of 86 µeq/g (equivalent to a surface charge density of 3.2E19 C/nm^2. |
| Streptavidin coated nanoparticles | Ademtech, France | 3121 | Batch had binding capacity of 4352 pmol/mg (188 nM theoretical DNA binding capacity) at a raw concentration of 1.1E11 particles/mL. |
| Biotinylated oligonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Biotin modification at 3' end; Lyophilised powders reconstituted to 100 µM using deionised water, and diluted as required. Sequences: CP 5'ATGGTTAAACCTCAC TACGCGTGGC[Btn]3' |
| Standard olignonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Lyophilised powders reconstituted to 100 µM using deionised water, and diluted as required. Sequences of DNA targets: Fully complementary – 5'GCCACGCGTAGTGAGGTTTAACCAT3', Middle binding – 5'GTAGTGAGGT3', End binding – 5'GTTTAACCAT3', Partially complementary overhanging – 5'GTGAGGTTTAACCAT TTTTTTTTTTTTTTT3'. |
| Izon qNano | Izon Science, NZ | Inherent pressure on system of 47 Pa, | |
| Izon Variable Pressure Module (VPM) | Izon Science, NZ | Each 'cm' of pressure is equivalent to approximately 1000 Pa. | |
| Polyurethane nanopore membranes | Izon Science, NZ | NP150 | Analyte size range 60-480 nm, pore diameter of calculated to be 799 nm at a 45 mm stretch. |
| Magrack 6 | GE Healthcare, UK | 28-9489-64 | |
| Sonic Bath | Fisher Scientific, UK | 10692353 | 80 Watts |
| Vortexer | IKA, Germany | 0003365000 | |
| Rotary Wheel | Labnet International, US | H5500-230 V |
Riferimenti
- Alexander, C. M., Maye, M. M., Dabrowiak, J. C. DNA-capped nanoparticles designed for doxorubicin drug delivery. Chem Commun. 47 (12), 3418-3420 (2011).
- Billinge, E. R., Platt, M. Aptamer based dispersion assay using tunable resistive pulse sensing (TRPS). Anal Methods. 7 (20), 8534-8538 (2015).
- Bulyk, M. L. Protein Binding Microarrays for the Characterization of Protein-DNA Interactions. Adv Biochem Eng Biotechnol. 104, 65-85 (2007).
- Platt, M., Rowe, W., Knowles, J., Day, P. J., Kell, D. B. Analysis of aptamer sequence activity relationships. Integr Biol. 1 (1), 116-122 (2009).
- Ruiz-Hernández, E., Baeza, A., Vallet-Regí, M. Smart Drug Delivery through DNA/Magnetic Nanoparticle Gates. ACS Nano. 5 (2), 1259-1266 (2011).
- Murdock, R. C., Braydich-stolle, L., Schrand, A. M., Schlager, J. J., Hussain, S. M. Characterization of Nanomaterial Dispersion in Solution Prior to In Vitro Exposure Using Dynamic Light Scattering Technique. Toxicol Sci. 101 (2), 239-253 (2008).
- Hupfield, S., Holsaeter, A. M., Skar, M., Frantzen, C. B., Brandl, M. Liposome size analysis by dynamic/static light scattering upon size exclusion-/field flow fractionation. J Nanosci Nanotechnol. 6 (7), 3025-3031 (2006).
- Roberts, G. S., et al. Tunable pores for measuring concentrations of synthetic and biological nanoparticle dispersions. Biosens Bioelectron. 31 (1), 17-25 (2012).
- Roberts, G. S., Kozak, D., Anderson, W., Broom, M. F., Vogel, R., Trau, M. Tunable nano/micropores for particle detection and discrimination: scanning ion occlusion spectroscopy. Small. 6 (23), 2653-2658 (2010).
- Vogel, R., et al. Quantitative sizing of nano/microparticles with a tunable elastomeric pore sensor. Anal Chem. 83 (9), 3499-3506 (2011).
- Booth, M. A., Vogel, R., Curran, J. M., Harbison, S., Travas-Sejdic, J. Detection of target-probe oligonucleotide hybridization using synthetic nanopore resistive pulse sensing. Biosens Bioelectron. 45, 136-140 (2013).
- Kozak, D., Anderson, W., Vogel, R., Chen, S. Simultaneous size and ζ-potential measurements of individual nanoparticles in dispersion using size-tunable pore sensors. ACS Nano. 6 (8), 6990-6997 (2012).
- Kozak, D., Anderson, W., Vogel, R., Trau, M. Advances in Resistive Pulse Sensors: Devices bridging the void between molecular and microscopic detection. Nano Today. 6 (5), 531-545 (2011).
- Weatherall, E., Willmott, G. R. Applications of tunable resistive pulse sensing. Analyst. 140, 3318-3334 (2015).
- Willmott, G. R., et al. Use of tunable nanopore blockade rates to investigate colloidal dispersions. J Phys Condens Matter. 22 (45), 454116 (2010).
- Blundell, E. L. C. J., Mayne, L. J., Billinge, E. R., Platt, M. Emergence of tunable resistive pulse sensing as a biosensor. Anal Methods. 7, 7055-7066 (2015).
- Platt, M., Willmott, G. R., Lee, G. U. Resistive Pulse Sensing of Analyte-Induced Multicomponent Rod Aggregation Using Tunable Pores. Small. 8 (15), 2436-2444 (2012).
- Vogel, R., Anderson, W., Eldridge, J., Glossop, B., Willmott, G. A variable pressure method for characterizing nanoparticle surface charge using pore sensors. Anal Chem. 84 (7), 3125-3131 (2012).
- Blundell, E. L. C. J., Vogel, R., Platt, M. Particle-by-Particle Charge Analysis of DNA-Modified Nanoparticles Using Tunable Resistive Pulse Sensing. Langmuir. 32 (4), (2016).
- Hunter, R. J. . Zeta Potential in Colloid Science: Principles and Applications. , (1981).
- Arjmandi, N., Van Roy, W., Lagae, L., Borghs, G. Measuring the electric charge and zeta potential of nanometer-sized objects using pyramidal-shaped nanopores. Anal Chem. 84 (20), 8490-8496 (2012).
- Bacri, L., et al. Dynamics of colloids in single solid-state nanopores. J Phys Chem B. 115 (12), 2890-2898 (2011).
- Cabello-Aguilar, S., et al. Dynamics of polymer nanoparticles through a single artificial nanopore with a high-aspect-ratio. Soft Matter. 10 (42), 8413-8419 (2014).
- Billinge, E. R., Muzard, J., Platt, M. Tunable resistive pulse sensing as a tool to monitor analyte induced particle aggregation. Nanomater Nanosci. 1 (1), 11 (2013).
- Li, J., Fan, C., Pei, H., Shi, J., Huang, Q. Smart Drug Delivery Nanocarriers with Self-Assembled DNA Nanostructures. Adv Mater. 25 (32), 4386-4396 (2013).
- Billinge, E. R., Broom, M., Platt, M. Monitoring aptamer-protein interactions using tunable resistive pulse sensing. Anal Chem. 86 (2), 1030-1037 (2014).
- Gold, L., et al. Aptamer-Based Multiplexed Proteomic Technology for Biomarker Discovery. PLoS One. 5 (12), e15004 (2010).
- Park, S. -. J., Taton, T. A., Mirkin, C. A. Array-Based Electrical Detection of DNA with Nanoparticle Probes. Science. 295 (5559), 1503-1506 (2002).

