Système d'expression multigène médié par l'ARN T7 à ADN polymère induible, pMGX
Summary
Cette étude décrit des méthodes pour la co-expression médiée par T7 de plusieurs gènes à partir d'un seul plasmide dans Escherichia coli en utilisant le système de plasmide pMGX.
Abstract
La co-expression de protéines multiples est de plus en plus essentielle pour la biologie synthétique, l'étude des complexes protéines-protéines, et la caractérisation et l'atténuation des voies de biosynthèse. Dans ce manuscrit, on décrit l'utilisation d'un système hautement efficace pour la construction d'opérons synthétiques multigènes sous le contrôle d'une ARN polymérase T7 inducible. Ce système permet d'exprimer plusieurs gènes simultanément à partir d'un plasmide. Ici, un ensemble de quatre vecteurs apparentés, pMGX-A, pMGX-hisA, pMGX-K et pMGX-hisK, avec le marqueur sélectionnable par résistance à l'ampicilline ou à la kanamycine (A et K) et possédant ou n'ayant pas d'hexahistidine N-terminale Tag (son) est divulgué. Des protocoles détaillés pour la construction d'opérons synthétiques utilisant ce système vectoriel sont fournis avec les données correspondantes, montrant qu'un système à base de pMGX contenant cinq gènes peut être facilement construit et utilisé pour produire les cinq protéines codées dans Escherichia coli . Ce systèmeEm et le protocole permet aux chercheurs d'exprimer systématiquement des modules et des chemins multi-composants complexes dans E. coli .
Introduction
La co-expression de multiples protéines est de plus en plus essentielle, en particulier dans les applications de biologie synthétique, où plusieurs modules fonctionnels doivent être exprimés 1 ; Dans l'étude des complexes protéine-protéine, où l'expression et la fonction nécessitent souvent une co-expression 2 , 3 ; Et dans la caractérisation et l'exploitation des voies de biosynthèse, où chaque gène dans la voie doit être exprimé 4 , 5 , 6 , 7 , 8 . Un certain nombre de systèmes ont été développés pour la co-expression, en particulier dans l'organisme hôte Escherichia coli , le cheval de travail pour l'expression de protéines recombinantes en laboratoire 9 . Par exemple, de multiples plasmides avec différents marqueurs sélectionnables peuvent être utilisés pour exprimer des protéines individuelles en utilisant une multitude d'ex différents Vecteurs de pression 10 , 11 . Des systèmes plasmidiques simples pour une expression de protéines multiples ont utilisé soit des promoteurs multiples pour contrôler l'expression de chaque gène 10 , 12 ; Opérons synthétiques, où plusieurs gènes sont codés sur une seule transcription 2 , 13 ; Ou, dans certains cas, un seul gène codant pour un polypeptide qui est finalement transformé de façon protéolytique, donnant les protéines désirées d'intérêt 14 .
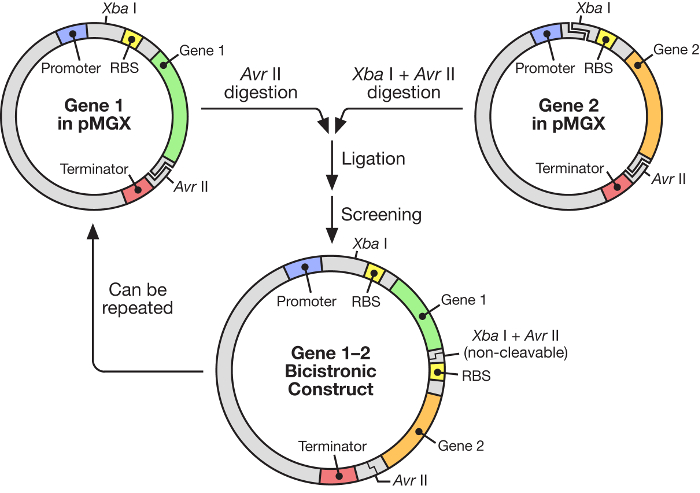
Figure 1: flux de travail pMGX montrant la construction d'un vecteur polycistronique. Le système pMGX offre une stratégie flexible et facile à utiliser pour la construction d'opérons synthétiques sous le contrôle d'un promoteur T7 inducible.E.com/files/ftp_upload/55187/55187fig1large.jpg "target =" _ blank "> Cliquez ici pour voir une version plus grande de cette figure.
Dans ce manuscrit, on décrit l'utilisation d'un système très efficace pour la construction d'opérons synthétiques multigène sous le contrôle d'une ARN polymérase T7 induite ( figure 1 ). Ce système permet d'exprimer plusieurs gènes simultanément à partir d'un plasmide. Il est basé sur un système plasmide, initialement appelé pKH22, qui a été utilisé avec succès pour un certain nombre d'applications différentes 6 , 7 , 8 . Ici, ce jeu de plasmide est développé pour inclure quatre vecteurs apparentés: pMGX-A, un vecteur d'expression dépourvu de marqueurs C ou N-terminaux et avec le marqueur de résistance à l'ampicilline; PMGX-hisA, un vecteur d'expression codant une étiquette d'hexahistidine N-terminal et avec le marqueur de résistance à l'ampicilline; PMGX-K, un vecteur d'expression lAvoir des marqueurs C ou N-terminaux et avec le marqueur de résistance à la kanamycine; Et pMGX-hisK, un vecteur d'expression codant une étiquette d'hexahistidine N-terminal et avec le marqueur de résistance à la kanamycine. Dans cette étude, le procédé de génération d'un vecteur polycistronique contenant cinq gènes utilisant le système pMGX, en particulier pMGX-A, est démontré avec la production réussie de chaque protéine individuelle dans Escherichia coli .
Protocol
Representative Results
Discussion
La co-expression de multiples gènes est de plus en plus essentielle, en particulier dans la caractérisation et la reconstitution des voies métaboliques multigène complexes 3 , 4 , 5 . Le système pMGX fait une co-expression multigène dans la routine E. coli 6 , 7 , 8 et accessible à divers chercheurs. Dans cette étude, …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Ce travail a été appuyé par le Conseil de recherches en sciences naturelles et en génie du Canada.
Materials
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50 x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm X 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box – PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
Riferimenti
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -. J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998 (2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp Available from: https://www.jove.com/science-education/5074/molecular-cloning (2016)
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp Available from: https://www.jove.com/science-education/5065/the-western-blot (2016)
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).

