Generering av fluorescerande protein Fusioner i<em> Candida</em> Arter
Summary
PCR-medierad gen modifiering kan användas för att generera fluorescerande proteinfusioner i Candida species, vilket underlättar visualisering och kvantifiering av jästceller och proteiner. Häri presenterar vi en strategi för att konstruera ett fluorescerande protein fusion (Eno1-FP) i Candida parapsilosis.
Abstract
Candida-arter, förhärskande kolonisatörer av tarm och urogenitala regionerna, är orsaken till de flesta av invasiva svampinfektioner hos människor. Således, är molekylära och genetiska verktyg som behövs för att underlätta studier av deras patogenes mekanismer. PCR-medierad gen modifiering är en enkel och snabb metod för att generera epitopmärkta proteiner för att underlätta deras detektering. I synnerhet fluorescerande protein (FP) -fusioner är kraftfulla verktyg som tillåter visualisering och kvantifiering av både jästceller och proteiner genom fluorescensmikroskopi och immunoblotting, respektive. Plasmider innehållande FP-kodande sekvenser, tillsammans med närings-markörgener som underlättar omvandlingen av Candida arter, har genererats i syfte att FP konstruktion och expression i Candida. Häri presenterar vi en strategi för att konstruera en FP fusion i en Candida-arter. Plasmider innehållande nourseothricin tåligiou transformation markörgen (NAT1) tillsammans med sekvenser för antingen grönt, gult eller körsbärs bps (GFP, YFP, mCherry) används tillsammans med primrar som inkluderar genspecifika sekvenser i en polymeraskedjereaktion (PCR) för att generera en FP kassett . Denna gen specifika kassetten har förmåga att integrera sig i 3'-änden av den motsvarande genlocus via homolog rekombination. Framgångsrik i-ram fusion av FP-sekvensen in i genlocus av intresse verifieras genetiskt, följt av analys av fusionsprotein-expression genom mikroskopi och / eller metoder immuno-detektion. Dessutom, för fallet med höggradigt uttryckta proteiner, framgångsrika fusioner kan screenas för i första hand genom fluorescensavbildningstekniker.
Introduction
Candida-arter är kommen svampar som koloniserar tarm och urogenitala skrifter av alla människor. Under förhållanden med nedsatt immunförsvar, såsom att inträffa med för tidig födsel eller immunsuppressiva effekter från behandlingar för cancer, kan Candida arter bli opportunistiska patogener. Av Candida arter, är Candida albicans den vanligaste svamp kolonisatör och orsakar majoriteten av invasiva svampinfektioner. Andra Candida arter såsom C. glabrata, C. parapsilosis, C. tropicalis och C. kruseii också orsaka allvarliga infektioner i patienter med nedsatt immunförsvar, med några uppvisar inneboende resistens mot vanligen använda anti-svamp antibiotika såsom flukonazol och amfotericin B. Därför infektioner med några av dessa arter följs oftare, särskilt hos patienter som behandlas profylaktiskt med antisvampmedel. Även med lämplig och snabb enNTI-svamp behandling, invasiva Candida infektioner fortsätter att förknippas med betydande morbiditet och mortalitet 1. På grund av betydelsen av Candida arter i människors hälsa, det finns ett behov av lättillgängliga molekylära verktyg som gör det möjligt att studera och klarlägga deras patogenes mekanismer.
Ett viktigt verktyg som gör det möjligt för forskarna att visualisera och kvantifiera mikrobiella celler och proteiner som de uttrycker är FP fusionsteknik. Polymeraskedjereaktion (PCR) -medierad genmodifiering, som beskrivs i detta dokument tillåter byggandet av fusioner mellan FP-sekvenser och en Candida proteinkodande sekvensen av intresse vid dess iska locus. Stabil integrering av konstruktionen underlättar analys av proteinuttryck samt proteinlokaliseringsdynamik. Plasmider innehållande FP-sekvenser, optimerad för uttryck i Candida albicans och som kan användas i PCR-medierad gene modifiering strategi, har tidigare konstruerats 2, 3, 4, 5. Plasmider innehåller FP transformations "kassetter": en FP-sekvens kopplad till en närings markörgen som underlättar omvandlingen av C. albicans och C. parapsilosis 2, 3, 4, 5, 6, 7. Närvarande tillgängliga plasmider innehåller en mängd olika selekterbara näringsmarkörgener (URA3, HIS1, ARG4) för transformation av auxotrofa stammar samt en dominerande läkemedelsresistensmarkör (NAT1), vilket underlättar omvandlingen av kliniska stammar som saknar auxotrophies. Dessutom plasmider innehåller alternativ för upp till fyra olika FP sekvenser (grön [GFP], yellow [YFP], cyan [GFP], och körsbär [mCherry]) och antingen en ADH1-termineringssekvens för konstruktion av karboxiterminus proteinfusioner, eller en promotorsekvens för konstruktion av amino-terminala proteinfusioner. Primrar är utformade med homologi med den plasmid-DNA som omger FP kassetten. Dessutom primers innehåller också 5'-förlängningssekvenser som bär homologi med jäst genen av intresse märkas, vilket underlättar integrering av kassetten i iska locus via homolog rekombination (figur 1). Genspecifika FP kassetter genereras med PCR och sedan omvandlas till Candida-celler som gjorts kompetenta för upptag av DNA genom behandling med litium-acetat.
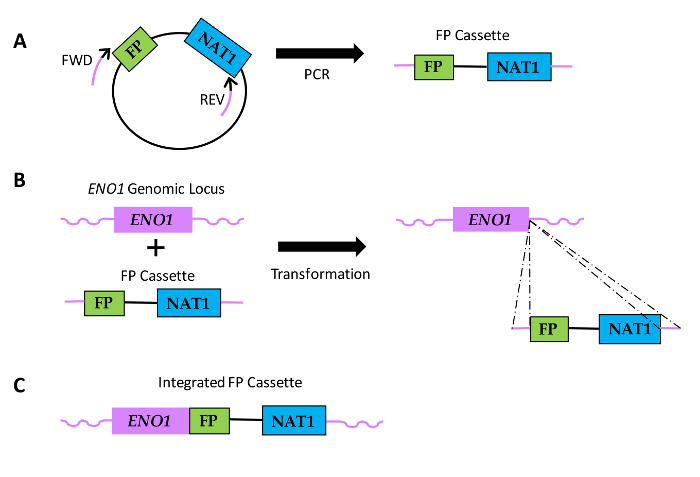
Figur 1: Diagram över hur FP sekvensfusioner genereras i Candida. (A) Plasmid-DNA includes en FP-sekvens och en sekvens som kodar nourseothricin resistens (NAT1). Relativa lägen i Forward (FWD) och bakåt (REV) primers visas med svarta delar av primers indikerar regionen av homologi med plasmiden sekvens och lila delarna betecknar den genspecifika homologi region eller primerförlängning. (B) FP kassetter förvandlas till Candida och integrera i ENO1 iska locus via homolog rekombination (streckade linjer). (C), vilket gav FP fusionssekvensen vid 3'-änden av ENO1. Klicka här för att se en större version av denna siffra.
Häri presenterar vi ett exempel på proteinfusion (Eno1-FP) konstruktioner i Candida. Vi använder märkning plasmider innehållande NAT1 omvandling markörgen tillsammans med sekvenser som kodar för GFP, YFP, ellermCherry (Figur 2). Dessa plasmider används tillsammans med primrar i PCR för att generera genspecifika kassetter som underlättar fusion av ramprogrammen till 3'-änden av ENO1, vilket resulterar i expression av Eno1 fuserad till FP vid dess karboxiterminus.
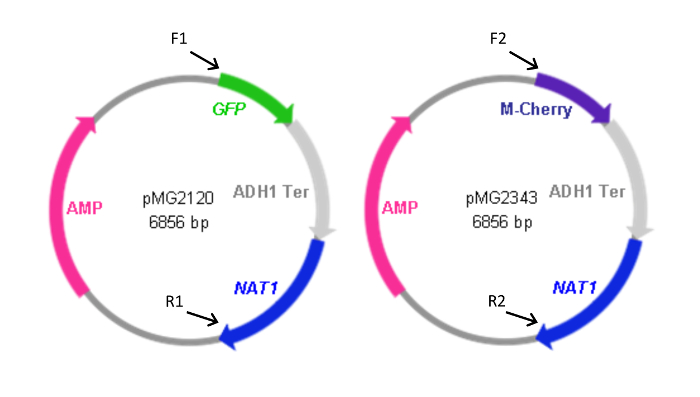
Figur 2: Kartor över FP kassettinnehållande plasmider. Framåt (F) och reverse (R) primrar som användes för att generera kassetterna från plasmiderna indikeras tillsammans med det relativa läget för deras homologi till plasmiderna. Primersekvenser är såsom anges i tabell 1. F1 och R1 användes också för att generera pYFP- NAT1 kassetten. Plasmiden innehållande YFP- NAT1 kassetten (pMG2263) är identisk med pMG2120 med undantag för YFP i stället för den GFP-sekvensen. Kassettstorlekar: GFP-NAT1, 3,7 kbp; mCherry- NAT1, 3,2 kbp; YFP- NAT1, 3.7 kbp. Denna siffra har modifierats Gerami-Nejad, et al. 4 Klicka här för att se en större version av denna siffra.
Protocol
Representative Results
Discussion
Konstruktion av epitopmärkt sekvenser i Candida-arter med hjälp av PCR-medierad genmodifiering strategi som beskrivs ovan kan sammanfattas som en process i tre steg. Först görs en kassett tillverkad genom PCR som kodar både den önskade för integrering och regioner homologa med den geometriska orten för insättning i jästgenomet sekvensen. För det andra är de jästceller som skall transforme göras kemiskt kompetenta med litiumacetat och saminkuberades med kassetten. Tredje cellerna ströks ut på sel…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Vi tackar N. Dean för att ge den ursprungliga mCherry FP sekvens, M. Gerami-Nejad för konstruktionen av plasmider, B. Larson för tekniskt stöd, och T. Heisel för råd under utvecklingen av detta projekt. JB stöddes av Europeiska forskningsrådet Advanced Award 340.087 (RAPLODAPT). Mikroskopi och bildsystem tillhandahölls av University of Minnesota Pediatrics Foundation och University of Minnesota Imaging Center.
Materials
| 100W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5ml, 1.5ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C ) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C ) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4°C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
Riferimenti
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).

