デジタル ホログラフィー顕微鏡 (DHM) を用いた低濃度で微生物の定量化
Summary
デジタル ホログラフィー顕微鏡法 (DHM) は、50 〜 100 倍焦点と明視野顕微鏡に匹敵する解像度よりも厚い実行画像サンプルを後処理により体積手法です。ここで DHM はカウントの識別に使用する、および低密度と光学密度計測、プレート カウント、直接カウントと比較して非常に微生物を追跡します。
Abstract
正確に検出し、まばらな細菌のサンプルを数える医療診断、他の惑星や太陽系の衛星にロボット ミッションによる生体検出、食品、飲料、医薬品加工業の多くのアプリケーションには。現在、文化のめっきや落射蛍光顕微鏡によるスパース細菌サンプルがカウントされます。培養皿を必要とする長いインキュベーション時間 (数日数週間から)、および広範な染色とサンプルの濃度、落射蛍光顕微鏡が必要です。ここでは、軸外デジタル ホログラフィー顕微鏡法 (DHM) を使用して、非常に希薄文化 (100 104セル/mL) 中の細菌を列挙する方法を示します。まず、カスタムの DHM の建設は、建物の低コスト計測器についての詳細と一緒に議論されています。ホログラフィーの原理は、説明、およびビデオにする必要がありますどのくらいの時間を推定する統計モデルを使用計測器の光学特性と (表 2) の細菌の溶液の濃度を基にセルを検出します。.10 セルのビデオ検出5104103、および 100 細胞/mL、非再建されたホログラムを使用してリアルタイムに示されて。振幅と位相像の再構成は、オープン ソースのソフトウェア パッケージを使用して示されています。
Introduction
非常に希薄サンプルで正確な細菌数の決定は多くのアプリケーションで重要である: 少数の例は水と食品品質解析1,2,3;血液、髄液、または喀痰4,5で病原体の検出滅菌水6; を含む医薬品の生産オープン海堆積物7,8,9などの低栄養環境で環境社会分析。木星や土星、特にヨーロッパ10、11エンケラドス12,13,の氷衛星における現存する微生物生命の検出に対する興味も高まっています14、地下の液体の海を持っている知られています。別の惑星で現存する生命を発見する 1978 年バイキング以来ミッションがないので、技術と細菌の同定と宇宙ミッション15時にカウントの楽器の開発を制限されています。
プレート カウントの伝統的な方法は、時々 環境系統種の少数を表すことができます、人為的セルのみを検索 < 116。プレートは、ひずみに応じて最大の成功のため数日あるいは数週間の培養が必要です。落射蛍光顕微鏡は、主として迅速かつ正確な生菌のためのゴールド スタンダードとしてプレート カウントを交換してください。核酸ラベリング酸蛍光染料 4 ‘, 6-diamidino-2-phenylindole 二塩酸塩 (DAPI)、SYBR グリーン アクリジン オレンジなど核酸に結合する、典型的な染料17,18,19、多くの研究を使用してグラムの蛍光インジケーター サイン20,21,22,23,24。1 mL あたり 10 ~5セルの検出 (Lod) の制限につながる前濃度ステップなしのこれらのメソッドを使用しています。LoD の改善がろ過を使用して可能です。液体試料は真空フィルター、膜、通常ポリカーボネートと理想的に黒背景を減らすために。低バック グラウンド染料のような上記の DNA 汚れはフィルター25に直接適用することがあります。正確な目でカウント、10 ~5セルがフィルターは、試料 1 mL あたり 10 ~5セルよりもさらに希釈を意味ごとに必要な重要なサンプル ボリュームを収集およびフィルタ リングする必要があります。レーザー スキャン デバイスは、フィルターのすべての領域を体系的に調査し、mL26あたり 10 ~2セルに至るまでの検出限界を迫り、カウントに必要なセルの数を減らすために開発されています。しかし、これらはほとんどの研究室で利用いただけません、高度なハードウェアだけでなくソフトウェア粒子を観察専門家の確認を許可するが細菌および残骸ではない必要があります。
参考のため敗血症大人通常開始で症状を示す < 100 細胞/mL の血とで乳児の < 10 セル/mL。大人からの血液は 10 mL と乳児、1 mL から。PCR 法が PCR 阻害成分血液27,28人間と非病原性植物 DNA の存在によって抑制されます。新興技術のさまざまな、にもかかわらず文化より農村地域や発展途上国を中心に血流感染症の診断のゴールド スタンダードのまま。他の惑星上の生命の検出、熱力学的計算は生命およびこうして予想可能なバイオマスのエネルギー予算を見積もることができます。1-100 細胞/mL は、ヨーロッパ29熱力学的に合理的であると予想されます。それは容易に水溶液の大量の細胞の非常に小さい数字の検出が重要な未解決問題であることこれらの数字から見ることができます。
本稿で示す 10 の濃度でセラチアとシェワネラ oneidensis (野生型および非運動性変異) の検出5104103、および軸オフを使用して 100 細胞/mLデジタル ホログラフィー顕微鏡 (DHM)。DHM の利点は従来の光学顕微鏡では高解像度での厚いサンプル ボリュームの同時イメージング-この実装では試料室は 0.8 mm 厚。これらのサンプル室に ± 50 μ m の公差の精密加工アルミ金型からポリジメチルシロキサン (PDMS) のソフト リソグラフィーにより建設されました。これは高倍率びきょわたってフィールドの深さの約 100 倍改善を表します。DHM はまた、光路長 (屈折率と膜厚の製品) の測定を可能にする定量的な位相情報を提供します。細菌や酵母の細胞周期および細菌乾燥質量30,31,32; の計算を監視するために使用されている DHM と類似の技術散乱の違いは、菌株33を区別するためにも使用する可能性があります。
使用楽器は具体的には以前に発行された34,35微生物で使用するため、その設計と建設を示したし、説明。水溶液は絶えずシリンジ ポンプを介して 0.25 μ L のボリュームのサンプル室に供給されます。流量は、サンプル全体のボリュームのイメージを確保するために、カメラのフレーム レートによって決まります。統計の計算は、特定の濃度で細胞のかなりの数を検出するためにイメージを作成する必要がありますサンプル ボリュームの数を予測します。
セル検出用ホログラムの振幅と位相の画像に再構築不要であった。生のホログラムの解析を行った。これは膨大な計算リソースとディスク領域を節約できます: 500 Mb のホログラム映像 1-2 Tb 再建されたときになります。ただし、深さ、ホログラムが希望の種を表すことを確認するためのサンプルの再構築に関する説明を行います。DHM の重要な機能は、強度と画像の位相の両方を監視する機能です。(ほとんどの生物細胞) など強度のほぼ透明な生物は、フェーズで明確に表示されます。ラベル無料テクニック、染料を使用していません。これは、可能な宇宙飛行用、染料はミッションの条件を生き残ることはできませんので dvantage と -さらに重要な-エンコードするため DNA または RNA を使用可能性があります地球外生物を操作するを想定できません。また、北極や南極、染料がリモートの場所に持参することは困難かもしれないし、収納時が低下するなど極端な環境での作業のための利点です。GitHub (シャンプー) を利用したオープン ソース ソフトウェア パッケージを使用してまたは ImageJ を用いた画像の位相と振幅に再構築が実行されます。
Protocol
Representative Results
Discussion
ホログラムの数値復元: ホログラムの数値復元、角度スペクトル法 (ASM) が使用されます。DHM のグリーン関数とホログラムの畳み込みが含まれます。特定焦点面での映像の複雑な波面は、次のようにフーリエ畳み込み定理を用いて計算できます。
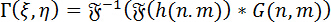 (1)
(1)
どこ<img alt="Equation 3" src="/files/ftp_upload/5634…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
著者は、この作業を資金調達のためカリフォルニア工科大学に 4038 とベティ ・ ムーア財団助成金 4037 ゴードンを認めます。
Materials
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
Riferimenti
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples – Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn’s moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).

