Indsamling og behandling af lymfeknuder fra store dyr til RNA analyse: forberede lymfeknude transkriptom undersøgelser af store dyrearter
Summary
Denne protokol giver et overblik over procedurerne til isolering af RNA for transkriptom profilering af lymfeknude væv fra store dyr, herunder trin i identifikation og excision af lymfeknuder fra husdyr og vilde dyr, prøveudtagning tilgange at give sammenhæng på tværs af flere dyr, og overvejelser samt repræsentative resultater for efter samling bevaring og forarbejdning til RNA analyse.
Abstract
Store dyr (husdyr og vilde dyr) tjener som vigtig reservoirer af zoonotiske patogener, herunder Brucella, Mycobacterium bovis, Salmonellaog E. coli, og er nyttig for studiet af patogenesen og/eller spredning af bakterier i naturlige værter. Med nøglefunktion i lymfeknuder i vært immunrespons tjene lymfeknude væv som en potentiel kilde til RNA for downstream transkriptom analyse, for at vurdere de tidsmæssige ændringer i genekspression i celler i løbet af en infektion. Denne artikel indeholder en oversigt over processen med lymfeknude samling, væv prøveudtagning og downstream RNA forarbejdning i husdyr, ved hjælp af kvæg (Bos taurus) som model, med yderligere eksempler hentet fra den amerikanske bison (Bison bison ). Protokollen indeholder oplysninger om placering, identifikation og fjernelse af lymfeknuder fra flere centrale steder i kroppen. Derudover præsenteres en biopsi prøveudtagning metodik der giver mulighed for en sammenhæng af prøveudtagning på tværs af flere dyr. Flere overvejelser for eksempel konservering er drøftet, herunder generation af RNA egnet til downstream metoder som RNA-sekventering og RT-PCR. På grund af de lange forsinkelser iboende i store dyr vs musen gang kursus undersøgelser, er repræsentative resultater fra bison og kvæg lymfeknude væv præsenteret for at beskrive tidsforløb nedbrydning i denne vævstype, i forbindelse med en gennemgang af tidligere metodologiske arbejde på RNA nedbrydning i andre væv. Samlet set vil denne protokol være nyttigt at begge veterinær Forskerne begynder transkriptom projekter på store dyr prøver og molekylær biologer interesseret i at lære teknikker i vivo væv prøveudtagnings-og in vitro- behandling.
Introduction
RNA-sekventering analyse af transkriptom af lymfeknuder giver mulighed for at karakterisere immunrespons i dyr til en bred vifte af patogener. Mens denne metode har været udnyttet flittigt i mus, har analyser for nylig ekspanderende til større pattedyr1,2. Husdyr/store dyr lymfeknuder kan bruges til at karakterisere vært svar til en infektion, ikke kun for deres anvendelse i vaccine eller genetiske modtagelighed undersøgelser og til identifikation af målene for udvikling af lægemidler, men også som modelsystemer for humane undersøgelser på zoonotiske sygdomme. For eksempel, i tilfælde af brucellose (en zoonotiske bakteriel sygdom at virkninger en halv million mennesker verden over hvert år), trods betydeligt øgede omkostninger, undersøgelser hos får eller geder er mere relevante for human infektion og menneskelige vaccine udvikling end laboratorium dyremodeller. Infektion musemodeller sammenfatte Retikuloendoteliale system infektion, men ikke den karakteristiske kliniske tegn3.
I store dyreforsøg i forhold til laboratoriet dyreforsøg indebærer processen med væv høst nødvendigvis en længere forsinkelse mellem eutanasi og samlingen væv, der udgør en potentiel udfordring for bevarelsen af høj kvalitet RNA. Intakt RNA er afgørende for generation af biologisk relevante transkriptom data. Generation af høj kvalitet RNA fra vævsprøver er særligt kritiske for store dyr patogen undersøgelser gennemført i indeslutningsfaciliteter. Sådanne undersøgelser er i sagens natur mere vanskeligt at udføre ikke blot kræver godkendt faciliteter og højt uddannede personale men også bære betydelige finansielle omkostninger, som afhængigt af arbejdet, kan variere fra titusinder til hundredtusinder af dollars. Disse typer af undersøgelser også omfatte et tæt tværfagligt samarbejde og tværfaglig viden for deres færdiggørelse, føje til deres kompleksitet. Derfor giver uddannelse, udvikling af og tilslutning til en strømlinet system for prøvetagning og bevarelse betydelige fordele for downstream molekylære studier af væv fra inficerede dyr.
Samling af større lymfeknuder præsenterer yderligere udfordringer for samlingen væv i forhold til lignende prøveudtagning af murine lymfeknuder. Forberedelsen af prøven excision nødvendiggør en grundlæggende forståelse af anatomien af lymfeknude, herunder de relevante interne strukturer. Strukturen i en lymfeknude består af lymfoide lobules omgivet af bihulerne fyldt med lymfeknuder. Disse strukturer er indkapslet i en hård, fibrøst kapsel. 4 en lymfoide lobule er “grundlæggende anatomiske og funktionelle enhed af lymfeknude” og er sammensat af hårsække, en dyb kortikale enhed, og medullær snore og bihuler4 (figur 1A). B og T lymfocytter er hjemsted for follikler og dybt kortikale enheder, henholdsvis. Disse strukturer giver en 3D stillads og lette samspillet mellem lymfocytter og antigen eller antigen præsentere celler.
Groft, follikler og dybt kortikale enheder kan identificeres på snitfladen da de indeholder en tættere retikulære meshwork og vises mørkere end bihuler, som består af en mere delikat retikulære meshwork og synes lysere (figur 1B). Af konventionen henviser patologer til områder af lymfeknuder som den overfladiske cortex (follikler), paracortex (dyb kortikale enheder) og medulla (medullær ledninger og bihulerne). En ordentlig undersøgelse af alle tre regioner har været anset som bedste praksis i rutinemæssig patologisk undersøgelse retningslinjer for lymfeknuder5. Bemærk at der er en betydelig variation i konsistensen, størrelse og farve af lymfeknuder, selv inden for et enkelt dyr. Som dyr alder, deres lymfeknuder tendens til at falde i størrelse og blive fastere end yngre dyr, typisk på grund af en stigning i deres bindevæv og en reduktion af den normale lymfoide strukturere6,7.
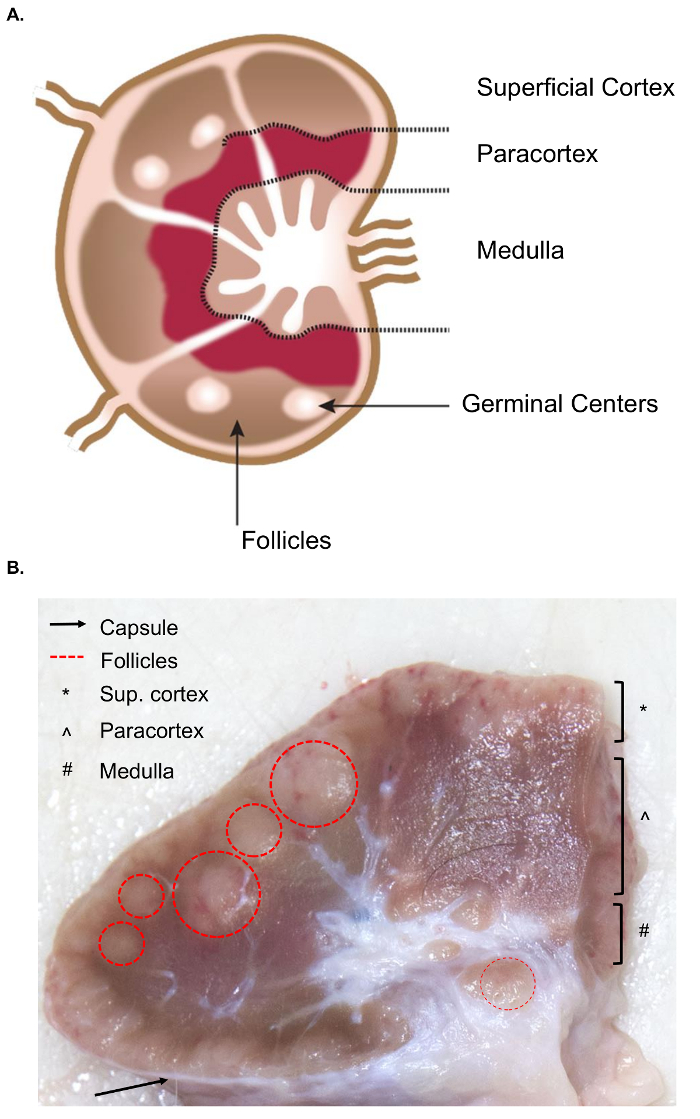
Figur 1. Anatomi af lymfeknude. (A) Denne tegneserie billede viser anatomien af lymfeknude, skildrer afgørende strukturer. (B) dette stadig billede viser en kvæg lymfeknude skåret i tværsnit. De relevante strukturer/lag, der er synlige for det blotte øje, er fremhævet. Venligst klik her for at se en større version af dette tal.
Afhængigt af den eksperimentelle spørgsmål, vil forskellige lymfeknuder være af interesse for indsamling og analyse. Perifere lymfeknuder er dem, der ligger dybt i det subkutane væv. I kvæg, perifere eller overfladiske lymfeknuder ofte anvendt i klinisk og eksperimentel praksis omfatter parotideale, submandibulære, retropharyngeale, bovlymfeknuden, glandel (precrural) og overfladiske lyskelymfeknuder (yverets i hunner, scrotalt hos mænd) () Figur 2). I tabel 1, er egenskaberne af centrale overfladiske lymfeknuder, som beskrevet i kvæg system8, skitseret. Nedenfor præsenteres nogle potentielle lymfeknude samling planer for smitsomme bakterielle sygdomme hos kvæg som udgangspunkt for undersøgelsen.
Brucella abortus/Brucella melitensis: Standard necropsies for B. abortus-inficeret kvæg og B. melitensis-inficerede geder på National Animal sygdom Center inddrive yverets, prescapular og parotideale lymfeknude væv , både til slibning til bakteriel tælling og for RNA forberedelse til vært RNA udtryk profilering. B. abortus kan inddrives regelmæssigt i hver af disse lymfeknuder i eksperimentelt inficeret kvæg9. Tilstedeværelsen af bakterier i hver af disse lymfeknude typer kan blive opdaget i B. melitensis-inficeret geder op til mindst ni måneder efter infektionen ved hjælp af de RNA-baserede metoder fra vores undersøgelser (Boggiatto et al., upubliceret). Salmonella Sp.: prescapular, subiliac (knæfoldslymfeknuderne), og mesenteriallymfeknuderne lymfeknuder har været nyttige i profilering af kvæg slagtekroppe for en Salmonella prævalens10,11,12 og ville være af potentiel interesse for transkriptom undersøgelser. E. coli O157: H7: mesenteriallymfeknuderne lymfeknuder (på den midterste tyndtarmen og distale tyndtarmen steder) kan være steder, hvor en lejlighedsvis inddrivelse af bakterier i inficerede kalve (men ikke i inficerede voksent kvæg)13. Leptospirose (Leptospira sp.): en kronisk persistens af bakterier er blevet observeret i lymfeknuderne dræning mælkekirtlen14. Mycobacterium bovis : I kvæg, er bakterier blevet genoprettet efter eksperimentel infektion fra lymfeknuder mediastinale og tracheobronchial kalve15. Derudover er lymfeknude RNA blevet udnyttet til at undersøge store dyr vært svar til vira, såsom porcint reproduktions- og respirationssyndrom virus2. Figur 2 viser placeringen af et undersæt af disse store lymfeknuder i selve kvæg.
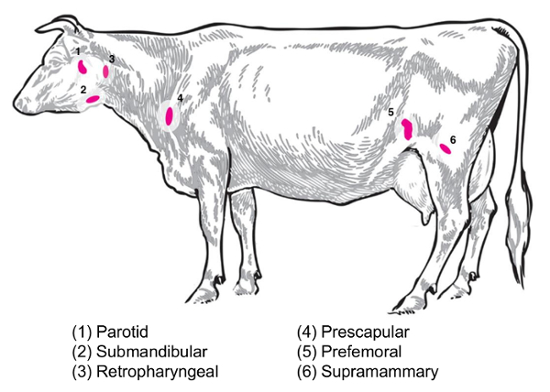
Figur 2: Tegneserien skildrer udvalgte lymfeknude steder i Bos taurus . De nummererede lymfeknuder er kommenteret. Venligst klik her for at se en større version af dette tal.
I dette papir og den tilhørende video præsenterer vi en protokol til isolering af store dyr lymfeknuder for RNA undersøgelser, designet til at være informative for Molekylær biologer involveret i transkriptom undersøgelser af store dyresygdomme. Først, vi giver et overblik over proceduren isolation for lymfeknuder, ved hjælp af stikprøver fra kvæg og bison væv som eksempler. Parret med denne demonstration, som vises i videoen, er en arbejdsproces for en reproducerbar væv stikprøveudtagning RNA isolering. Næste, vi beskriver vigtige overvejelser i forbindelse med behandling af en inficeret lymfeknude, med fokus på sikkerhed, konsistens og RNA kvalitet.
Forberedelse af RNA fra væv med et syrnet phenol-guanidin isothiocyanat reagens er baseret på den oprindelige metode til Chomczynski og Sacchi16,17, med en rensning over silica-baserede spin kolonner i chaotropic agenter baseret på det oprindelige arbejde af Vogelstein og Gillespie18. Vi undersøger også mulighederne for inddrivelse af RNA for transcriptomics fra kvæg lymfeknuder bevaret af alternative metoder. Endelig vil vi udforske virkningen af variablen tid på RNA kvalitet i stor animalsk necropsies, herunder en repræsentant eksperiment skildrer virkningen af en stigning i tid mellem eutanasi og prøveudtagning på den gendannede RNA profil fra bison og kvæg lymfeknuder. Denne artikel vil være nyttigt ikke kun til molekylærbiologer, men også til veterinær Forskerne begynder transkriptom undersøgelser.
Protocol
Representative Results
Discussion
Fleste af transkriptom undersøgelser og de tilknyttede protokoller fokusere på mus, rotte eller post mortem-menneskelige prøver. Men undersøgelser i husdyr og vilde dyr og giver en bred vifte af muligheder for karakterisering af immunrespons til sygdom, både som gælder for veterinærmedicin og med hensyn til zoonoser, offentlige sundhedsfare. Denne protokol leveret en oversigt over centrale overvejelser i forbindelse med høj-integritet RNA udvinding fra væv fra store dyr som kvæg, bison, geder og får. Størrels…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Forfatterne vil gerne takke James Fosse for hans fremragende arbejde på alle Videografi og video forarbejdning; Michael Marti for hans fremragende arbejde i generation af digitaliserede kvæg billeder; Lilia Walther for hendes hjælp med RNA udvinding og Bioanalyzer kører; Mitch Palmer og Carly Kanipe for deres nyttige anmeldelse og feedback på lymfeknude billeder; og pasning af dyr og veterinaere personale på National Animal sygdom Center for alle deres hårde arbejde og bistand med husdyrhold og forberedelse til necropsies.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
Riferimenti
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

