Verzameling en verwerking van lymfklieren uit grote dieren RNA p.a.: lymfeklier Transcriptomic Studies van grote diersoorten voorbereiden
Summary
Dit protocol biedt een overzicht van de procedures voor de isolatie van RNA voor de profilering van de transcriptomic van de lymfeklier weefsels van grote dieren, met inbegrip van de stappen in de identificatie en de besnijdenis van lymfeklieren van vee en wilde dieren, bemonstering benaderingen te leveren consistentie in meerdere dieren, en overwegingen plus representatieve resultaten voor de post collectie bewaring en verwerking ten behoeve van RNA analyse.
Abstract
Grote dieren (vee en wild) dienen als belangrijk reservoirs van zoönotische pathogenen, met inbegrip van Brucella, Mycobacterium bovis, Salmonellaen E. coli, en zijn nuttig voor de studie van de pathogenese en/of verspreiding van de bacteriën in natuurlijke gastheren. Met de belangrijkste functie van de lymfklieren in de immuunrespons van de gastheer dienen lymfeklier weefsels als een potentiële bron van RNA voor downstream transcriptomic analyses, teneinde de tijdelijke wijzigingen in genexpressie in cellen in de loop van een infectie. Dit artikel geeft een overzicht van het proces van de lymfeklier collectie, bemonstering van weefsel en downstream RNA verwerking in vee, van runderen (Bos taurus) als een model, met extra voorbeelden van de Amerikaanse bison (Bison bison ). Het protocol bevat informatie over de locatie, identificatie en verwijdering van lymfeklieren van meerdere belangrijke plaatsen in het lichaam. Bovendien wordt een biopsie bemonsteringsmethode gepresenteerd dat voorziet in een consistentie van bemonstering over meerdere dieren. Verscheidene overwegingen voor behoud van de steekproef worden besproken, met inbegrip van de generatie van RNA geschikt voor downstream methodologieën zoals RNA-sequencing en RT-PCR. Vanwege de lange vertragingen die inherent zijn aan grote dierlijke vs. muis tijd cursus studies, worden representatieve resultaten van bison en boviene lymfeklier weefsels voorgesteld om te beschrijven van het tijdsverloop van de degradatie in dit weefseltype, in het kader van een herziening van vorige methodologische werkzaamheden op aantasting van het RNA in andere weefsels. Over het geheel genomen zal dit protocol nuttig aan beide veterinaire onderzoekers beginnen transcriptome projecten op grote dieren monsters en moleculaire biologen geïnteresseerd in het leren van technieken voor in vivo weefsel bemonstering en in vitro verwerking zijn.
Introduction
RNA-sequencing analyse van transcriptome lymfklieren biedt de mogelijkheid om het karakteriseren van de immuunrespons van dieren aan een verscheidenheid van pathogenen. Terwijl deze methodologie is gebruikt uitgebreid in muizen, analyses hebben onlangs geweest zich uitbreiden naar grotere zoogdieren1,2. Dieren/grote dierlijke lymfeklieren kan worden gebruikt voor het karakteriseren van host reacties op een infectie, niet alleen voor hun gebruik in het vaccin of genetische studies en voor de identificatie van doelen voor Geneesmiddelenontwikkeling, maar ook als modelsystemen voor menselijke studies op zoönotische ziekten. Bijvoorbeeld in het geval van brucellose (een zoönotische bacteriële ziekte dat effecten een half miljoen mensen over de hele wereld elk jaar), ondanks aanzienlijk hogere kosten, studies in schapen of geiten meer relevant zijn voor de menselijke infectie en menselijk vaccin ontwikkeling dan laboratorium diermodellen. Infectie Muismodellen recapituleren de reticuloendotheliaal systeem infectie maar niet de karakteristieke klinische symptomen3.
In grote dierproeven ten opzichte van dierproeven laboratorium bestaat het proces van weefsel oogsten noodzakelijkerwijs uit een langere vertraging tussen de euthanasie en de weefsel-collectie, die een potentiële uitdaging voor het behoud van kwalitatief hoogwaardige RNA. Intact RNA is essentieel voor de generatie van biologisch relevante transcriptomic gegevens. De generatie van kwalitatief hoogwaardige RNA van weefselmonsters is bijzonder kritisch voor grote dieren pathogen studies uitgevoerd in insluiting faciliteiten. Dergelijke studies zijn inherent moeilijker uit te voeren zoals ze niet alleen goedgekeurde voorzieningen en hoogst opgeleid personeel vereisen, maar ook aanzienlijke financiële kosten, die, afhankelijk van het werk voeren, van tientallen tot honderden duizenden dollars variëren kunnen. Deze soorten studies ook betrekking hebben op een interdisciplinaire samenwerking en multidisciplinaire kennis ten behoeve van hun voltooiing, toe te voegen aan hun complexiteit. Daarom, training op, ontwikkeling van en aanhankelijkheid aan een gestroomlijnd systeem voor de sample collectie en het behoud biedt aanzienlijke voordelen voor downstream moleculaire studies van weefsels van besmette dieren.
De collectie van grotere lymfklieren presenteert extra uitdagingen voor de collectie van weefsel ten opzichte van de vergelijkbare bemonstering van lymfkliertest lymfklieren. De voorbereiding voor de steekproef besnijdenis vereist een basiskennis van de anatomie van de lymfeklier, met inbegrip van de relevante interne structuren. De structuur van een lymfeklier bestaat uit lymfoïde melkklieren omgeven door sinussen gevuld met lymfe. Deze structuren zijn ingesloten in een harde, vezelige capsule. 4 een lymfoïde lobule is de “fundamentele anatomische en functionele eenheid van de lymfeklier” en bestaat uit een diep corticale eenheid, en Wallenberg koorden, follikels en sinussen4 (figuur 1A). B en T-lymfocyten zijn de thuisbasis van de follikels en diep corticale eenheden, respectievelijk. Deze structuren bieden een 3D steiger en faciliteren van de interactie tussen de lymfocyten en antigeen of antigeen presentatie van cellen.
Grove, follikels en diep corticale eenheden kunnen worden geïdentificeerd op het snijvlak geconstateerd als ze een dichtere reticulaire gevlochten bevatten en donkerder dan de sinussen, die bestaan uit een meer delicate reticulaire gevlochten en lichter weergegeven (figuur 1B). Door conventie verwijzen pathologen naar de regio’s van de lymfeknopen zoals de oppervlakkige cortex (follikels), de paracortex (diep corticale eenheden) en de medulla (Wallenberg koorden en sinussen). Een nauwkeurig onderzoek van de drie gewesten werd geacht als beste praktijk routine pathologisch onderzoek richtsnoeren voor lymfklieren5. Merk op dat er een aanzienlijke variatie in de consistentie, grootte en kleur van de lymfeknopen, zelfs binnen een enkel dier. Naarmate de dieren ouder, hun lymfklieren zal meestal kleiner en worden steviger dan die van jongere dieren, meestal als gevolg van een toename van hun bindweefsel en een vermindering van de normale lymfoïde structuur6,7.
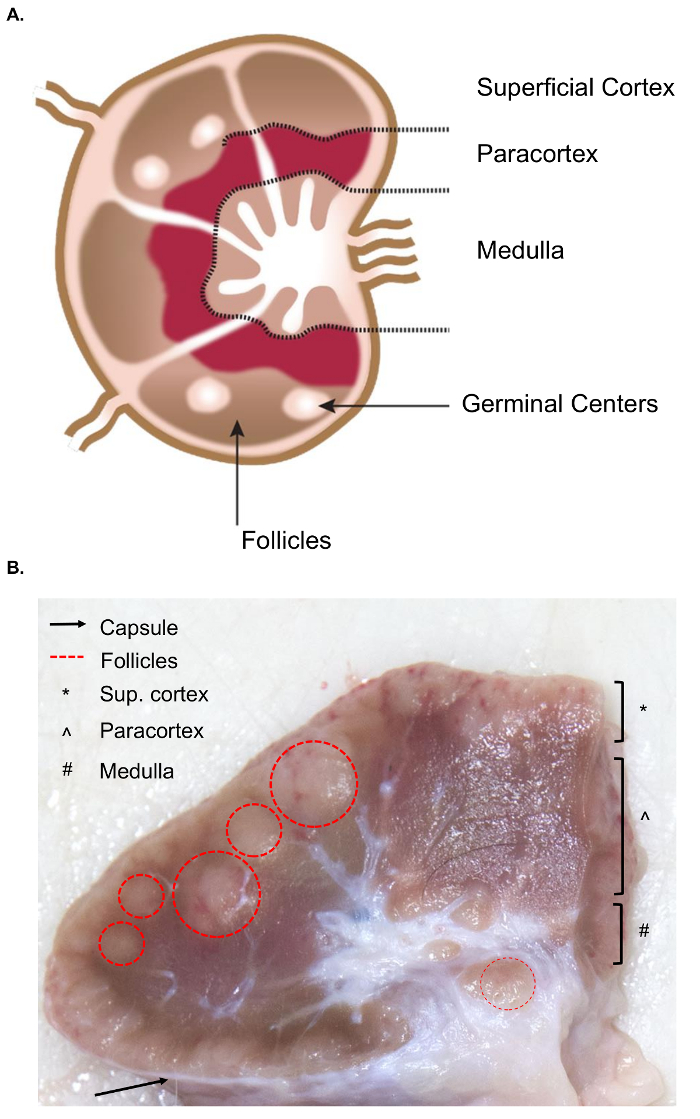
Figuur 1. Anatomie van de lymfeklier. (A) deze cartoon afbeelding toont de anatomie van de lymfeklier, beeltenis van belangrijke structuren. (B) dit nog beeld toont een boviene lymfeklier gesneden in doorsnede. De relevante structuren/lagen die zichtbaar met het blote oog zijn worden gemarkeerd. Klik hier voor een grotere versie van dit cijfer.
Afhankelijk van de experimentele vraag zullen verschillende lymfklieren van belang zijn voor het verzamelen en analyseren. Perifere lymfklieren zijn gelegen diep in het onderhuidse weefsel. Bij runderen, perifere of oppervlakkige lymfklieren vaak gebruikt in de klinische en experimentele praktijk omvatten parotide, submandibulaire retrofaryngeale, prescapulares, prefemoral (precrural) en de oppervlakkige inguïnale (uier worden bij vrouwtjes, scrotum bij mannen) () Figuur 2). In tabel 1, worden de eigenschappen van de belangrijkste oppervlakkige lymfklieren, zoals beschreven in de vee-systeem8, schetste. Hieronder worden enkele potentiële lymfeklier collectie plannen voor bacteriële infectieziekten van runderen gepresenteerd als een uitgangspunt voor het onderzoek.
Brucella abortus/Brucella melitensis: standaard necropsies voor B. abortus-besmet vee en B. melitensis-besmette geiten aan het National dier ziekte Center herstellen uier worden, prescapular en oorspeeksellymfklieren lymfeklier weefsel , zowel voor het slijpen voor de bacteriële opsomming alsmede voor de bereiding van het RNA voor de host RNA expressie profilering. B. abortus kunnen regelmatig worden hersteld in elk van deze lymfeklieren in experimenteel geïnfecteerde runderen9. De aanwezigheid van bacteriën in elk van deze typen lymfeklier kan worden opgespoord in B. melitensis-besmet geiten tot ten minste negen maanden na infectie met behulp van de methoden voor RNA gebaseerde uit onze studies (Boggiatto et al., onuitgegeven). Salmonella sp.: de prescapular, subiliac (prefemorale), en de mesenterische lymfklieren zinvol zijn geweest tijdens de profilering van runderkarkassen voor een Salmonella prevalentie10,11,12 en zou van potentieel belang voor transcriptomic studies. E. coli O157:H7: mesenterische lymfklieren (op de middelste dunne darm en distale dunne darm locaties) kunnen de sites van een occasionele herstel van de bacteriën in geïnfecteerde kalveren (maar niet in geïnfecteerde volwassen runderen)13. Leptospirose (Leptospira sp.): een chronische persistentie van de bacteriën is waargenomen in de lymfeknopen aftappen van de melkklier14. Mycobacterium bovis : Bij runderen, zijn de bacteriën herstelde na experimentele infectie van de lymfeknopen mediastinale en tracheobronchial van kalveren15geweest. Bovendien is lymfeklier RNA te onderzoeken dieren talloze reacties op virussen, zoals de varkens reproductieve en respiratoir syndroom virus2gebruikt. Figuur 2 toont de locatie van een subset van deze grote lymfeklieren in het lichaam van vee.
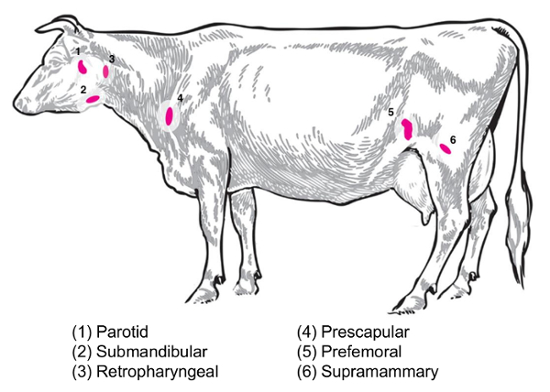
Figuur 2: Cartoon beeltenis van geselecteerde lymfeklier locaties in Bos taurus . De genummerde lymfeklieren zijn geannoteerd. Klik hier voor een grotere versie van dit cijfer.
In dit document en de bijbehorende video presenteren wij een protocol voor de isolatie van grote dieren lymfklieren voor RNA studies, ontworpen als informatief voor moleculaire biologen die betrokken zijn bij transcriptomic studies van grote dierlijke infecties. Eerst, wij bieden een overzicht van de isolatie-procedure voor de lymfeknopen, met behulp van bemonstering van weefsels van runderen en bison als voorbeelden. Gekoppeld aan deze demonstratie, zoals weergegeven in de video, is een werkstroom voor een reproduceerbare weefsel bemonstering voor RNA isolatie. Vervolgens beschrijven we belangrijke overwegingen voor de verwerking van een geïnfecteerde lymfe-knooppunt, met een focus op veiligheid, consistentie en kwaliteit van RNA.
De voorbereiding van RNA van het weefsel met een aangezuurde fenol-guanidine isothiocyanaat reagens is gebaseerd op de originele methode van Chomczynski en Sacchi16,17, met een zuivering over silica gebaseerde spin kolommen in aanwezigheid van chaotropic agenten op basis van het oorspronkelijke werk van Vogelstein en Gillespie18. We onderzoeken ook de mogelijkheden voor het herstel van RNA voor transcriptomics van vee lymfklieren geconserveerd door toepassing van alternatieve methoden. Ten slotte onderzoeken we de invloed van de variabele van de tijd op de kwaliteit van de RNA in grote dierlijke necropsies, met inbegrip van een representatieve experiment beeltenis van het effect van een toename van de tijd tussen de euthanasie en de bemonstering op de herstelde RNA-Profiel van bison en boviene lymfklieren. Dit artikel zal nuttig zijn niet alleen voor moleculaire biologen maar ook aan veterinaire onderzoekers vanaf transcriptomic studies zijn.
Protocol
Representative Results
Discussion
De meerderheid van de studies van de transcriptomic en de bijbehorende protocollen richten op muis, rat of post-mortemlaesies menselijke specimens. Onderzoeken in vee en wild bieden echter een breed scala van mogelijkheden voor de karakterisering van de immuunrespons tegen ziekten, zowel als toepassing op geneesmiddelen voor diergeneeskundig gebruik en, met betrekking tot de bestrijding van zoönotische ziekten, om de volksgezondheid. Dit protocol verstrekt een overzicht van de belangrijkste overwegingen bij hoge-integri…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
De auteurs wil James Fosse bedanken voor zijn uitstekende werk op alle videografie en video-verwerking; Michael Marti voor zijn uitstekende werk in de generatie van gedigitaliseerde vee beelden; Lilia Walther voor haar hulp bij RNA extractie en Bioanalyzer loopt; Mitch Palmer en Carly Kanipe voor hun nuttige beoordeling en feedback op lymfeklier beelden; verzorging van de dieren en de veterinaire personeel bij de nationale dier ziekte centrum voor al hun harde werk en bijstand met de veehouderij en de voorbereiding voor necropsies.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
Riferimenti
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

