Сбор и обработка лимфатических узлов от крупных животных для анализа РНК: подготовка исследований транскриптомики Лимфоузел крупных видов животных
Summary
Этот протокол обеспечивает обзор процедур для изоляции РНК для профилирования транскриптомики Лимфоузел тканей от крупных животных, включая шаги в процессе идентификации и иссечение лимфоузлов от скота и диких животных, выборки подходы чтобы обеспечить согласованность несколько животных и соображения плюс представитель результаты для сохранения после сбора и обработки для анализа РНК.
Abstract
Крупные животные (скота и диких животных) служат в качестве важных водохранилищ зоонозными патогенами, включая бруцеллы, Mycobacterium bovis, сальмонеллаи кишечная палочкаи являются полезными для изучения патогенеза и/или распространение бактерий в естественных хостов. С помощью ключевых функции лимфатических узлов в иммунного ответа тканей лимфатических узлов служат потенциальным источником РНК для вниз по течению транскриптомики анализа, для того чтобы оценить временные изменения в экспрессии генов в клетках в течение инфекции. Эта статья содержит обзор процесса лимфатических узлов коллекции, ткани проб и ниже по течению РНК переработки скота, используя крупного рогатого скота (Bos taurus) в качестве модели, с дополнительные примеры из американских бизонов (Бизон, Бизон ). Протокол включает в себя сведения о местонахождении, идентификации и удаления лимфатических узлов из нескольких ключевых узлов в организме. Кроме того методология отбора проб биопсии представлена который позволяет для соответствия выборки через несколько животных. Обсуждаются несколько соображений охраны образцов, включая поколения РНК подходит для вниз по течению методологии как РНК последовательности и RT-PCR. Вследствие длительных задержек, присущих крупных животных против мыши время курса исследований представитель от зубров и говядину Лимфоузел тканей результаты для описания время курс деградации в этом типе ткани, в контексте обзора предыдущие методологической работы по деградации РНК в других тканях. В целом этот протокол будет полезным для обоих ветеринарных исследователей, начиная с транскриптом проекты на больших животных образцы и молекулярные биологи заинтересованы в изучении методов для обработки в пробирке и в естественных условиях выборки ткани.
Introduction
РНК последовательности анализа транскриптом лимфатических узлов обеспечивает возможность характеризовать иммунная реакция животных на целый ряд патогенов. Хотя эта методология широко использовались в мышей, анализ недавно расширяется в больших млекопитающих1,2. Скот и крупных животных лимфатические узлы могут использоваться для характеризуют разместить ответы на инфекции, не только для их использования в вакцины или генетической предрасположенности исследований, так и для определения целевых показателей для разработки лекарственных средств, но и как модель системы для человека исследования на зоонозных заболеваний. Например в случае бруцеллеза (зоонозных бактериальных заболеваний, что воздействие полмиллиона людей во всем мире каждый год), несмотря на значительно увеличить расходы, исследования в овец или коз более актуальны для инфицирования человека и человеческой вакцины развития, чем лабораторные животные модели. Модели мыши инфекции пилки ретикулоэндотелиальной системы инфекции, но не характерных клинических признаков3.
В крупных животных эксперименты по сравнению с лабораторных исследований на животных процесс ткани, уборки обязательно включает в себя более длительную задержку между эвтаназии и ткани коллекции, которая представляет собой потенциальный вызов для сохранения высокое качество РНК. Нетронутыми РНК имеет важное значение для поколения биологически соответствующих транскриптомики данных. Поколения высокого качества РНК из образцов ткани особенно важно для крупных животных патогенов исследований проводится в изолирующего оснащения. Такие исследования являются по своей сути сложнее выполнять, поскольку они не только требуют утверждения зал и высоко квалифицированного персонала, но также осуществляем значительные финансовые расходы, которые, в зависимости от работы, может варьироваться от нескольких десятков до сотен тысяч долларов. Эти типы исследований также включать кросс дисциплинарного сотрудничества и кросс дисциплинарного знания для их завершения, добавляя к их сложности. Таким образом, обучение, развитие и приверженность рациональной системы для сбора и сохранения обеспечивает значительные преимущества течению молекулярных исследований тканей от зараженных животных.
В коллекции более крупных лимфатических узлов представлены дополнительные проблемы для коллекции тканей, по сравнению с аналогичными выборки мышиных лимфатических узлов. Подготовка образца иссечение требует базового понимания анатомии лимфатических узлов, включая соответствующих внутренних структур. Структуры лимфатического узла состоит из лимфоидных долек, окруженный пазухи заполнены с лимфы. Эти структуры заключаются в жесткой, фиброзной капсулы. 4 лимфоидных долька является «основной анатомической и функциональной единицей лимфатических узлов» и состоит из фолликулов, глубоко корковых подразделение и Медуллярного шнуры и пазухах4 (рис. 1A). Лимфоцитов T и B являются домом для фолликулов и глубоко корковых единиц, соответственно. Эти структуры обеспечивают 3D леску и облегчить взаимодействие между лимфоцитов и антиген или антиген представляющих клеток.
Грубо, фолликулы и глубоко корковые блоки могут быть определены на поверхности среза как они содержат более плотной сети ретикулярные и темнее чем придаточных пазух носа, которые состоят из более тонкие ретикулярных сеть и светлее (рис. 1B). Конвенцией патологов относятся к области лимфатических узлов как поверхностные коры (фолликулов), paracortex (глубоко корковых единиц) и продолговатого мозга (Медуллярного шнуры и придаточных пазух носа). Надлежащее рассмотрение всех трех регионов был признан как лучший практика в обычной патологических экспертизе лимфатические узлы5. Обратите внимание, что существуют значительные различия в последовательности, размер и цвет лимфатических узлов, даже в пределах одного животного. Как возраст животных их лимфатических узлов, как правило, будет уменьшаться в размерах и становятся тверже, чем у молодых животных, обычно за счет увеличения их соединительной ткани и сокращение нормальных лимфоидных структуры6,7.
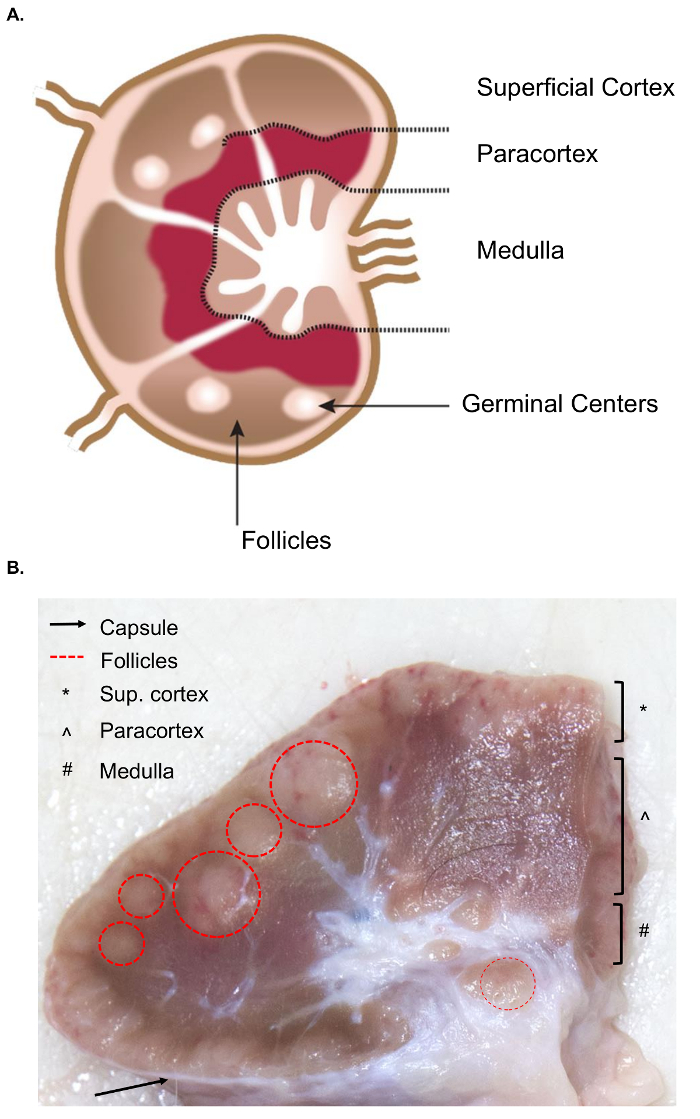
Рис. 1. Анатомия лимфатического узла. (A) это изображение мультфильм показывает анатомию лимфоузлов, изображающие ключевых структур. (B) это еще изображение показывает бычий лимфоузлов, сократить в поперечном сечении. Будут выделены соответствующие структуры/слои, которые видны невооруженным глазом. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
В зависимости от экспериментальных вопрос различные лимфатические узлы будут представлять интерес для сбора и анализа. Периферические лимфатические узлы являются те расположен глубоко в подкожной клетчатке. Крупного рогатого скота, периферийных устройств или поверхностные лимфатические узлы часто используются в клинической и экспериментальной практике включают околоушной, подчелюстные, заглоточный, предлопаточные, предбедренные (precrural) и поверхностные паховые (supramammary у самок, мошонки у самцов) () Рисунок 2). В таблице 1изложены свойства ключевых поверхностных лимфатических узлов, как описано в8системы крупного рогатого скота. Ниже представлены некоторые потенциальные лимфатических узлов коллекции планы инфекционных бактериальных болезней крупного рогатого скота как отправной точки для проведения расследования.
Бруцеллы абортуса/бруцеллы melitensis: стандарт necropsies для B. абортуса-инфицированных скот и B. melitensis-инфицированных коз в национальном центре болезни животных восстановить supramammary, prescapular и околоушной лимфатический узел ткани , как для измельчения для бактериальных перечисления, а также для подготовки РНК для профилирования выражение принимающей РНК. B. абортуса может восстанавливаться регулярно в каждом из этих лимфатических узлов в экспериментально зараженных скота9. Присутствие бактерий в каждом из этих типов лимфатических узлов может быть обнаружен в B. melitensis-инфицированных коз до по меньшей мере девять месяцев послеоперационные инфекции с использованием методологии на базе РНК из нашего исследования (Boggiatto et al., неопубликованные). Сальмонелла SP.: prescapular, лимфатического (предбедренные), и брыжеечных лимфатические узлы были полезны во время профилирования туш крупного рогатого скота для сальмонеллы распространенности10,и11,12 и бы потенциальный интерес для транскриптомики исследования. Кишечной палочки O157: H7: брыжеечных лимфатические узлы (в среднем тонкой кишки и дистальной части тонкой кишки местах) могут быть сайты время от времени восстановления бактерий в зараженных телят (но не в инфицированного взрослого скота)13. Лептоспироз (Лептоспире sp.): хронический сохранение бактерий наблюдается в лимфатических узлах, осушение молочной железы14. Mycobacterium bovis : Крупного рогатого скота бактерии были восстановленные после экспериментальной инфекции от средостения и Трахеобронхиальные лимфоузлы телят15. Кроме того лимфоузлов РНК была использована для изучения крупных животных принимающих ответы вирусов, таких как вирус свиного репродуктивного и респираторных синдром2. Рисунок 2 изображает расположение подмножество этих крупных лимфатических узлов в организме скота.
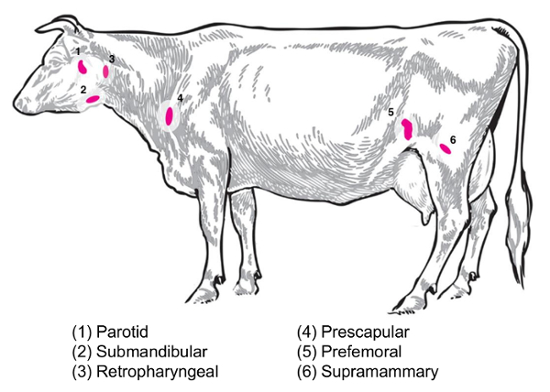
Рисунок 2: Мультфильм с изображением выбранной Лимфоузел мест в Bos taurus . Аннотированная пронумерованных лимфатические узлы. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
В этом документе и связанные видео мы представляем собой протокол для изоляции крупных лимфатических узлов животных РНК исследований, разработан, чтобы быть информативным для молекулярные биологи, участвующих в исследованиях транскриптомики крупных животных инфекций. Во-первых мы предоставляем обзор процедуры изоляции лимфатических узлов, используя выборки из тканей говядину и зубров в качестве примеров. В паре с этой демонстрации, как показано в видео, — это рабочий процесс для воспроизводимых ткани выборки для изоляции РНК. Далее мы описывают важные соображения для обработки зараженных лимфатических узлов, с уделением особого внимания безопасности, согласованности и качества РНК.
Подготовка РНК из ткани с Изотиоцианаты реактивом подкисленных фенол Гуанидин основывается на оригинальный метод Chomczynski и Сакки16,17, с очисткой столбцов на основе силики спина в присутствии chaotropic агенты, основанный на оригинальной работой Фогельштейна и Гиллеспи18. Мы также изучить возможности для восстановления РНК для transcriptomics от скота лимфатических узлов, консервированные в альтернативных методов. Наконец, мы исследуем влияние переменной среды на качество РНК в крупных животных necropsies, включая представителя эксперимент с изображением эффект увеличения времени между эвтаназии и выборки на восстановленные профиль РНК из зубров и говядину лимфатические узлы. Эта статья будет полезна не только молекулярные биологи, но также для ветеринарных исследователей, начиная с исследования транскриптомики.
Protocol
Representative Results
Discussion
Большинство транскриптомики исследований и связанных с ними протоколов сосредоточиться на мыши, крысы или post-mortem образцах. Однако исследования на домашний скот и диких животных предоставляют широкий спектр возможностей для характеризации иммунной реакции на болезни, как применимые ?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Авторы хотели бы поблагодарить Джеймса Fosse за его отличную работу на всех видеосъемка и видео обработка; Майкл Marti за его прекрасную работу в генерации изображения оцифрованных скота; Lilia Walther за ее помощь с РНК добыча и Bioanalyzer бежит; Митч Палмер и Карли Канип за их полезный обзор и обратной связи на изображениях лимфатических узлов; и уход за животными и ветеринарных сотрудников национального центра болезни животных для всех их напряженную работу и помощь с животноводством и подготовки necropsies.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
Riferimenti
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

