Korrelat ljus elektronmikroskopi (CLEM) för att spåra och Imaging virala Protein associerade strukturer i Cryo-orörlig celler
Summary
En korrelat ljus elektronmikroskopi (CLEM) metod tillämpas på bild virus-inducerad intracellulära strukturer via elektronmikroskopi (EM) i celler som tidigare valts av ljusmikroskop (LM). LM och EM kombineras som en hybrid imaging strategi att uppnå en integrerad vy av virus-host interaktioner.
Abstract
På grund av dess hög upplösning är elektronmikroskopi (EM) ett oumbärligt verktyg för virologer. En av de största svårigheterna när du analyserar virus-infekterade eller transfekterade celler via EM är dock låg effektivitetsvinsterna av infektion eller transfection, hindrar undersökningen av dessa celler. För att övervinna denna svårighet, kan ljusmikroskopi (LM) utföras först för att allokera subpopulationen av infekterade eller transfekterade celler. Således att dra nytta av användningen av fluorescerande proteiner (FPs) smält till virusproteiner, LM används här att registrera positionerna för ”positiv-transfekterade” cellerna, uttrycker en FP och växer på ett stöd med ett alfanumeriskt mönster. Därefter celler bearbetas ytterligare för EM via högtryck frysning (HPF), frysa substitution (FS) och harts inbäddning. Det Ultra snabb frysning steget säkerställer utmärkt membran bevarandet av markerade celler som sedan kan analyseras på ultrastrukturella nivå av transmissionselektronmikroskopi (TEM). Här tillhandahålls en stegvisa korrelat ljus elektronmikroskopi (CLEM)-arbetsflödet, som beskriver provpreparering, imaging och korrelation i detalj. Experimentell design kan också användas för att åtgärda många cellbiologi frågor.
Introduction
Idén att kombinera två mikroskopi modaliteter för att få en bättre bild av en specifik biologisk process är ganska gammal. Således publicerades den första studien om virus använder ”korrelat mikroskopi” 1960 som två separata publikationer1,2. I denna studie analyseras författarna förändringarna i morfologi av kärnan inducerad av adenoviruses med hjälp av två tekniker som mikroskopi. I den första publikationen var elektronmikroskopi (EM) observationer som beskriver de morfologiska detaljer i samband med adenovirus infektion rapporterade1. I en andra publikation, var olika strukturer observeras av EM korrelerade med ljusmikroskop (LM) bilder av histochemical färgning mönster, att definiera arten av de strukturer som tidigare observerats av EM2.
I dessa tidiga studier, men utfördes deras observationer med olika infekterade celler förberedda som oberoende experiment. ”Sambandet”, faktiskt, betyddes som kombinationen av information som kommer från två avbildningsmetoder att förstå ett visst fenomen, jämföra alla de resultat som har erhållits med olika analyser för att förstå en viss biologisk processen.
Numera appliceras termen korrelat microscopyen, även känd som korrelat ljus- och elektronmikroskopi (CLEM), på ett ökande antal metoder (över referenser3,4,5), med Gemensamheten att båda avbildningstekniker (LM och EM) utförs på samma prov. Kombinationen av båda metoderna resultat, därmed, i en multimodal, flerskalig och flerdimensionell analys av att prov3. Fördelarna är att LM kan ge en bred översikt av många olika celler, möjliggör identifiering av cell subpopulations uttrycker ett protein eller proteiner av intresse inom en heterogen cell befolkning. EM övervinner resolution gränsen för LM, vilket ger en högre upplösning bild av en viss intracellulära händelse. Dessutom möjliggör EM visualisering av icke-fluorescerande subcellulär ramen, inklusive alla membran bunden organeller, stora makromolekylärt komplex (t.ex. ribosomer, centrioles, etc.) och cytoskeletal element, därmed att tillhandahålla ytterligare rumsliga information, den så kallade ”referens utrymme”6, och ge sammanhang till fluorescerande plats upptäcks av LM.
Under de senaste åren, CLEM har blivit ett kraftfullt verktyg inte bara för cell biologer5, men också för virologer (ses i referens7) villig att förstå de komplexa virus-cell interaktioner som leder till en framgångsrik virus förökning. Thus, förstå hur virus ändra cellmembran och organeller till egen vinning är nödvändigt att utveckla antivirala läkemedel för att utrota lågpatogena virus.
Här, beskrivs en CLEM metod som gör upptäckten av LM av celler som uttrycker virusproteiner smält till en fluorescerande protein (FP). Dessa celler är därefter cryo-orörlig och vidare beredda för ultrastrukturella analys via transmissionselektronmikroskopi (TEM) att få nya insikter i hur uttrycket av dessa proteiner ordna intracellulära membran (figur 1). CLEM har utförts med kemiskt fast celler i de flesta virologi studier publicerade till datum8,9,10,11,12,13,14 ,15,16,17,18,19. Detta beror främst på behovet av inactivating smittsamt material för biosäkerhet skäl biosäkerhetsnivå-2 och-3 (BSL-2 och BSL-3) laboratorier, där cryo-immobilisering av celler inte är oftast möjligt. För de frågor som kräver en optimal bevarandet av cellmembranen, förglasning via högtryck frysning (HPF), dock rekommenderas20. I dessa fall kan det CLEM-protokollet som beskrivs här tillämpas. Intressant, särskilt vid arbete med infektiösa exemplaren, kan HPF utföras på prover som varit tidigare kemiskt inaktiverade, till exempel i BSL-2 och BSL-3 laboratorier. Kombinationen av kemisk fixering följt av HPF är en möjlighet att åtminstone delvis utnyttja fördelarna med kryo-bevarande metoder21,22.
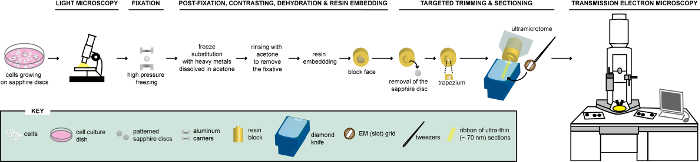
Figur 1 : Schematisk representation av arbetsflödet för analys av celler via CLEM. Celler som växer på mönstrade safir skivor analyseras först av LM att lokalisera celler som uttrycker FPs innan deras bearbetning för EM. När ligger, korrigeras omedelbart celler av HPF och FS bäddas därefter i kåda. Vid polymerisation av harts, stöd där cellerna växer (= safir skivor) måste tas bort från blocket harts. Blocket som innehåller inbäddade celler trimmas till en liten trapezium som de resterande celler, som uttrycker FPs, är sektioneras med diamond kniv. Ultratunna sektioner samlas in på slot rutnät och undersökas ytterligare genom TEM få ultrastrukturella information av dessa celler. Denna siffra är anpassade och modifierad med tillstånd från referens29. Klicka här för att se en större version av denna siffra.
Protocol
Representative Results
Discussion
CLEM metoden presenteras här för att studera effekterna av en viral proteinuttryck på cellmembran har använts framgångsrikt innan för att belysa den HCV replikering-associerade strukturer, främst dubbla membran blåsor (DMVs)21, samt att bestämma de kritiska byggstenar som krävs för att bilda dessa HCV-inducerad strukturer23. Observera att i vårt första arbete använder CLEM för att studera HCV replikering21, tillämpades en något modifierad version av protokollet som beskrivs här. Däri studie, konventionella 0,05 mm tjock safir skivor, utan alfanumeriskt mönster, användes som en referens mönster skapades av kol beläggning dem med ett finder rutnät ovanpå (se Tabell för material). Denna version av det nuvarande protokollet kan tillämpas så småningom, med fördelen att ”A” transportören för HPF kan användas direkt, utan att behöva skära som i figur 3A. Alternativt, som nämns i protokollet, en tjockare ”en” carrier kan utnyttjas (se Tabell för material) med mönstrade safir skivor, utan behov av en ”B-bärare.
Intressant, kan detta protokoll tillämpas inte bara att studera BSL-1 prover, såsom celler transfekterade med virusproteiner som beskrivs här och annorstädes19,23, men också att studera virusinfekterade celler. Även arbeta med mänskliga patogener är vanligtvis begränsad till BSL-2 och BSL-3 laboratorier, i vissa länder är det fortfarande möjligt att utföra cryo-immobilisering under dessa förhållanden för biosäkerhet. I dessa BSL-2 och BSL-3 laboratorier där förglasning inte är möjligt, på grund av lokala föreskrifter eller avsaknad av en HPF maskin, kan virus-infekterade celler fortfarande förberedas med den här metoden om kemiska fixering med aldehydes utförs i förväg, nämligen innan de lämnar de BSL-2 eller BSL-3 faciliteterna. Vidare behöver aldehyder kvävas omedelbart efter fixering att hålla fluorescensen, medan resten av protokollet är identisk med den som beskrivs här. Denna teknik kan anses överflödigt eftersom cellerna är fasta två gånger, kemiskt och via förglasning. Detta Dubbelrum fixering protokoll leder dock faktiskt till en mycket bättre bevarande av de HCV-inducerad DMVs i jämförelse med de DMVs som finns i celler utsätts för kemiska fixering enbart21.
För ytterligare ändringar och felsökning läsaren hänvisas till anteckningarna under hela den protokoll delen av detta manuskript. Dessa anteckningar beskriva fallgropar att undvika, samt alternativ till övervinna eventuella svårigheter som kan uppstå när du utför denna metod.
De huvudsakliga erforderliga för tillämpning av denna teknik är en HPF maskin. När cryo-immobilisera celler via HPF inte är genomförbar (på grund av frånvaron av en HPF maskin) eller inte krävs (när membranet bevarandet inte behöver vara optimal), celler kan kemiskt fast och därefter förberedas och analyseras av EM8, 9,10,11,12,13,14,15,16,17,18 ,19. Detta alternativ kräver inte användning av safir skivor, men cellen kultur rätter med inrutade mönster för flyttar celler eller cell kluster. Den största fördelen med användningen av dessa rätter är deras större diameter jämfört med safir skivor, vilket gör att screening av större ytor. Tillämpningen av detta CLEM protokoll har således tillämpats framgångsrikt att studera effekten av en antiviral förening mot HCV15 eller att visualisera de membran rearrangements inducerad av ickestrukturella proteiner av Norovirus19. En annan fråga som kan begränsa prestanda för denna metod är avsaknaden av en kommersiell FS-enhet. I det här fallet kan grundläggande hemmagjord FS system utnyttjas istället. Även automatisk FS enheter kan minska hantering missöden, Hemmagjord enheter används framgångsrikt exempel på Kent Mcdonald’s30 och Paul Walther labs.
Med avseende på kemiska fixering säkerställer protokollet beskrivs här ett optimalt bevarande av intracellulära strukturer20. Därför, om de ovannämnda HPF och FS-enheterna är tillgängliga, vitrifying celler av intresse skulle vara att föredra.
Framtida alternativ till detta CLEM synsätt inkluderar möjligheten att använda denna metod inte bara att förvärva 2D information på ultrastrukturella nivå, men också att få 3D information om arkitekturen av membran och organell förändringar orsakade av virus. 3D-EM metoderna, inklusive electron tomografi (ET) och fokuserad ion beam-scanning elektronmikroskopi (FIB-SEM) (utförligt beskrivs i29), kan också tillämpas på celler som har upprättats efter detta nuvarande protokoll21, 31. Dessutom kan 3D-information också erhållas på LM nivå, när du använder en confocal Mikroskop, vilket gör förvärvet av z-stackar. I själva verket rekommenderas detta alternativ när ett exakt samband mellan LM och EM datamängder är önskad (se till exempel17). Den information som ingår i 3D z-stackar aids att förbättra överensstämmelsen med 2D TEM bilder. Således, i ett sådant scenario, bäst passande LM och EM bilder kan väljas och sedan genomgå en av de tillgängliga, såsom EG-CLEM plugin av ICY (http://icy.bioimageanalysis.org/)32 eller landmärke korrespondenser plugin av korrelation Bild J (http://imagej.net/Landmark_Correspondences), vilket resulterar i generationen av överlappande LM-EM bilder.
När temporal information som behövs för att förstå kineticsen av en viss händelse, kan time-lapse imaging användas för att övervaka dynamiken i levande celler i kombination med EM. Under evenemanget av intresse korrigeras celler omedelbart generera en ”frusen ögonblicksbild” som kan analyseras därefter via EM, som ger detaljerad ultrastrukturella information om just det ögonblicket vid tidpunkten för immobilisering. För att få den ”frusen ögonblicksbilden”, efter observationen i realtid, kan celler vara antingen kemiskt fast33 eller cryo-orörlig6. Eftersom många cellulära processer sker snabbare än diffusion processerna för kemiska fixering, om möjligt, bör Ultra snabb nedfrysning utföras. Det är dock viktigt att beakta att HPF maskinerna skiljer sig åt i deras effektiva tiden resolution34.
Vidare, även om detta protokoll har utformats för för inbäddning av celler i ett epoxiharts, celler kan också bäddas in i låg viskositet hartser, såsom Lowicryls, LR vit eller LR guld. Användningen av dessa bädda media gör det möjligt för att bevara den antigenicitet35,36, samt fluorescens37,38 och, därför används mestadels för efter inbäddning på-sektionen immunolabeling39 , 40 och på-avsnitt CLEM41,42,43,44, där LM görs efter för inbäddning. Båda metoderna (immuno-EM och på-avsnitt CLEM) måste vara avgörande för dessa experiment där icke-karakteristiska strukturer kan lätt hittas via TEM eller som en kontroll mot miscorrelation mellan LM och EM signaler. Jämväl, märkning med antikroppar som kan visualiseras genom båda avbildningsmetoder (LM och EM) kan utföras45 för att, till exempel identifiera transfekterade celler i LM (pre inbäddning scenen) och uppnå en mycket mer exakt lokalisering av den GFP signal via dess särskilda märkning av immuno-EM (efter inbäddning Stadium). Det måste beaktas, men att permeabilisering görs före LM att tillåta åtkomst av antikroppar till intracellulära utrymmet, vilket kan resultera i ett optimalt bevarande av cell arkitektur på EM-nivå. Intressant, detta protokoll är också väl lämpad för flerfärgade experiment som kan uppnås med användning av andra fluorescerande taggar, än GFP (som visas här). Sammanfattningsvis finns det många förmodade möjligheter att anpassa detta protokoll, både på LM eller på EM sidorna, beroende på de frågor som behandlas. En omfattande beskrivning av andra alternativa protokoll hänvisas till5,46. Oavsett hur modaliteterna mikroskopi kombineras, är tillsammans resultatet en vinst av information, gör det möjligt för oss att bättre förstå hur virus och deras proteiner interagerar med sina värdar i verkliga livet.
Det mest kritiska steget inom denna metod är samlingen av seriell avsnitt från cellerna av intresse. Så framhävde i avsnittet representativa resultat kräver detta sakkunnig personal, samt en hel del tålamod. Detta steg är viktigt, viktigt att hitta celler tillbaka på EM-nivå av två skäl. Först i denna typ av CLEM protokoll använder pre inbäddning LM, visas koordinaterna endast på LM och i harts block ansiktet efter för inbäddning. Dock kommer de inte vara synliga på avsnitten av TEM. Därför skall riktade trimning i block ansiktet ner till regioner i intresse (ROIs) utföras noggrant med ett rakblad för att säkerställa att de avsnitt som erhålls därefter innehåller de celler som uttrycker en viss FP. Andra är ”skanna” flera sektioner nödvändigt att hitta bästa överlägget mellan LM och EM förvärven. Till skillnad från metoder där LM utförs efter inbäddning skede, är i detta fall överlägget LM-EM inte så exakt. Låg overlay noggrannheten beror på skillnader i axiell resolution LM och EM, krympning under provet bearbetningen av EM och komprimering vid snittning42. Dock bidra effektiv spårningsmetoder, såsom användning av sevärdheter, till att hitta celler tillbaka. Detta inkluderar en cell till en annan position, liksom formen av cellerna och deras kärna. I detta avseende, såsom förklaras i protokollet, ger DIC bilder ”anatomiska” information av de celler som är avgörande för att förbättra sambandet. Alternativt, kärnor eller andra väl-kända cell organeller (såsom mitokondrier eller lipid droppar) kan färgas innan LM och användas som landmärken.
Slutligen är det anmärkningsvärt att nämna att även detta manuskript är inriktat på användningen av denna teknik för virologi studier, omfattningen av denna experimentella design kan utökas för att mer generella biologiska frågor.
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Vi är mycket tacksamma för anställda av elektronmikroskopi Core faciliteter (EMCF) vid EMBL (Heidelberg) och vid universitetet i Heidelberg. Vi vill också tacka Ulrike Herian, Stephanie Kallis och Andrea Hellwig (Heidelberg universitet), samt Eberhardt Schmidt och Renate Kunz (universitetar av Ulm) för teknisk experthjälp. Arbete av R.B. och hans team (U.H. och I.R.-B.) stöddes av Deutsche Forschungsgemeinschafts, SFB1129, TP11 och TRR83, TP13.
Materials
| UV Crosslinker | Stratagene | 400072 | Stratalinker 1800, 230Vac, equipped with 254-nm UV light bulbs, 8-watts/each. |
| Patterned sapphire discs | Engineering Office M. Wohlwend, Sennwald, Switzerland | 627 | They have a 0.3-mm diameter and are 0.16-mm thick, as well as an an etched alphanumeric pattern to allow for re-location of the cells. They can be only ordered per e-mail: martin-wohlwend@bluewin.ch. |
| Slim and long tweezers | Electron Microscopy Sciences, Hatfield, PA, USA | 72919-SS | "Style SS", for handling sapphire discs. |
| Cell culture medium | Thermo Fisher Scientific, Waltham, MA, USA | 41965-062 | Dulbecco's modified Eagle medium (DMEM) supplemented with 2 mM L-glutamine, nonessential amino acids, 100 units penicillin per ml, 100 µg streptomycin per ml and 10% fetal calf serum (DMEM complete, see below). Package containing 10 bottles, 500 ml/each. |
| L-glutamine | Thermo Fisher Scientific, Waltham, MA, USA | 25030-123 | Package containing 20 bottles of 100 ml/each. |
| Nonessential amino acids | Thermo Fisher Scientific, Waltham, MA, USA | 11140-068 | Package containing 20 bottles of 100 ml/each. |
| Penicillin/Streptomycin | Thermo Fisher Scientific, Waltham, MA, USA | 15140-163 | Package containing 20 bottles of 100 ml/each. |
| Fetal calf serum | Sigma Aldrich, St. Louis, MI, USA | F7524 | Bottle of 50 ml. |
| Inverted microscope | Olympus Deutschland, Hamburg, Germany | CKX41 | It is an inverted microscope with trinocular head options and fluorescence upgrade capability. |
| Huh7-Lunet cells stably expressing the T7 RNA-polymerase | Avaliable at Prof. Dr. Ralf Bartenschlager laboratory | Not available | Contact Prof. Bartenschlager at: ralf.bartenschlager@med.uni-heidelberg.de. |
| Mirus TransIT-LT1 Transfection Reagent | Mirus Bio LLC, Madison, USA | MIR 2304 | Broad spectrum, low toxicity, DNA transfection reagent. Vial of 0.4 ml. |
| Inverted widefield fluorescence microscope | Carl Zeiss Microscopy GmbH, Germany | Zeiss Observer.Z1 | Inverted fluorescence microscope for experiments involving living cell cultures. |
| 1-hexadecene | Merck, Darmstadt, Germany | 8220640100 | Bottle of 100 ml. |
| "A" aluminium holders for high pressure frezing (HPF) | Engineering Office M. Wohlwend, Sennwald, Switzerland | 241 | This is the holder that has been used for this protocol and has 2 different depths of 0.1 and 0.2-mm. It can be only ordered per e-mail: martin-wohlwend@bluewin.ch. |
| "B" aluminium holders for HPF | Engineering Office M. Wohlwend, Sennwald, Switzerland | 242 | This is the holder that has been used for this protocol.It is 0.3-mm thick.They can be only ordered per e-mail: martin-wohlwend@bluewin.ch. |
| "A" aluminium holder for HPF (special for working with patterned sapphire discs) | Engineering Office M. Wohlwend, Sennwald, Switzerland | 737 | This is the holder than can be used alone with the patterned sapphire disc (as an alternative to the current protocol), without the need of a "B" holder or the edition of the "A" holder. It is 0.84-mm thick. It can be only ordered per e-mail: martin-wohlwend@bluewin.ch. |
| Sapphire discs | Engineering Office M. Wohlwend, Sennwald, Switzerland | 405 | These sapphires discs are the "conventional" one, with a 0.3-mm diameter and a thickness of 0.05 mm . They can be also used for CLEM as an alternative instead of patterned sapphire discs. |
| Finder grids | Electron Microscopy Sciences, Hatfield, Philadelphia, USA | LF135-Cu | These 135 mesh grids are used to create an alphanumeric pattern on "conventional" sapphire discs (described above) via carbon coating, so that they can be used for CLEM. 100 grids/vial. |
| HPF machine | ABRA Fluid AG, Widnau, Switzerland | HPM 01 | This HPF machine has been used for this protocol. |
| HPF machine | Leica Microsystems, Vienna, Austria | EMPACT 2 | "EMPACT 2", as an alternative to the use of the HPM machine that it has been used in this protocol (described above). |
| Cryo-tubes | Thermo Fisher Scientific, Waltham, MA, USA | Z763667-500EA | For long term-storage of cryo-immobilized samples in liquid nitrogen. Package containing 500 cryovials. |
| Liquid nitrogen dewar | Cole-Parmer GmbH, Wertheim, Germany | GZ-05094-60 | For long term-storage of cryo-immobilized samples in liquid nitrogen, equipped with cryo-racks with a capacity of 1600 cryo-tubes. |
| Automatic freeze substitution (AFS) machine | Leica Microsystems, Vienna, Austria | EM AFS2 | This machine performs freeze substitution and progressive lowering of temperature (PLT) techniques and allows low temperature embedding and polymerization of resins. |
| Osmium tetroxide (OsO4) | Electron Microscopy Sciences, Hatfield, PA, USA | 19150 | 4% aqueous solution, one box containing 10 x 2 ml ampules. |
| Uranyl-Acetate (UA) | Serva, Heidelberg, Germany | 77879.01 | Bottle containing 25 grs of (UA)-2 H2O. |
| Sonicator | Bandelin Electronic, Berlin, Germany | 329 | "Sonorex Super RK 31" is a high-power ultrasonic cleaning bath for aqueous cleaning solution that is used in this protocol to mix OsO4 and Ua when preparing the FS medium. |
| Glass-distilled acetone | Electron Microscopy Sciences, Hatfield, PA, USA | 10015 | Bottle of 100 ml. |
| Stereomicroscope | Leica Microsystems, Vienna, Austria | Leica M80 | Routine stereomicroscope for daily inspections, equipped with a camera for capturing images. |
| Epoxy resin | Electron Microscopy Sciences, Hatfield, PA, USA | 14120 | Embed 812 is a kit containing several components that must be mix together in the proportions given by the manufacturers. |
| Flow through rings | Leica Microsystems, Vienna, Austria | 16707157 | Package containing 100 pieces. |
| Reagent baths | Leica Microsystems, Vienna, Austria | 16707154 | Package containing 100 pieces. |
| Ultramicrotome | Leica Microsystems, Vienna, Austria | Leica EM UC7 | Ultramicrotome for preparation of semi- and ultrathin sections at room temperature. |
| Ultra 35° diamond knife | Diatome Ltd., Nidau Switzerland |
AGG339-735 | This knife have an edge length of 3 mm. |
| Ultra fine tweezers | Electron Microscopy Sciences, Hatfield, PA, USA | E78318 | "Style 3X", for handling EM grids. |
| EM slot grids | Electron Microscopy Sciences, Hatfield, PA, USA | G2010-Cu | 100 grids/vial. |
| EM grid storage box | Electron Microscopy Sciences, Hatfield, PA, USA | 71155 | It has capacity for 100 grids. |
Riferimenti
- Morgan, C., Godman, G. C., Breitenfeld, P. M., Rose, H. M. A correlative study by electron and light microscopy of the development of type 5 adenovirus. I. Electron microscopy. Journal of Experimental Medicine. 112, 373-382 (1960).
- Godman, G. C., Morgan, C., Breitenfeld, P. M., Rose, H. M. A correlative study by electron and light microscopy of the development of type 5 adenovirus. II. Light microscopy. Journal of Experimental Medicine. 112, 383-402 (1960).
- Caplan, J., Niethammer, M., Taylor, R. M., Czymmek, K. J. The power of correlative microscopy: multi-modal, multi-scale, multi-dimensional. Current Opinion in Structural Biology. 21, 686-693 (2011).
- de Boer, P., Hoogenboom, J. P., Giepmans, B. N. Correlated light and electron microscopy: ultrastructure lights up. Nature Methods. 12, 503-513 (2015).
- Müller-Reichert, T., Verkade, P. . Correlative light and electron microscopy III, First edition. , (2017).
- Brown, E., Mantell, J., Carter, D., Tilly, G., Verkade, P. Studying intracellular transport using high-pressure freezing and Correlative Light Electron Microscopy. Seminars in Cell and Developmental Biology. 20, 910-919 (2009).
- Bykov, Y. S., Cortese, M., Briggs, J. A., Bartenschlager, R. Correlative light and electron microscopy methods for the study of virus-cell interactions. FEBS Letters. , (2016).
- Spuul, P., et al. Assembly of alphavirus replication complexes from RNA and protein components in a novel trans-replication system in mammalian cells. Journal of Virology. 85, 4739-4751 (2011).
- Nagel, C. H., Dohner, K., Binz, A., Bauerfeind, R., Sodeik, B. Improper tagging of the non-essential small capsid protein VP26 impairs nuclear capsid egress of herpes simplex virus. PLoS One. 7, 44177 (2012).
- Sharma, M., Kamil, J. P., Coughlin, M., Reim, N. I., Coen, D. M. Human cytomegalovirus UL50 and UL53 recruit viral protein kinase UL97, not protein kinase C, for disruption of nuclear lamina and nuclear egress in infected cells. Journal of Virology. 88, 249-262 (2014).
- Kallio, K., et al. Template RNA length determines the size of replication complex spherules for Semliki Forest virus. Journal of Virology. 87, 9125-9134 (2013).
- Martinez, M. G., Snapp, E. L., Perumal, G. S., Macaluso, F. P., Kielian, M. Imaging the alphavirus exit pathway. Journal of Virology. 88, 6922-6933 (2014).
- Lebrun, M., et al. Varicella-zoster virus induces the formation of dynamic nuclear capsid aggregates. Virology. 454-455, 311-327 (2014).
- Madela, K., et al. A simple procedure to analyze positions of interest in infectious cell cultures by correlative light and electron microscopy. Methods in Cell Biology. 124, 93-110 (2014).
- Berger, C., et al. Daclatasvir-like inhibitors of NS5A block early biogenesis of hepatitis C virus-induced membranous replication factories, independent of RNA replication. Gastroenterology. 147, 1094-1105 (2014).
- van der Schaar, H. M., et al. Illuminating the Sites of Enterovirus Replication in Living Cells by Using a Split-GFP-Tagged Viral Protein. mSphere. 1, (2016).
- Vale-Costa, S., et al. Influenza A virus ribonucleoproteins modulate host recycling by competing with Rab11 effectors. Journal of Cell Science. 129, 1697-1710 (2016).
- Wang, L., et al. Visualization of HIV T Cell Virological Synapses and Virus-Containing Compartments by Three-Dimensional Correlative Light and Electron Microscopy. Journal of Virology. 91, (2017).
- Doerflinger, S. Y., et al. Membrane alterations induced by nonstructural proteins of human norovirus. PLOS Pathogens. 13, 1006705 (2017).
- Dahl, R., Staehelin, L. A. High-pressure freezing for the preservation of biological structure: theory and practice. Journal of Electron Microscopy Technique. 13, 165-174 (1989).
- Romero-Brey, I., et al. Three-dimensional architecture and biogenesis of membrane structures associated with hepatitis C virus replication. PLOS Pathogens. 8, 1003056 (2012).
- Sosinsky, G. E., et al. The combination of chemical fixation procedures with high pressure freezing and freeze substitution preserves highly labile tissue ultrastructure for electron tomography applications. Journal of Structural Biology. 161, 359-371 (2008).
- Romero-Brey, I., et al. NS5A Domain 1 and Polyprotein Cleavage Kinetics Are Critical for Induction of Double-Membrane Vesicles Associated with Hepatitis C Virus Replication. MBio. 6, 00759 (2015).
- Dixit, R., Cyr, R. Cell damage and reactive oxygen species production induced by fluorescence microscopy: effect on mitosis and guidelines for non-invasive fluorescence microscopy. The Plant Journal. 36, 280-290 (2003).
- Jou, M. J., Jou, S. B., Guo, M. J., Wu, H. Y., Peng, T. I. Mitochondrial reactive oxygen species generation and calcium increase induced by visible light in astrocytes. Annals of the New York Academy of Sciences. 1011, 45-56 (2004).
- McDonald, K. L., Morphew, M., Verkade, P., Muller-Reichert, T. Recent advances in high-pressure freezing: equipment- and specimen-loading methods. Methods in Molecular Biology. 369, 143-173 (2007).
- Walther, P., Ziegler, A. Freeze substitution of high-pressure frozen samples: the visibility of biological membranes is improved when the substitution medium contains water. Journal of Microscopy. 208, 3-10 (2002).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314, 1-340 (1986).
- Romero-Brey, I., Bartenschlager, R. Viral Infection at High Magnification: 3D Electron Microscopy Methods to Analyze the Architecture of Infected Cells. Viruses. 7, 6316-6345 (2015).
- McDonald, K. L., Webb, R. I. Freeze substitution in 3 hours or less. Journal of Microscopy. 243, 227-233 (2011).
- Villinger, C., Neusser, G., Kranz, C., Walther, P., Mertens, T. 3D Analysis of HCMV Induced-Nuclear Membrane Structures by FIB/SEM Tomography: Insight into an Unprecedented Membrane Morphology. Viruses. 7, 5686-5704 (2015).
- Paul-Gilloteaux, P., et al. eC-CLEM: flexible multidimensional registration software for correlative microscopies. Nature Methods. 14, 102-103 (2017).
- Polishchuk, R. S., Mironov, A. A. Correlative video light/electron microscopy. Current Protocols in Cell Biology Supplement. , 8 (2001).
- McDonald, K. L. A review of high-pressure freezing preparation techniques for correlative light and electron microscopy of the same cells and tissues. Journal of Microscopy. 235, 273-281 (2009).
- Newman, G. R., Jasani, B., Williams, E. D. A simple post-embedding system for the rapid demonstration of tissue antigens under the electron microscope. The Histochemical Journal. 15, 543-555 (1983).
- Schwarz, H., Humbel, B. M. Influence of fixatives and embedding media on immunolabelling of freeze-substituted cells. Scanning Microscopy. 3, 57-63 (1989).
- Luby-Phelps, K., Ning, G., Fogerty, J., Besharse, J. C. Visualization of identified GFP-expressing cells by light and electron microscopy. Journal of Histochemistry and Cytochemistry. 51, 271-274 (2003).
- Nixon, S. J., et al. A single method for cryofixation and correlative light, electron microscopy and tomography of zebrafish embryos. Traffic. 10, 131-136 (2009).
- McDonald, K. L. Rapid embedding methods into epoxy and LR White resins for morphological and immunological analysis of cryofixed biological specimens. Microscopy and Microanalysis. 20, 152-163 (2014).
- Webster, P., Schwarz, H., Griffiths, G. Preparation of cells and tissues for immuno EM. Methods in Cell Biology. 88, 45-58 (2008).
- Kukulski, W., et al. Correlated fluorescence and 3D electron microscopy with high sensitivity and spatial precision. Journal of Cell Biology. 192, 111-119 (2011).
- Peddie, C. J., et al. Correlative and integrated light and electron microscopy of in-resin GFP fluorescence, used to localise diacylglycerol in mammalian cells. Ultramicroscopy. 143, 3-14 (2014).
- Hampoelz, B., et al. Pre-assembled Nuclear Pores Insert into the Nuclear Envelope during Early Development. Cell. 166, 664-678 (2016).
- Lemercier, N., et al. Microtome-integrated microscope system for high sensitivity tracking of in-resin fluorescence in blocks and ultrathin sections for correlative microscopy. Scientific Reports. 7, 13583 (2017).
- Takizawa, T., Powell, R. D., Hainfeld, J. F., Robinson, J. M. FluoroNanogold: an important probe for correlative microscopy. Journal of Biological Chemistry. 8, 129-142 (2015).
- Romero-Brey, I., Yamauchi, Y. 3D electron microscopy (EM) and correlative light electron microscopy (CLEM) methods to study virus-host interactions. Methods in Molecular Biology: Influenza Virus Methods & Protocols. , (2018).

