Um ensaio de espectroscopia de flutuação de fluorescência das interações da proteína-proteína em contatos do celular
Summary
Este protocolo descreve uma abordagem de baseados em espectroscopia de flutuação de fluorescência para investigar interações entre proteínas mediando as interações célula-célula, ou seja, as proteínas localizadas em cruzamentos de célula, diretamente em células vivas. Nós fornecemos orientações detalhadas na calibração de instrumentos, aquisição de dados e análise, incluindo correcções de fontes possível artefato.
Abstract
Uma variedade de processos biológicos envolve interações célula-célula, normalmente mediadas por proteínas que interagem na interface entre células vizinhas. De interesse, apenas alguns ensaios são capazes de sondagem especificamente tais interações diretamente em células vivas. Aqui, apresentamos um ensaio para medir a ligação de proteínas expressado nas superfícies das células vizinhas, em contatos do celular. Este ensaio consiste em duas etapas: mistura de celulas que expressam as proteínas de interesse fundido a diferentes proteínas fluorescentes, seguido por medições de espectroscopia de flutuação de fluorescência em contatos de celular usando um laser confocal microscópio. Podemos demonstrar a viabilidade deste teste em um contexto biologicamente relevante medindo as interações da proteína amiloide precursora, como 1 (APLP1) através de junções célula-célula. Nós fornecemos protocolos detalhados sobre a aquisição de dados usando técnicas baseadas em fluorescência (varredura de espectroscopia de correlação cruzada de fluorescência, número de correlação cruzada e análise de brilho) e as calibrações de instrumentos necessários. Além disso, discutimos os passos críticos na análise dos dados e como identificar e corrigir as variações de sinal externo, espúrios, tais como as causadas por movimento fotobranqueamento ou célula.
Em geral, o ensaio apresentado é aplicável a qualquer homo – ou interação da proteína-proteína heterotypic em contatos de celular, entre as células dos tipos iguais ou diferentes e pode ser implementado em um comercial laser confocal microscópio. Um requisito importante é a estabilidade do sistema, que deve ser suficiente para sondar difusiva dinâmica das proteínas de interesse ao longo de vários minutos.
Introduction
Muitos processos biológicos ocorrem aos sítios de interações célula-célula, por exemplo, célula-célula adesão1,2,3, celular fusão4 e reconhecimento celular5. Tais eventos são particularmente importantes durante o desenvolvimento de organismos multicelulares e para a comunicação célula-célula, por exemplo, durante as respostas imunes. Esses processos são normalmente mediados por proteínas que são localizadas na superfície, ou seja, na membrana plasmática (PM) de vizinhos células e passam por interações específicas no contato célula-célula que são precisamente regulamentados no espaço e no tempo. Em muitos casos, essas interações são homo direto – ou heterotypic-interações da proteína proteína trans , mas também podem envolver íons ou ligantes atuando como extracelular linkers1. Apesar de fundamental importância, há uma falta de ensaios de sondagem essas interações proteína-proteína específica diretamente no ambiente nativo de células vivas. Muitos métodos exigem também o rompimento da pilha (por exemplo, ensaios bioquímicos como co-imunoprecipitação6), fixação (por exemplo, algumas das técnicas de microscopia óptica de super-resolução e microscopia eletrônica de célula-célula entra em contato com7), ou são não-específicos, por exemplo, agregação /8,9ensaios de aderência. Para superar esse problema, técnicas de fluorescência foram implementadas com base na ressonância de fluorescência a energia transferência (FRET)10 ou fluorescência complementação11. No entanto, para atingir suficientemente pequenas distâncias entre fluorophores, esses métodos exigem etiquetas fluorescentes no lado extracelular das proteínas10, potencialmente interferir com interações trans .
Aqui, apresentamos um ensaio alternativo baseado em fluorescência para interações da proteína-proteína em contatos do celular. Esta abordagem combina abordagens de correlação cruzada de fluorescência (espectroscopia de correlação cruzada de fluorescência digitalização (sFCCS), número de correlação cruzada e brilho (ccN & B)) e a mistura de células expressando um construto de fusão da proteína de interesse, por exemplo, um receptor de adesão. Os receptores investigados nas duas células interagindo são rotulados com duas proteínas fluorescentes espectralmente separadas (FPs), do intracelular lateral (veja figura 1A).
Os métodos empregados são baseados na análise estatística das flutuações da fluorescência induzida pelo movimento difusiva de proteínas fluorescentes fusão através do volume focal de um laser confocal microscópio. Mais detalhadamente, o ensaio de sondas a difusão co das proteínas de interesse em ambos os PMs vizinhas em contatos do celular. Se as proteínas sofrem interações trans , estes complexos trans vão levar proteínas fluorescentes emitem em ambos os canais espectrais, causando a fluorescência correlacionados flutuações de ambos os emissores. Por outro lado, se nenhuma ligação ocorre, as flutuações números de proteínas para enfrentar a TPM será independentes, causando flutuações não correlacionadas. A aquisição pode ser realizada de duas maneiras: 1) sFCCS é baseado em uma varredura em forma de linha através do contato célula-célula e sondas efetivamente as interações em um ponto localizado na região de contato. Através de uma análise temporal das flutuações de fluorescência, sFCCS fornece também informação dinâmica, ou seja, os coeficientes de difusão de complexos de proteínas; 2) ccN & B baseia-se numa análise pixel-wise de uma sequência de imagens adquiridas nas regiões de contato célula-célula. Tem a capacidade de sondar e interações do mapa ao longo de toda a região (em um plano focal) entre em contato com, mas não fornece informações sobre a dinâmica. Ambos os métodos podem ser combinados com uma análise do brilho molecular, ou seja, o sinal de média fluorescência emitida na unidade de tempo por simples difusão complexos de proteínas e, assim, proporcionar estimativas a estequiometria de complexos de proteínas no contatos do celular.
Neste artigo, nós fornecemos protocolos detalhados para preparação de amostras, calibração de instrumentos, aquisição de dados e análise realizar o ensaio apresentado em um comercial laser confocal microscópio. As experiências podem ser executadas em qualquer instrumento equipado com contagem de fótons ou detectores analógicos e um objectivo com abertura numérica elevada. Mais discutimos os passos críticos do protocolo e fornecem esquemas de correção para vários processos, causando flutuações artefactual sinal, por exemplo, de detector de ruído, movimento fotobranqueamento ou célula. Originalmente desenvolvida para sonda interações entre as células aderentes, o ensaio pode ser modificado para células de suspensão, ou adaptados para sistemas de membrana modelo, por exemplo, vesículas de gigante unilamellar (GUVs) ou plasma gigante vesículas de membrana (GPMVs), permitindo que o quantificação de interações em ambientes diferentes lipídios ou, na falta de um citoesqueleto organizado12,13.
Espectroscopia de fluorescência correlação cruzada de digitalização é uma versão modificada da fluorescência espectroscopia de correlação cruzada14 e foi projetado especificamente para sondar a dinâmica difusiva lenta em lipídios membranas15. É baseado na aquisição de digitalizar linha perpendicular ao PM contendo as proteínas fluorescentes de interesse. Para sondar as interações de duas espécies de proteína rotulada de forma diferente, a aquisição é realizada em dois canais espectrais usando duas linhas de laser e duas janelas de deteção para fluorophores espectralmente separados. Devido a dinâmica de difusão lenta de proteínas no PM (D≤ ~ 1 µm2/s), uma cruz-conversa livre de medição pode ser realizada alternando-se o esquema de excitação de linha a linha15. A análise começa com: 1) é um algoritmo de alinhamento corrigindo para o movimento lateral da célula com base na block-wise uma média de ~ 1000 linhas, 2) determinação da posição com fluorescência máximo sinalou seja, a PM posição, em cada bloco e 3) deslocando de todos os blocos para uma comum origem12,15, separadamente em cada canal. Em seguida, uma seleção automática de pixels correspondentes ao PM é executada selecionando a região central de um ajuste Gaussian da soma de todas as linhas alinhadas (i.e., centro ± 2.5σ). Integração do sinal em cada linha produz a série de tempo de fluorescência do membrana f (t) em cada canal (g = green channel, r = canal vermelho). Nota que o tamanho do pixel deve ser pequeno o suficiente, por exemplo, < 200 nm, para reconstruir a forma do ponto de espalhar a função e encontrar o seu centro, correspondente à posição da PM. Na presença de fotobranqueamento substancial, a série de tempo de fluorescência em cada canal pode ser modelada com uma função dupla-exponencial e então corrigida com a seguinte fórmula:16
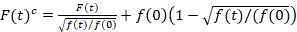 . (1)
. (1)
É importante notar que esta fórmula efetivamente corrige o amplitudes e tempos de difusão resultantes da análise de correlação de f (t)c, em comparação com as estimativas do parâmetro que seria obtidas da não corrigida f (t). Então, as funções de autoe correlação cruzada (ACFs / CCFs) da fluorescência, calculam-se os sinais:
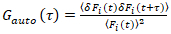 , (2).
, (2).
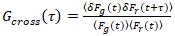 , (3).
, (3).
onde δFeu = Feu(t) – 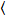 Feu(t)
Feu(t)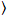 e eu = g, r.
e eu = g, r.
Um modelo de difusão bidimensional então é equipado para todas as funções de correlação (CFs):
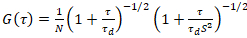 . (4)
. (4)
Aqui, N denota o número de proteínas fluorescentes no volume de observação e τd o tempo de difusão para cada canal. Este modelo leva em conta que no cenário experimental descrito, difusão de proteínas no PM ocorre no plano x-z, em contraste com a configuração comumente usada de correlação de fluorescência espectroscopia (FCS) experiências com membranas de sondagem difusão no plano x-y do confocal volume17. A cintura w0 e o fator de estrutura S, descrevendo o alongamento wz do volume focal em z, S = wz/w0, são obtidos a partir de uma medição de calibração de ponto FCS realizada com corantes espectralmente similares e mesmas configurações ópticas usando valores já está disponíveis para o coeficiente de difusão Dtintura:
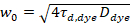 , (5).
, (5).
onde τd, a tintura é o tempo medido difusão média das moléculas do corante, obtidos a partir da montagem de um modelo para difusão tridimensional para os dados, tendo em transições de conta de uma fração T de todas as moléculas de N para um Estado de trio com uma constante de tempo ττ:
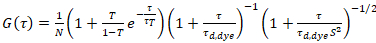 . (6)
. (6)
Finalmente, coeficientes de difusão (D), valores de brilho molecular (ε) e a correlação cruzada relativa dos dados de sFCCS (rel.cc.) são calculados como segue:
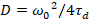 , (7).
, (7).
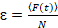 , (8).
, (8).
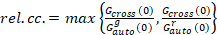 , (9).
, (9).
onde GCruz(0) é a amplitude da função de correlação cruzada e 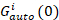 é a amplitude da função de autocorrelação no canal eu-th.
é a amplitude da função de autocorrelação no canal eu-th.
Esta definição da relativa correlação cruzada, ou seja, usando o máximo em vez de dizer na equação 9, leva em conta que o número máximo de complexos de duas espécies de proteínas presentes em diferentes concentrações é limitado pelo espécies presentes em um número menor.
Brilho e número de correlação cruzada é baseado em uma análise do momento da intensidade da fluorescência para cada pixel de uma pilha de imagem adquirida ao longo do tempo em uma posição fixa na amostra, geralmente constituídos por ~ 100-200 quadros, com espectral dois canais ( g = verde canal, r = canal vermelho). Da média temporal 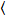 eu
eu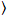 eu e a variância
eu e a variância 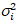 , o brilho molecular εi e número n são calculados em cada pixel e canal espectral (eu = g, r)18:
, o brilho molecular εi e número n são calculados em cada pixel e canal espectral (eu = g, r)18:
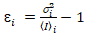 , (10).
, (10).
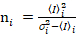 . (11)
. (11)
É importante notar que as equações determinadas se aplicam para o caso ideal de um verdadeiro detector de contagem de fótons. Para sistemas de detecção analógica, as equações seguintes aplicam-se a19,20:
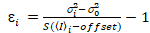 , (12).
, (12).
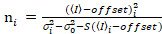 . (13)
. (13)
Aqui, S é o fator de conversão entre fótons detectados e as contagens de Digitas gravadas, 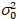 é o ruído de leitura e deslocamento refere-se ao deslocamento de intensidade do detector. Geralmente, essas quantidades devem ser calibradas, para qualquer tipo de detector, com base na variação do detector de medição em função da intensidade de iluminação constante19, por exemplo, uma superfície reflexiva de metal ou solução corante seco. O deslocamento pode ser determinada medindo-se a taxa de contagem para uma amostra sem luz de excitação. Realizando-se uma regressão linear da variância associada detector
é o ruído de leitura e deslocamento refere-se ao deslocamento de intensidade do detector. Geralmente, essas quantidades devem ser calibradas, para qualquer tipo de detector, com base na variação do detector de medição em função da intensidade de iluminação constante19, por exemplo, uma superfície reflexiva de metal ou solução corante seco. O deslocamento pode ser determinada medindo-se a taxa de contagem para uma amostra sem luz de excitação. Realizando-se uma regressão linear da variância associada detector 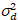 contra o enredo de intensidade (eu), S e
contra o enredo de intensidade (eu), S e 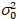 pode ser determinado19:
pode ser determinado19:
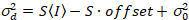 . (14)
. (14)
Finalmente, o brilho de correlação cruzada é calculado em cada pixel e é definido em geral como21
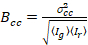 , (15).
, (15).
onde 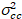 é a Cruz-variância
é a Cruz-variância 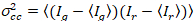 .
.
Para filtrar as flutuações long-lived, todos ccN & cálculos B são executados seguindo um vagão de filtragem, de forma independente para cada pixel de22. Brevemente, ni, εeu (eu = g, r) e Bcc são calculados em deslizamento segmentos de , por exemplo, 8-15 frames. Assim obtidos podem ser então a média dos valores para obter o último pixel valores número e brilho.
Análise de estequiometria
A fim de estimar a estequiometria de complexos de proteínas em contatos do celular, o brilho molecular pode ser analisado separadamente em cada canal espectral para o sFCCS ou ccN & B dados. No sFCCS, um valor de brilho é obtido por medição em cada canal. Em ccN & B, um histograma de brilho de todos os pixels correspondentes para o contato célula-célula é obtido e o valor médio (ou mediano) pode ser usado como brilho representativo para a medição. Realizando a mesma análise em uma referência monomérica, todos os valores de brilho podem ser normalizados para obter diretamente o estado oligoméricos médio dos complexos proteína detectada. Neste ponto, é importante corrigir a presença de FPs não-fluorescente, que pode resultar em uma subestimação do estado oligoméricas. Isto normalmente é realizado medindo-se o brilho de uma proteína dimérica homo referência do23,24 usando uma cor sFCS ou número e brilho (N & B).
Protocol
Representative Results
Discussion
O procedimento experimental descrito aqui permite a investigação da proteína-proteína trans interações em contatos de celular, empregando técnicas de espectroscopia de flutuação de fluorescência, ou seja, sFCCS e ccN & B. Esses métodos envolvem uma análise estatística das flutuações de fluorescência emitida por dois espectralmente separados FPs fundido para as somáticas de interesse em um contato de duas células vizinhas, cada uma expressando uma ou a outra proteína de fusão. A presença de c…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Este trabalho foi parcialmente financiado pela Deutsche Forschungsgemeinschaft (DFG) conceder 254850309. Os autores agradecer Madlen Luckner leitura crítica do manuscrito.
Materials
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
Riferimenti
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -. M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, 273 (2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Biologia dello sviluppo. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -. Y., Mok, L. -. P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634 (2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- . Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010)
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494 (2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073 (2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487 (2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412 (2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545 (2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).

