Oligomerisierungsdynamik von Zelloberflächenrezeptoren in lebenden Zellen durch totale interne Reflexionsfluoreszenzmikroskopie kombiniert mit Zahlen- und Helligkeitsanalyse
Summary
Wir beschreiben einen bildgebenden Ansatz zur Bestimmung des durchschnittlichen oligomeren Zustands von mEGFP-getaggten Rezeptor-Oligomeren, die durch Ligandenbindung in der Plasmamembran lebender Zellen induziert werden. Das Protokoll basiert auf der Total Internal Reflection Fluorescence (TIRF) Mikroskopie in Kombination mit der Number and Brightness (N&B)-Analyse.
Abstract
Trotz der Bedeutung und Allgegenwart der Rezeptor-Oligomerisierung sind nur wenige Methoden zum Erkennen von Clustering-Ereignissen und zur Messung des Clustering-Grades anwendbar. Hier beschreiben wir einen bildgebenden Ansatz zur Bestimmung des durchschnittlichen oligomeren Zustands von mEGFP-getaggten Rezeptor-Homokomplexen in der Membran lebender Zellen. Das Protokoll basiert auf der Total Internal Reflection Fluorescence (TIRF) Mikroskopie in Kombination mit der Number and Brightness (N&B)-Analyse. N&B ist eine Methode ähnlich der Fluoreszenz-Korrelationsspektroskopie (FCS) und dem Photonenzählhistogramm (PCH), die auf der statistischen Analyse der Schwankungen der Fluoreszenzintensität von Fluorophoren basieren, die in und aus einer Beleuchtung diffundieren. Volumen während einer Beobachtungszeit. Insbesondere ist N&B eine Vereinfachung von PCH, um Informationen über die durchschnittliche Anzahl von Proteinen in oligomeren Mischungen zu erhalten. Die Intensitätsschwankungsamplituden werden durch die molekulare Helligkeit des Fluorophors und die durchschnittliche Anzahl von Fluorophoren innerhalb des Beleuchtungsvolumens beschrieben. N&B berücksichtigt daher nur den ersten und zweiten Moment der Amplitudenverteilung, nämlich die mittlere Intensität und die Varianz. Dies ist gleichzeitig die Stärke und schwächelinde Methode. Da nur zwei Momente berücksichtigt werden, kann N&B den molaren Anteil unbekannter Oligomere in einer Mischung nicht bestimmen, sondern schätzt nur den durchschnittlichen Oligomerisierungszustand der Mischung. Dennoch kann es auf relativ kleine Zeitreihen (im Vergleich zu anderen Momentmethoden) von Bildern von lebenden Zellen pixelweise angewendet werden, einfach durch die Überwachung der Zeitschwankungen der Fluoreszenzintensität. Es reduziert die effektive Zeit pro Pixel auf wenige Mikrosekunden, wodurch die Erfassung im Zeitbereich von Sekunden bis Millisekunden möglich ist, was für eine schnelle Oligomerisierungskinetik erforderlich ist. Schließlich können große Zellbereiche sowie subzelluläre Kompartimente erkundet werden.
Introduction
Wir beschreiben einen Bildansatz der totalen inneren Reflexionfluoreszenz-Anzahl und Helligkeit (TIRF-N&B) zur Bestimmung des durchschnittlichen oligomeren Zustands von Rezeptormolekülen an der Plasmamembran lebender Zellen mit dem Ziel, die Rezeptor-Baugruppe zu verknüpfen. Dynamik zur biologischen Funktion der Proteine (Abbildung 1).
Bei extrazellulärer Ligandenbindung initiieren Rezeptoren die intrazelluläre Signaltransduktion in Abhängigkeit von ihrer Konformation, Oligomerisierung, potenziellen Co-Rezeptoren und Membranzusammensetzung. Trotz der Bedeutung und Allgegenwart der Rezeptor-Oligomerisierung, anerkannt als schlüsseleines Ereignis in der zellulären Signalisierung1,2,3,4,5,6, 7können nur wenige Methoden Clustering-Ereignisse erkennen und den Grad des Clusterings experimentell messen8,9. Das konfokale Volumen (x,y bei 300 nm, z x 900 nm) ist für den Nachweis der molekularen Interaktion und Stoichiometrie nicht ausreichend aufgelöst, auch nach der Optimierung durch Bildwiederherstellungsalgorithmen10. Die Untereinheitszusammensetzung von Proteinoligomeren kann nicht auch nur durch Superauflösungsmethoden bei einer Auflösung von 20-70 nm wie PALM11, STORM12und STED13rein räumlich gelöst werden. Darüber hinaus kann ihre zeitliche Auflösung (in der Reihenfolge von Minuten pro Bild) der Kinetik im Sekundenbereich nicht folgen. Einzelne Molekül-Stepbleichung löst die Stoichiometrie von Protein-Oligomeren nur, wenn sie unbeweglich sind14.
Eine der vielseitigsten Methoden zur Messung der Dichte und Oligomerisierung fluoreszierender Proteine in Einzelbildern ist die räumliche Intensitätsverteilungsanalyse (SpIDA), die auf räumlicher Probenahme beruht. Es ist sowohl auf chemisch fixierte als auch auf lebende Zellen anwendbar und ermöglicht die gleichzeitige Analyse mehrerer Interessenbereiche der Zelle mit Standardfluoreszenzmikroskopie15. Alternativ eignen sich Momentmethoden wie Fluoreszenz-Korrelationsspektroskopie (FCS)16, Photonenzählhistogramm (PCH)17und Zahl und Helligkeit (N&B)18,19, für quantitative oligomere Messungen. Diese Methoden analysieren die Fluoreszenzintensitätsschwankungen, die in der Zeit beobachtet werden können, wenn die Fluorophore in und aus einem Beleuchtungsvolumen diffundieren. Die Amplituden der Intensitätsschwankungen können eindeutig durch die molekulare Helligkeit des Fluorophors () und die durchschnittliche Anzahl der Fluorophore (n) innerhalb des Beleuchtungsvolumens17 (Abbildung 2) beschrieben werden. Typischerweise können der Diffusionskoeffizient der Fluorophore und die durchschnittliche Anzahl der Moleküle (umgekehrt bezogen auf den G(0)-Wert) innerhalb des Beleuchtungsvolumens durch FCS20ermittelt werden. Da die Diffusionszeit jedoch nur mit der kubischen Wurzel der Masse skaliert, ist FCS nicht empfindlich genug, um Veränderungen in der Molekularmasse21zu erkennen. In der Praxis kann einfarbiges FCS die Dimerisierung von Membranrezeptoren nicht erkennen. PCH löst Mischungen verschiedener Oligomere genau auf. Mit mehr als zwei Momenten der Amplitudenverteilung erkennt es Moleküle unterschiedlicher Helligkeit, die das gleiche Beleuchtungsvolumen belegen. Scannen von FCS22 und Entwicklungen, wie z. B. der interessante Paarkorrelationsansatz der molekularen Helligkeit (pCOMB)23, eingeführt, um den Alikatbereich der Anwendbarkeit von Fluoreszenzkorrelationsmethoden in biologischen Systemen zu erweitern24 bleiben Einzelpunktmethoden, denen die Fähigkeit zu schnellen Messungen in einem großen Zellbereich fehlt, was viele aufeinander folgende Beobachtungen an jedem Pixel und die Datenerfassung in der Reihenfolge von Sekunden erfordert.
N&B ist eine vereinfachte Version von PCH, die nur den ersten und zweiten Moment der Amplitude der Fluoreszenzverteilung berücksichtigt, nämlich die mittlere Intensität, , und die Varianz,2 (Abbildung 2)18,19 und aus diesem Grund kann sie den mollaren Anteil unbekannter Oligomere in einer Mischung nicht bestimmen, sondern schätzt nur den durchschnittlichen Oligomerisierungszustand der Mischung. Dennoch hat N&B den Vorteil, mit relativ kleineren Zeitreihen von Bildern von Lebendenzellen als PCH pixelweise zu arbeiten, indem einfach die Zeitlichen schwankungen der Fluoreszenzintensität überwacht werden. Da N&B die Zeit pro Pixel auf wenige Mikrosekunden reduziert, kann es einer schnellen Oligomerisierungskinetik über große Zellbereiche folgen und eine Bildaufnahme auf einer Zeitskala von Sekunden in der Raster-Scanmikroskopie (z. B. konfokale, 2-photon) und Millisekunden ermöglichen. in der kamerabasierten Mikroskopie (z.B. TIRFM).
Mehrere Berichte haben die Fähigkeit von N&B gezeigt, die Anzahl der Untereinheiten in Proteinclustern zu quantifizieren, indem erweiterte Zellregionen geabbildungen. Paxillin-EGFP-Cluster wurden an den Haftstellen in den CHO-K1-Zellen25nachgewiesen und die intrazelluläre Aggregation des pathogenen Httex1p-Peptids in COS-7-Zellen26beschrieben. N&B wurde nach der Liganden-gesteuerten Oligomerisierung des ErbB-Rezeptors27und der Wirkung des Liganden FGF21 auf Klothob (KLB) und FGFR1c in den HeLa-Zellen28angewendet. Die Kombination von TIRF-Bildgebung und N&B-Analyse wurde verwendet, um zu zeigen, dass Dynamin-2 in erster Linie tetramerisch in der gesamten Zellmembran29ist. Wir haben N&B sowohl auf Raster-Scanning als auch auf TIRF-Bilder angewendet, um die Liganden-gesteuerte Dimerisierung von uPAR- und FGFR1-Zellmembranrezeptoren30,31zu beweisen.
Fluoreszenzkorrelationsmethoden wie N&B, FCS und PCH basieren auf der Vorstellung, dass in einem offenen Volumen die Besatzungszahl der Teilchen einer Poisson-Verteilung folgt. Da nur die Photonen, die die Fluorophore emittieren, erkannt werden können, ist der Mittelwert  für eine gemessene Fluoreszenzintensität im Vergleich zur Zeit in einem Pixel des Bildes, das Produkt der durchschnittlichen Anzahl von Fluorophoren im Beleuchtungsvolumen, n, und molekulare Helligkeit, Nr.17:
für eine gemessene Fluoreszenzintensität im Vergleich zur Zeit in einem Pixel des Bildes, das Produkt der durchschnittlichen Anzahl von Fluorophoren im Beleuchtungsvolumen, n, und molekulare Helligkeit, Nr.17:

wobei die Anzahl der Photonen, die pro Zeiteinheit (konventionell pro Sekunde) pro Molekül emittiert werden, wenn sich das Molekül im Zentrum des Beleuchtungsvolumens befindet.
Helligkeit ist eine Eigenschaft jedes Fluorophors in einer bestimmten Anschaffung eingerichtet, während Intensität ist die Summe aller Beiträge aus allen Fluorophoren. In biologischen Wettbewerben wird die Helligkeit mit der Zunahme der Anzahl der Fluorophore, die zusammen schwanken, zunehmen, was Informationen über den Oligomerisierungszustand des fluoreszierend getaggten Proteins gibt. Die Fluktuationsamplituden an einem bestimmten Pixel werden anhand der Varianz des Fluoreszenzsignals gemessen,2:

Wobei der Mittelwert des Quadrats der Intensität  , und das
, und das  Quadrat des Mittelwerts der Intensität , aus den individuellen Intensitätswerten in jedem Pixel jedes Frames berechnet werden:
Quadrat des Mittelwerts der Intensität , aus den individuellen Intensitätswerten in jedem Pixel jedes Frames berechnet werden:
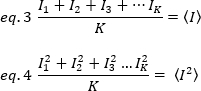
wobei K die Anzahl der Gesamtframes in der Zeitreihe ist. Experimentell ist es notwendig, für die gesamte Bildreihe die Varianz zu berechnen, die die Streuung der einzelnen Intensitätswerte an jedem Pixel eines einzelnen Bildes um den mittleren Intensitätswert beschreibt. Die Varianz umfasst alle Schwankungen unterschiedlicher Herkunft. In einer ersten Annäherung kann die Varianz durch die diffundierenden Partikel im Beleuchtungsvolumen,20, durch das Detektor-Schussrauschen von der Varianz getrennt werden,2d. Die beiden Varianzen sind unabhängig; somit wird die Gesamtabweichung durch ihre Summe angegeben:

Die Varianz aufgrund molekularer Schwankungen im und aus dem Nachweisvolumen ist linear abhängig von der molekularen Helligkeit und Intensität:

Neuanordnung eq. 6 nach 1q. 1:

Nach dem typischen Konzept der Fluoreszenzkorrelationsspektroskopie besagt Gleichung 7, dass die Varianz aufgrund der Anzahl der Schwankungen vom Quadrat der Partikelhelligkeit abhängt.
Dann ist die Varianz aufgrund von Detektorschwankungen eine lineare Funktion der erfassten Intensität, unter der Annahme, dass der Detektor unterhalb seiner Sättigungsgrenze19betrieben wird:

Bei Photonenzähldetektoren a=1 und c=0 entspricht die Detektorvarianz also der durchschnittlichen Intensität:

Um diese Konzepte auf reale Messungen in lebenden Zellen anzuwenden, definieren Gratton und Kollegen18 die scheinbare Helligkeit B für jedes Pixel als Verhältnis der Varianz zur durchschnittlichen Intensität:

B ist der Parameter, der experimentell gemessen wird. In dieser Arbeit werden Zeitreihenbilder von FGFR1-Rezeptoren an der Plasmamembran von HeLa-Zellen durch die TIRF-Mikroskopie erfasst und die durchschnittliche scheinbare Helligkeit B wird durch die N&B-Analyse bestimmt. Dann, nach Zugabe von FGF2, werden aufeinander folgende Zeitreihen erfasst, um die Veränderungen in der Selbstmontage der Rezeptormoleküle in der Membranoberfläche nach der Stimulation des Rezeptors mit dem kanonischen Liganden zu verfolgen.
Da es sich bei dem Detektor des TIRF-Mikroskops jedoch um eine EMVD-Kamera handelt, muss der Ausdruck für die scheinbare Helligkeit als19geändert werden:

wobei der Offset der Intensitätsausgleich der Erkennungselektronik ist, der ein Merkmal der Detektoreinstellungen ist. Die Varianz und die durchschnittliche Intensität eines analogen Detektors werden jeweils angegeben durch:
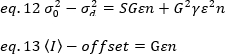
wobei G die analoge Verstärkung in digitalen Pegeln (DL/Photonen), S, die digitalen Pegel pro Photon19, durch die Steigung eines Intensitäts-Gegen-Varianzdiagramms für eine Lichtquelle mit konstanter Intensität (keine zeitlichen Schwankungen) angegeben wird. Der Faktor ist mit der Form des Pixelerkennungsvolumens verknüpft. Nach Hassler et al.32beträgt der Faktor 0,3 für die TIRF-Bildgebung, die mit der maximalen Verstärkung der Detektionskamera19arbeitet. Die Offset-, S- und G-Parameter sind Merkmale der Kamera und des Mikroskops. Die scheinbare Helligkeit B wird durch Neuanordnung von 11 nach 12 und 13 erreicht:

Experimentell ist die s eine komplexe Funktion der Laserintensität und der Detektionseffizienz des Systems. Da B/S jedoch linear von der Abhängigkeit von – abhängig ist, ist es nur wichtig, den relativen Wert von . für einen bestimmten Erkennungsmodus zu bestimmen:

wobei das S’ proportional zu . Dennoch wird eine Kalibrierung mit einem internen Verweis durchgeführt.
Protocol
Representative Results
Discussion
N&B erfordert mehrere Vorsichtsmaßnahmen bei der Wahl des Zellmodells und der Kennzeichnungsstrategie. Sie kann nur auf lebende Zellen angewendet werden, die während der Bildaufnahmezeit stabil haften bleiben. Zusätzliche Schwankungen durch die gesamte Zelle starre Verschiebung kann mit entsprechenden Bildwiederherstellungsansätzen38behandelt werden. Wenn sich jedoch eine Zelle bewegt, verformt sich die Zellmembran ebenfalls, und die Strukturverformung führt zu einer erheblichen zusätzlichen…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Die CNIC wird vom Ministerium für Ciencia, Innovacion y Universidades und der Pro CNIC Foundation unterstützt und ist ein Severo Ochoa Center of Excellence (SEV-2015-0505). Wir werden auch vom Europäischen Fonds für regionale Entwicklung (FEDER) “Una manera de hacer Europa” unterstützt. Die UC würdigt die Unterstützung der Associazione Italiana Ricerca sul Cancro, der Association for International Cancer Research (heute als Worldwide Cancer Research bekannt) und des italienischen Gesundheitsministeriums. A.T. würdigt die “Fondazione Banca del Monte di Lombardia” für die teilweise Unterstützung seiner Arbeit mit dem PV-Stipendium “Progetto Professionalit” Ivano Becchi” 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Riferimenti
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochimica. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

