ליגריזציה דינמיקה של קולטני פני שטח תא בתאי חיים על ידי סך הכל השתקפות פנימית מיקרוסקופ פלואורסצנטית בשילוב עם ניתוח מספר ובהירות
Summary
אנו מתארים את גישת ההדמיה לקביעת המצב oligomeric הממוצע של megfp-מתויג-קולטן מתויגים oligomeric הנגרמת על ידי ליגנד קשירה ממברנה פלזמה של תאים חיים. הפרוטוקול מבוסס על סך הכל מיקרוסקופ של השתקפות פנימית (TIRF) בשילוב עם ניתוח מספר ובהירות (N & B).
Abstract
למרות החשיבות והייתה של הקולטן, שיטות מעטות ישימות לגילוי אירועים באשכולות ומדידת מידת הקיבוץ. כאן, אנו מתארים גישה להדמיה כדי לקבוע את המצב oligomeric הממוצע של mEGFP-מתויג-קולטן הומוקומפלקסי בקרום של תאים חיים. הפרוטוקול מבוסס על סך הכל מיקרוסקופ של השתקפות פנימית (TIRF) בשילוב עם ניתוח מספר ובהירות (N & B). N & B היא שיטה דומה ספקטרוסקופיית מתאם פלואורסצנטית (FCS) והיסטוגרמה ספירת פוטון (PCH), אשר מבוססים על ניתוח סטטיסטי של תנודות של עוצמת הקרינה הפלואורסצנטית של fluorophores מפזרת פנימה והחוצה של תאורה אמצעי אחסון במהלך זמן התבוננות. בפרט, N & B הוא פישוט של PCH כדי להשיג מידע על המספר הממוצע של חלבונים בתערובות oligomeric. תנודות אינטנסיביות המוני מתוארים על ידי בהירות מולקולרית של fluorophore ואת המספר הממוצע של fluorophores בתוך עוצמת התאורה. כך, N & B רואה רק את הרגעים הראשונים והשני של התפלגות משרעת, כלומר, את העוצמה הממוצע ואת השונות. זה, באותו זמן, את הכוח והחולשה של השיטה. כי רק שני רגעים נחשבים, N & B לא יכול לקבוע את החלק הטוחנת של oligomers לא ידוע בתערובת, אבל זה רק מעריך את מצב oligomeriטיזציה הממוצע של התערובת. עם זאת, זה יכול להיות מיושם על סדרת זמן קטן יחסית (לעומת שיטות רגע אחר) של תמונות של תאים חיים על בסיס פיקסל על ידי פיקסל, פשוט על ידי ניטור תנודות הזמן של עוצמת הקרינה. זה מפחית את הזמן האפקטיבי לפיקסל לכמה מיקרו שניות, המאפשר רכישה בטווח הזמן של שניות לאלפיות השניה, אשר הכרחי עבור הקינטיקה מהירה של oligomerization. לבסוף, שטחי תאים גדולים כמו גם תאים תת סלולריים ניתן לחקור.
Introduction
אנו מתארים כולל השתקפות פנימית מספר ובהירות (TIRF-N & B) גישה להדמיה לקביעת מצב oligomeric הממוצע של מולקולות הקולטן בקרום פלזמה של תאים חיים, מכוון בקישור הרכבה הקולטן דינמיקה לתפקוד הביולוגי של החלבונים (איור 1).
עם ליגתאי ומחייב, קולטנים ליזום את האות תאיים התמרה האותות בהתאם הקונפורמציה שלהם, oligomerization, שיתוף הפוטנציאלי קולטנים והרכב קרום. למרות החשיבות והייתה של קולטן oligomerization, מוכר כאירוע מפתח באיתות סלולרי1,2,3,4,5,6, 7, כמה שיטות יכול לזהות אירועים באשכולות ולמדוד את מידת הניסויים באשכולות8,9. כרך קונפוקלית וקד (x, y ≈ 300 ננומטר, z ≈ 900 nm) הוא פתר מספיק להוכחת אינטראקציה מולקולרית וסטויכמטריה, גם לאחר אופטימיזציה על ידי אלגוריתמים שיקום התמונה10. הרכב התת-יחידתי של חלבון oligomers אינו ניתן לפענוח על בסיס מרחבי גרידא גם על ידי שיטות ברזולוציה גבוהה ב-x, y ברזולוציה של 20-70 ננומטר כגון PALM11, סטורם12, ו ההיסטד13. יתר על כן, הרזולוציה הטמפורלית שלהם (בסדר של דקות לכל תמונה) לא יכול לעקוב אחר הקינטיקה בטווח של שניות. מולקולה בודדת צעד הלבנת פותרת את סטויכיומטריה של oligomers חלבון רק אם הם משותק14.
אחת השיטות הרב-תכליתיות ביותר כדי למדוד צפיפות ו oligomerization של חלבונים מתויגים פלואורוסקופים בתוך תמונות בודדות היא התפלגות בעוצמה מרחבית ניתוח (SpIDA), אשר נשענת על דגימה מרחבית. הוא חל על תאים כימיים קבועים ולחיות, ומאפשר ניתוח של מספר אזורים של העניין של התא בו זמנית באמצעות מיקרוסקופ קרינה רגילה15. לחילופין, שיטות רגע, כגון ספקטרוסקופיית מתאם פלואורסצנטית (fcs)16, היסטוגרמה לספירת פוטון (PCH)17, ומספר ובהירות (N & B)18,19, מתאימים לאולייומר כמותית דידות. שיטות אלה לנתח תנודות בעוצמה פלואורסצנטית כי ניתן להבחין בזמן כאשר fluorophores לפזר והחוצה של נפח התאורה. המוני התנודות באינטנסיביות ניתן לתאר באופן ייחודי על-ידי הבהירות המולקולרית של הזוהר (ε) והמספר הממוצע של fluorophores (n) בתוך כרך התאורה17 (איור 2). בדרך כלל, מקדם דיפוזיה של fluorophores ואת המספר הממוצע של מולקולות (הקשורים באופן הפוך לערך G (0) בתוך עוצמת התאורה ניתן להשיג על ידי FCS20. עם זאת, מאז זמן הדיפוזיה רק עם השורש העוקב של המסה, FCS אינו רגיש מספיק כדי לזהות שינויים במסה מולקולרית21. בפועל, לצבע אחד FCS אין אפשרות לזהות הדיסמזציה של קולטני הממברנה. PCH פותר תערובות של oligomers שונים במדויק. באמצעות יותר משני רגעים של התפלגות משרעת, הוא מזהה מולקולות של בהירות שונה התופסים את אותו נפח תאורה. סריקת FCS22 ופיתוחים, כגון מתאם זוג מעניין של בהירות מולקולרית (pcomb) גישה23, הציג כדי להאריך את מגוון הישימות של שיטות מתאם פלואורסצנטית במערכות ביולוגיות24 , השארו שיטות חד שונות חסרות יכולת של מדידות מהירות בשטח גדול של תא, הדורשות תצפיות רצופות רבות בכל פיקסל ורכישת נתונים בסדר השניות.
N & B הוא גרסה פשוטה של PCH הרואה רק את הרגעים הראשונים והשני של משרעת התפלגות הזריחה, כלומר את העוצמה ממוצע, , ואת השונות, σ2 (איור 2)18,19 ובגלל זה, זה לא יכול לקבוע את החלק הטוחנת של oligomers לא ידוע בתערובת, אבל רק מעריך את המצב הממוצע oligomeriטיזציה של התערובת. עם זאת, N & B יש את היתרון של עבודה עם סדרת זמן קטן יחסית של תמונות של תאים חיים מאשר PCH על בסיס פיקסל על ידי פיקסל, פשוט על ידי ניטור תנודות בזמן של עוצמת הקרינה. כיוון ש-N & B מפחית את הזמן בפיקסל למספר מיקרו-שניות, הוא יכול לעקוב אחר מהירות הקינטיקה המנותרת באזורי תאים גדולים, ולאפשר רכישת תמונה בסרגל זמן של שניות במיקרוסקופ סריקת רסטר (למשל, confocal וקד, 2-פוטון) ואלפיות השניה במיקרוסקופיה המבוססת על מצלמה (למשל, TIRFM).
מספר דוחות הפגינו את היכולת של N & B כדי לכמת את מספר יחידות המשנה באשכולות חלבונים על-ידי הדמיה של אזורי תאים מורחבים. אשכולות פעבלין-EGFP זוהו באתרי הדבקה ב-CHO-K1 תאים25, והצבירה התאיים של הפפטיד Httex1p הפתוגניים תוארה ב-COS-7 תאים26. N & B הוחל לאחר oligomerization מונחה ליגאן של הקולטן ErbB27, ואת ההשפעה של ליגFGF21 על קלותוב (klb) ו FGFR1c בתאי הלה28. השילוב של הדמיה TIRF ו-N & B ניתוח שימש כדי להראות כי דינמיות ב-2 היא בעיקר tetrameric לאורך כל קרום התא29. החלתי N & B לשני סריקת רסטר ותמונות tirf להוכיח dimerization מונחה ליגור של upar ו FGFR1 תא קולטנים קרום30,31.
שיטות מתאם פלואורסצנטית, כגון N & B, FCS ו-PCH, מבוססות על הרעיון שבנפח פתוח מספר הכיבוש של החלקיקים עוקב אחר התפלגות פואסון. כי רק את הפוטונים כי פולטות fluorophores ניתן להבחין, ערך ממוצע של עוצמת הזריחה נמדד לעומת הזמן בפיקסל של התמונה  ,, הוא התוצר של המספר הממוצע של fluorophores נפח התאורה, n, ואת ה בהירות מולקולרית, ε17:
,, הוא התוצר של המספר הממוצע של fluorophores נפח התאורה, n, ואת ה בהירות מולקולרית, ε17:

כאשר ε מבוטא כמספר הפוטונים הנפלטים ליחידת זמן (מקובל לשניה) לכל מולקולה כאשר המולקולה נמצאת במרכז עוצמת התאורה.
בהירות היא תכונה של כל fluorophore ברכישה נתונה להגדיר, בעוד האינטנסיביות הוא הסכום של כל התרומות מכל fluorophores. בתחרויות ביולוגיות, הבהירות יגדל עם הגידול של מספר fluorophores המתאחד יחד, נותן מידע על המצב oligomerization של חלבון fluoresc, מתויג. מגביר תנודות בפיקסל נתון נמדד מן השונות של האות הפלואורסצנטית, σ2:

כאשר המשמעות של ריבוע העוצמה  , והריבוע של העוצמה
, והריבוע של העוצמה  , מחושבים מערכי העוצמה האינדיווידואליים בכל פיקסל בכל מסגרת:
, מחושבים מערכי העוצמה האינדיווידואליים בכל פיקסל בכל מסגרת:
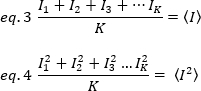
כאשר K הוא מספר המסגרות הכוללות בסידרת הזמן. ניסויים, יש צורך לחשב עבור סדרת התמונות כולה את השונות המתארת את פיזור ערכי העוצמה הבודדת בכל פיקסל של תמונה אחת סביב ערך העוצמה הממוצע. השונות כוללת את כל התנודות במקורות שונים. בקירוב ראשון, השונות הנובעת מהחלקיקים הניתנים לפיזור בעוצמת התאורה, σ20, יכולה להיות מופרדת מן השונות עקב רעש שוט הגלאי, σ2ד. שני השונויות הן עצמאיות; לפיכך, השונות הכוללת ניתנת על-ידי הסכום שלהם:

השונות, עקב תנודות מולקולריות של עוצמת האיתור והיציאה, תלויה בצורה לינארית בבהירות ובעוצמה המולקולרית:

מסידור מחודש של eq 6 לפי eq .1:

על פי המושג האופייני ספקטרוסקופיית מתאם פלואורסצנטית, משוואה 7 קובעת כי השונות בשל מספר התנודות תלויה בריבוע של בהירות החלקיקים.
לאחר מכן, השונות עקב תנודות גלאי היא פונקציה קווית של העוצמה שזוהתה, תחת ההנחה כי הגלאי מופעל מתחת למגבלת הרוויה שלה19:

במקרה של גלאים של ספירת פוטון a= 1 ו- c= 0, ולכן שונות הגלאי שווה לעוצמה הממוצעת:

כדי להחיל מושגים אלה על מדידות אמיתיות בתאים חיים, Gratton ועמיתים18 להגדיר את הבהירות לכאורה, B, עבור כל פיקסל כיחס של שונות על העוצמה הממוצעת:

B הוא הפרמטר הנמדד באמצעות הניסויים. בעבודה זו, סדרת הזמן תמונות של קולטני FGFR1 בקרום פלזמה של תאי הלה נלכדים על ידי מיקרוסקופ TIRF ואת הבהירות לכאורה הממוצעת, B, נקבעת על ידי ניתוח N & B. לאחר מכן, לאחר הוספת FGF2, סדרת הזמן ברציפות נלכדים לבצע את השינויים העצמית הרכבה של מולקולות הקולטן במשטח הקרום לאחר גירוי של הקולטן עם ligand קאנוני.
עם זאת, מאז גלאי המיקרוסקופ TIRF הוא מצלמה EMCCD, הביטוי לבהירות לכאורה צריך להיות שונה19:

כאשר היסט הוא היסט האינטנסיביות של האלקטרוניקה לאיתור שהוא מאפיין של הגדרות הגלאי. השונות והעוצמה הממוצעת עבור גלאי אנלוגי ניתנות בהתאמה על-ידי:
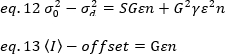
כאשר G הוא רווח אנלוגי ברמות דיגיטליות (DL/פוטונים), S, הרמות הדיגיטליות לכל פוטון19, ניתנת על ידי השיפוע של עוצמה לעומת מזימה שונות עבור מקור אור עם עוצמה מתמדת (אין תנודות בזמן). הפקטור הγ קשור לצורה של אמצעי האחסון לזיהוי פיקסלים. לדברי האסלר ואח ‘32, הפקטור γ שווה 0.3 עבור הדמיה tirf עבודה ברווח המקסימלי של מצלמת האיתור19. היסט, S ו-G פרמטרים הם מאפיינים של המצלמה ואת המיקרוסקופ. הבהירות לכאורה, B, מתקבלת על ידי סידור משנה של eq. 11 לפי eq .12 ו -13:

ניסויים, ε היא פונקציה מורכבת של עוצמת לייזר ויעילות הזיהוי של המערכת. עם זאת, מאחר ש-B/S תלוי באופן ליניארי ב ε, חשוב רק לקבוע את הערך היחסי של ε עבור מצב זיהוי נתון:

כאשר ε ‘ הוא פרופורציונלי לε. עדיין, כיול מבוצע באמצעות הפניה פנימית.
Protocol
Representative Results
Discussion
N & B דורש מספר אמצעי זהירות בבחירת דגם התא ואסטרטגיית התוויות. ניתן להחיל אותו רק על תאים חיים שנשארים ללא הפסק בזמן לכידת התמונה. תנודות נוספות בשל העקירה הנוקשה של התא כולו עשוי להיות מטופל עם גישות שחזור התמונה המתאים38. עם זאת, בדרך כלל כאשר התא זז, קרום התא גם deforms, ואת מבנה דפ…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
CNIC נתמך על ידי משרד של Ciencia, מרכז האוניברסיטה והקרן הפרו CNIC, והוא המרכז המרכזי של אוצ’ואה מצוינות (צב-2015-0505). אנחנו גם נתמכים על ידי הקרן האירופית לפיתוח האזור (פדר) “אונה סון נרה דה האג אירופה”. אוניברסיטת קליפורניה מכירה בתמיכה של האגודה לחקר הסרטן הבינלאומי (הידוע כיום כ מחקר הסרטן העולמי), ומשרד הבריאות האיטלקי. א. ש. מכירה בעמותת “פוננדאלקה דל מונטה די לומברדיה” לתמיכה חלקית בעבודתו עם מלגת PV “המקצוענים בצ” 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Riferimenti
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochimica. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

