استخراج الجينوم الموجه بالطيف الكتلي كأداة للكشف عن المنتجات الطبيعية الجديدة
Summary
يتم إنشاء و وصف بروتوكول تعدين الجينوم الموجه قياس الطيف الكتلي هنا. وهو يستند إلى معلومات تسلسل الجينوم وتحليل LC-MS/MS ويهدف إلى تسهيل تحديد الجزيئات من المستخلصات الميكروبية والنباتية المعقدة.
Abstract
المساحة الكيميائية التي تغطيها المنتجات الطبيعية هائلة وغير معترف بها على نطاق واسع. ولذلك، فإن المنهجيات الملائمة لإجراء تقييم واسع النطاق لوظائفها من حيث طبيعتها والفوائد البشرية المحتملة (مثل تطبيقات اكتشاف العقاقير) هي المنـزل. يصف هذا البروتوكول الجمع بين تعدين الجينوم (GM) والشبكات الجزيئية (MN)، وهما نهجان معاصران يطابقان التعليقات التوضيحية المشفرة العنقودية الجينية في تسلسل الجينوم الكامل مع توقيعات البنية الكيميائية من المستخلصات الأيضية الخام. هذه هي الخطوة الأولى نحو اكتشاف كيانات طبيعية جديدة. هذه المفاهيم، عند تطبيقها معا، يتم تعريفها هنا على أنها تعدين الجينوم الموجه MS. في هذه الطريقة، يتم تعيين المكونات الرئيسية مسبقًا (باستخدام MN)، وترتبط المرشحين الجدد ذات الصلة هيكلياً بالتعليقات التوضيحية لتسلسل الجينوم (باستخدام جنرال موتورز). الجمع بين جنرال موتورز وMN هو استراتيجية مربحة لاستهداف العمود الفقري جزيء جديد أو حصاد ملامح التمثيل الغذائي من أجل تحديد نظائرها من المركبات المعروفة بالفعل.
Introduction
غالبًا ما تتكون تحقيقات التمثيل الغذائي الثانوي من فحص المستخلصات الخام لأنشطة بيولوجية محددة يتبعها تنقية وتحديد وتوصيف المكونات التي تنتمي إلى كسور نشطة. وقد أثبتت هذه العملية كفاءتها، مما يعزز عزل العديد من الكيانات الكيميائية. ومع ذلك ، في الوقت الحاضر يعتبر هذا غير ممكن ، ويرجع ذلك أساسا إلى ارتفاع معدلات إعادة اكتشاف. وبما أن صناعة الأدوية أحدثت ثورة دون معرفة أدوار ووظائف الأيض المتخصصة، فقد تم تحديدها في ظروف مختبرية لا تمثل الطبيعة بدقة1. اليوم ، هناك فهم أفضل لتأثيرات الإشارات الطبيعية ، والإفراز ، ووجود معظم الأهداف بتركيزات منخفضة بشكل لا يمكن اكتشافه. بالإضافة إلى ذلك، سيساعد تنظيم العملية المجتمع الأكاديمي وصناعة الأدوية على الاستفادة من هذه المعرفة. كما سيفيد البحوث التي تنطوي على العزل المباشر للمستقلبات المتعلقة بمجموعات الجينات الاصطناعية البيولوجية الصامتة (BGCs)2.
وفي هذا السياق، جدد التقدم في التسلسل الجينومي الاهتمام بفرز نواتج الأيض الدقيقة. وذلك لأن تحليل المعلومات الجينومية لمجموعات التركيب الحيوي المكشوف ة يمكن أن يكشف عن الجينات ترميز المركبات الجديدة لا لوحظ أو تنتج في ظل ظروف المختبر. تتوفر العديد من مشاريع الجينوم الميكروبية الكاملة أو المسودات اليوم ، ويتزايد العدد كل عام ، مما يوفر آفاقًا هائلة للكشف عن جزيئات جديدة نشطة بيولوجيًا من خلال تعدين الجينوم3،4.
أطلس مجموعات الجينات التركيبية الحيوية هو أكبر مجموعة حالية من مجموعات الجينات الملغومة تلقائيًا كمكون من منصة الجينوم الميكروبي المتكامل لمعهد الجينوم المشترك (JGI IMG-ABC)2. وفي الآونة الأخيرة، عززت مبادرة توحيد مجموعات الجينات الاصطناعية الأحيائية الحد الأدنى من المعلومات (MIBiG) الشرح اليدوي لـ BGCs، مما يوفر مجموعة بيانات مرجعية منسقة للغاية5. في الوقت الحاضر ، تتوفر الكثير من الأدوات لتمكين التعدين الحسابي للبيانات الوراثية واتصالها بالأيض الثانوي المعروف. كما تم وضع استراتيجيات مختلفة للوصول إلى منتجات طبيعية جديدة نشطة بيولوجياً (أي التعبير غير المتجانس، وحذف الجينات المستهدفة، وإعادة التشكيل المختبري، والتسلسل الجينومي، والفحص الموجه بالنظائر [نهج جنوميسوتوبي]، والتلاعب بالمنظمين المحليين والعالميين، والتعدين القائم على الأهداف المقاومة، والتعدين المستقل للثقافة، ومؤخراً،نهجMS/code الموجهة2،6،,77،,8،9،, 10،11،12،13،14،15).
يتطلب تعدين الجينوم كاستراتيجية فريدة بذل جهود لتعليقات توضيحية لمجموعة واحدة أو صغيرة من الجزيئات. وبالتالي، لا تزال هناك ثغرات في العملية التي تعطى فيها الأولوية للمركبات الجديدة من أجل عزلها وتوضيح هيكلها. ومن حيث المبدأ، لا تستهدف هذه النهج سوى مسار واحد للتركيب الحيوي لكل تجربة، مما يؤدي إلى بطء معدل الاكتشاف. في هذا المعنى ، فإن استخدام جنرال موتورز جنبا إلى جنب مع نهج الشبكات الجزيئية يمثل تقدما هاما لبحوث المنتجات الطبيعية14،15.
إن تعدد الاستخدامات والدقة والحساسية العالية لقياس الطيف اللوني الكتلي السائل (LC-MS) يجعلها طريقة جيدة لتحديد المركب. حاليا، وقد استثمرت العديد من المنصات الخوارزميات وأجنحة البرمجيات لmetabolomics غير المستهدفة16،,17،,18،,19، 20,20. يتضمن جوهر هذه البرامج اكتشاف الميزات (ذروة الانتقاء)21 ومحاذاة الذروة ، مما يسمح بمطابقة الميزات المتطابقة عبر مجموعة من العينات والبحث عن الأنماط. الخوارزميات المستندة إلى نمط MS22،23 مقارنة أنماط التجزئة المميزة وتطابق أوجه التشابه MS2 التي تولد العائلات الجزيئية التي تشترك في الميزات الهيكلية. ويمكن بعد ذلك تسليط الضوء على هذه الميزات وتتجمع، ومنح القدرة على اكتشاف بسرعة جزيئات2معروفة وغير معروفة من استخراج البيولوجية المعقدة من قبل MS جنبا إلى جنب2،24،,25. ولذلك، فإن التصلب المتعدد المترادف هو طريقة متعددة الاستخدامات للحصول على معلومات هيكلية عن العديد من الأنماط الكيميائية الواردة في كمية كبيرة من البيانات في وقت واحد.
تستخدم خوارزمية الشبكات الجزيئية الاجتماعية للمنتجات الطبيعية العالمية (GNPS)26 كثافة الأيونات المجزأة العادية لبناء ناقلات متعددة الأبعاد ، حيث تتم مقارنة أوجه التشابه باستخدام وظيفة جيب الجيب. يتم رسم العلاقة بين الأيونات الأصلية المختلفة في تمثيل مخطط، حيث يتم تصور كل تجزئة كعقدة (دوائر)، ويتم تعريف الصلة بين كل عقدة بواسطة حافة (خطوط). يتم تعريف التصور العالمي للجزيئات من مصدر واحد كشبكة جزيئية. الجزيئات المتباينة هيكليا التي تتفتت بشكل فريد ستشكل مجموعة خاصة بهم أو كوكبة، في حين أن الجزيئات ذات الصلة تتجمع معا. يسمح الأنماط الكيميائية العنقودية باتصال افتراضي من الميزات الهيكلية المماثلة لأصولها التركيبية الحيوية.
الجمع بين كل من النمط الكيميائي إلى النمط الجيني ونهج النمط الجيني إلى النمط الكيميائي هو قوي عند إنشاء روابط المعلوماتية الحيوية بين BGCs ومنتجاتها جزيء صغير27. ولذلك، فإن تعدين الجينوم الموجه بالتصلب المتعدد هو طريقة سريعة واستراتيجية منخفضة الاستهلاك للمواد، ويساعد على سد الأيون اتّصال الوالدين والمسارات الاصطناعية الحيوية التي كشفت عنها WGS لسلالة واحدة أو أكثر في ظل ظروف استقلابية وبيئية متنوعة.
سير العمل من هذا البروتوكول(الشكل 1)يتكون من تغذية البيانات WGS في منصة الشرح الكتلة الجينية الاصطناعية مثل antiSMASH28،29،30. فهو يساعد على تقدير مجموعة متنوعة من المركبات وفئة من المركبات المشفرة من قبل الجينوم. يجب اعتماد استراتيجية لاستهداف مجموعة الجينات الاصطناعية الحيوية ترميز كيان كيميائي ذات فائدة، ومقتطفات الثقافة من سلالة نوع البرية و / أو سلالة غير متجانسة تحتوي على BGC يمكن تحليلها لتوليد الأيونات المجمعة على أساس أوجه التشابه باستخدام GNPS26،31. وبالتالي، فمن الممكن تحديد جزيئات جديدة ترتبط بـ BGC المستهدفة وغير متوفرة في قاعدة البيانات (نظائرها غير معروفة بشكل رئيسي، وتنتج في بعض الأحيان في تاتر منخفضة). ومن المهم النظر في أن المستخدمين يمكن أن تسهم في هذه المنصات وأن توافر المعلوماتية الحيوية وبيانات MS / MS يتزايد بسرعة ، مما يؤدي إلى تطوير مستمر ورفع مستوى الأدوات الحسابية الفعالة والخوارزميات لتوجيه اتصالات فعالة من مقتطفات معقدة مع الجزيئات.
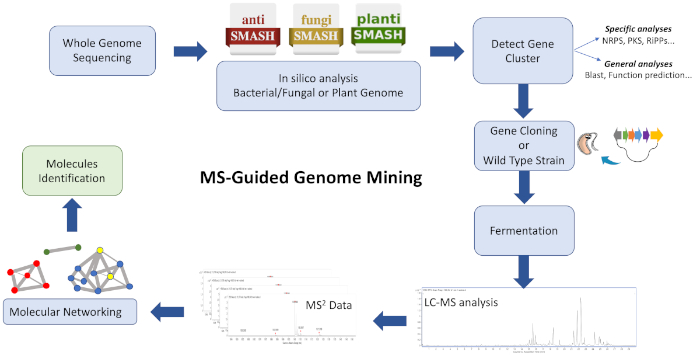
الشكل 1: نظرة عامة على سير العمل بأكمله. يُظهر هذا الشكل توضيحًا لخطوات المعلوماتية الحيوية والاستنساخ والشبكات الجزيئية التي تنطوي عليها نهج تعدين الجينوم الموجه بالتصلب المتعدد الموصوف لتحديد الأيض الجديدة. يرجى الضغط هنا لعرض نسخة أكبر من هذا الرقم.
يصف هذا البروتوكول سير العمل السريع والفعال للجمع بين تعدين الجينوم والشبكات الجزيئية كنقطة انطلاق لخط أنابيب اكتشاف المنتج الطبيعي. على الرغم من أن العديد من التطبيقات قادرة على تصور تكوين وعلاقة الجزيئات التي يمكن اكتشافها بالتصلب المتعدد في شبكة واحدة ، يتم اعتماد العديد منها هنا لتصور جزيئات مجمعة متشابهة هيكلياً. باستخدام هذه الاستراتيجية، يتم تحديد منتجات سيكلوبسيبتيد الرواية التي لوحظت في المستخلصات الأيضية من Streptomyces sp. CBMAI 2042 بنجاح. تسترشد التعدين الجينوم، يتم التعرف على كامل ترميز الكتلة الجينية الاصطناعية الحيوية لفالينوميسين واستنساخها في سلالة المنتج Streptomyces coelicolor M1146. وأخيراً، وبعد إقامة الشبكات الجزيئية القائمة على نمط MS، ترتبط الجزيئات التي تم اكتشافها بواسطة التصلب المتعدد بـ BGCs المسؤولة عن تكوينها الحيوي32.
Protocol
Representative Results
Discussion
الميزة الأقوى لهذا البروتوكول هي قدرته على إزالة الملامح الأيضية بسرعة وجسر المعلومات الجينومية مع بيانات التصلب المتعدد من أجل توضيح هياكل الجزيئات الجديدة ، وخاصة نظائرها الهيكلية2. واستناداً إلى المعلومات الجينية، يمكن تحري الأنماط الكيميائية للمنتجات الطبيعية المختلف…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
تم تقديم الدعم المالي لهذه الدراسة من قبل مؤسسة ساو باولو للأبحاث – FAPESP (2019/10564-5، 2014/12727-5 و2014/50249-8 إلى L.G.O؛ 2013/12598-8 و 2015/01013-4 إلى R.S.؛ و 2019/08853-9 إلى C.F.F.A). حصلت شركة B.S.P وC.F.F.A. و L.G.O. على زمالات من المجلس الوطني للتنمية العلمية والتكنولوجية – CNPq (205729/2018-5 و 162191/2015-4 و 313492/2017-4). كما أن L.G.O. ممتنة للدعم المقدم من المنح ة من قبل برنامج المرأة في العلوم (2008، الطبعة البرازيلية). جميع المؤلفين يعترفون بـ CAPES (التنسيق لتحسين موظفي التعليم العالي) لدعمبرامج ما بعد التخرج في البرازيل.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
Riferimenti
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

