Massespektrometristyrt genomgruvedrift som et verktøy for å avdekke nye naturlige produkter
Summary
En massespektrometristyrt genomgruveprotokoll er etablert og beskrevet her. Den er basert på informasjon om genomsekvens og LC-MS/MS-analyse og har som mål å lette identifiseringen av molekyler fra komplekse mikrobielle og planteekstrakter.
Abstract
Det kjemiske rommet dekket av naturlige produkter er enormt og allment ukjent. Derfor er praktiske metoder for å utføre omfattende evaluering av sine funksjoner i naturen og potensielle menneskelige fordeler (f.eks. for narkotikaoppdagelsesapplikasjoner) ønsket. Denne protokollen beskriver kombinasjonen av genomgruvedrift (GM) og molekylært nettverk (MN), to samtidige tilnærminger som samsvarer med genklyngekodet merknader i hele genomsekvensering med kjemiskstruktursignaturer fra rå metabolske ekstrakter. Dette er det første skrittet mot oppdagelsen av nye naturenheter. Disse begrepene, når de brukes sammen, er definert her som MS-guidet genomgruvedrift. I denne metoden er hovedkomponentene tidligere utpekt (ved hjelp av MN), og strukturelt relaterte nye kandidater er forbundet med genomsekvensmerknader (ved hjelp av GM). Kombinere GM og MN er en lønnsom strategi for å målrette nye molekyl ryggbein eller høste metabolske profiler for å identifisere analoger fra allerede kjente forbindelser.
Introduction
Undersøkelser av sekundær metabolisme består ofte av screening av råoljeekstrakter for spesifikke biologiske aktiviteter etterfulgt av rensing, identifikasjon og karakterisering av bestanddelene som tilhører aktive fraksjoner. Denne prosessen har vist seg å være effektiv, fremme isolasjon av flere kjemiske enheter. Men i dag er dette sett på som umulig, hovedsakelig på grunn av de høye gjenoppdagelsene. Som den farmasøytiske industrien revolusjonert uten kunnskap om rollene og funksjonene til spesialiserte metabolitter, ble deres identifikasjon utført under laboratorieforhold som ikke nøyaktig representerer natur1. I dag er det en bedre forståelse av naturlige signalpåvirkninger, sekresjon og tilstedeværelsen av de fleste mål ved undetectably lave konsentrasjoner. I tillegg vil regulering av prosessen hjelpe fagmiljøet og farmasøytisk industri til å dra nytte av denne kunnskapen. Det vil også være til nytte for forskning som involverer direkte isolering av metabolitter relatert til stille biosyntetiske genklynger (BGCs)2.
I denne sammenheng har fremskritt i genomisk sekvensering fornyet interessen for screening av mikroorganismemetabolitter. Dette er fordi analyse av genomisk informasjon om avdekkede biosyntetiske klynger kan avsløre gener koding nye forbindelser som ikke er observert eller produsert under laboratorieforhold. Mange mikrobielle hele genomprosjekter eller utkast er tilgjengelige i dag, og antallet vokser hvert år, noe som gir massive utsikter for å avdekke nye bioaktive molekyler gjennom genomgruvedrift3,4.
Atlas of Biosynthetic Gene Clusters er den nåværende største samlingen av automatisk utvunnet genklynger som en del av den integrerte mikrobielle genomer plattformen til Joint Genome Institute (JGI IMG-ABC)2. Senest har minimumsinformasjonen for biosyntetiske genklynger (MIBiG) standardiseringsinitiativ fremmet manuell reannotasjon av BGCer, noe som gir et svært kuratert referansedatasett5. I dag er mange verktøy tilgjengelig for å muliggjøre beregningsmessig gruvedrift av genetiske data og deres forbindelse til kjente sekundære metabolitter. Ulike strategier har også blitt utviklet for å få tilgang til nye bioaktive naturlige produkter (dvs. heterologøst uttrykk, mål gensletting, in vitro rekonstituering, genomsekvens, isotop-guidet screening [genomisotopisk tilnærming], manipulering av lokale og globale regulatorer, resistens målbasert gruvedrift, kulturuavhengig gruvedrift, og, mer nylig, MS-guidet / kode tilnærminger2,6,7,8,9, 10,11,12,13,14,15).
Genomgruvedrift som en enestående strategi krever innsats for å kommentere en enkelt eller liten gruppe molekyler; Dermed forblir hull i prosessen der nye forbindelser prioriteres for isolasjon og strukturbelysning. I prinsippet retter disse tilnærmingene seg bare mot én biosyntetisk vei per eksperiment, og resulterer dermed i en langsom oppdagelsesfrekvens. I denne forstand, ved hjelp av GM sammen med en molekylær nettverk tilnærming representerer et viktig fremskritt for naturlig produktforskning14,15.
Allsidigheten, nøyaktigheten og den høye følsomheten til flytende kromatografi-massespektrometri (LC-MS) gjør det til en god metode for sammensatt identifikasjon. Foreløpig har flere plattformer investert algoritmer og programvaresuiter for umålrettede metabolomikk16,17,18,19,20. Kjernen i disse programmene inkluderer funksjonsdeteksjon (toppplukking)21 og toppjustering, noe som gjør det mulig å matche identiske funksjoner på tvers av en gruppe prøver og søke etter mønstre. MS mønsterbaserte algoritmer22,23 sammenligner karakteristiske fragmenteringsmønstre og samsvarer med MS2-likheter som genererer molekylære familier som deler strukturelle funksjoner. Disse funksjonene kan deretter fremheves og grupperes, og gir muligheten til raskt å oppdage kjente2og ukjente molekyler fra et komplekst biologisk ekstrakt av tandem MS2,24,,25. Derfor er tandem MS en allsidig metode for å få strukturell informasjon om flere kjemotyper som finnes i en stor mengde data samtidig.
Den globale naturlige produkter social molecular networking (GNPS)26 algoritmen bruker normalisert fragment ioner intensitet for å konstruere flerdimensjonale vektorer, der likheter sammenlignes ved hjelp av en cosinusfunksjon. Forholdet mellom ulike overordnede ioner tegnes inn i en diagramrepresentasjon, der hver fragmentering visualiseres som en node (sirkler), og sammenhengen til hver node defineres av en kant (linjer). Den globale visualiseringen av molekyler fra en enkelt kilde er definert som et molekylært nettverk. Strukturelt divergerende molekyler som fragmenterer unikt vil danne sin egen spesifikke klynge eller konstellasjon, mens relaterte molekyler klynger seg sammen. Clustering kjemotyper tillater hypotetisk tilkobling av lignende strukturelle funksjoner til deres biosyntetiske opprinnelse.
Kombinere både kjemotype-til-genotype og genotype-til-kjemotype tilnærminger er kraftig når du oppretter bioinformatikk koblinger mellom BGCs og deres små molekylprodukter27. Derfor er MS-guidet genomgruvedrift en rask metode og lav materialkrevende strategi, og det bidrar til å bygge bro over foreldreioner og biosyntetiske veier avslørt av WGS av en eller flere stammer under ulike metabolske og miljømessige forhold.
Arbeidsflyten til denne protokollen (Figur 1) består av fôring AV WGS-data til en biosyntetisk genklyngemerknadsplattform som antiSMASH28,,29,30. Det bidrar til å estimere mangfoldet av forbindelser og klasse av forbindelser kodet av genomet. En strategi for å målrette en biosyntetisk genklynge som koding en kjemisk enhet av interesse må vedtas, og kulturekstrakter fra en vill type stamme og / eller heterologous stamme som inneholder BGC kan analyseres for å generere grupperte ioner basert på likheter ved hjelp av GNPS26,31. Følgelig er det mulig å identifisere nye molekyler som forbinder med målrettet BGC og er utilgjengeligi databasen (hovedsakelig ukjente analoger, noen ganger produsert i lave titers). Det er relevant å vurdere at brukerne kan bidra til disse plattformene, og at tilgjengeligheten av bioinformatikk og MS/ MS-data øker raskt, og driver til en konstant utvikling og oppgradering av effektive beregningsverktøy og algoritmer for å veilede effektive forbindelser av komplekse ekstrakter med molekyler.
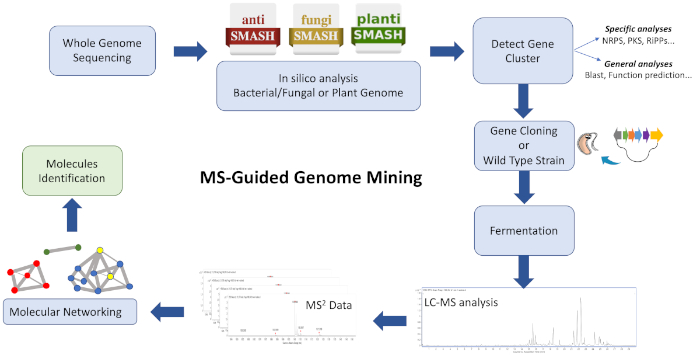
Figur 1: Oversikt over hele arbeidsflyten. Vist er en illustrasjon av bioinformatikk, kloning og molekylære nettverkstrinn involvert i den beskrevne MS-guidede genomgruvetilnærmingen for å identifisere nye metabolitter. Vennligst klikk her for å se en større versjon av denne figuren.
Denne protokollen beskriver en rask og effektiv arbeidsflyt for å kombinere genomgruvedrift og molekylært nettverk som utgangspunkt for den naturlige produktoppdagelsesrørledningen. Selv om mange applikasjoner er i stand til å visualisere sammensetningen og sammenhengen til MS-påviselige molekyler i ett nettverk, er flere vedtatt her for å visualisere strukturelt lignende grupperte molekyler. Ved hjelp av denne strategien, romanen cyclodepsipeptid produkter observert i metabolske ekstrakter av Streptomyces sp. CBMAI 2042 er vellykket identifisert. Guidet av genom gruvedrift, hele biosyntetiske genklynge koding for valinomycins er anerkjent og klonet inn i produsenten stamme Streptomyces coelicolor M1146. Til slutt, etter et MS-mønsterbasert molekylært nettverk, er molekylene som oppdages av MS korrelert med BGCs ansvarlig for deres biogenesis32.
Protocol
Representative Results
Discussion
Den sterkeste fordelen med denne protokollen er dens evne til raskt å degjenskape metabolske profiler og bygge bro genominformasjon med MS-data for å belyse strukturene til nye molekyler, spesielt strukturelle analoger2. Basert på genominformasjon kan ulike fysiske produkter kjemotyper undersøkes, for eksempel polyketider (PK), ikke-ribosomale peptider (NRP) og glykoerte naturlige produkter (GNP), samt kryptiske BGCs. Metabolomic screening bevis på aktiverte BGC-profiler og kjemisk mangfold p…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Den økonomiske støtten til denne studien ble gitt av São Paulo Research Foundation – FAPESP (2019/10564-5, 2014/12727-5 og 2014/50249-8 til L.G.O; 2013/12598-8 og 2015/01013-4 til R.S.; og 2019/08853-9 til C.F.F.A). B.S.P, C.F.F.A., og L.G.O. mottok stipend fra National Council for Scientific and Technological Development – CNPq (205729/2018-5, 162191/2015-4, og 313492/2017-4). L.G.O. er også takknemlig for tilskuddsstøtten fra programmet For Women in Science (2008, Brazilian Edition). Alle forfattere anerkjenner CAPES (Koordinering for forbedring av høyere utdanning personell) for å støtte ettereksamen programmer i Brasil.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
Riferimenti
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

