Масс-спектрометрии-управляемый геном добычи в качестве инструмента для выявления новых природных продуктов
Summary
Здесь создан и описан протокол добычи генома, направленного масс-спектрометрией. Он основан на информации о последовательности генома и анализе LC-MS/MS и направлен на облегчение идентификации молекул из сложных микробных и растительных экстрактов.
Abstract
Химическое пространство, покрытое натуральными продуктами, огромно и широко не признано. Поэтому необходимы удобные методологии для проведения широкомасштабной оценки их функций в природе и потенциальных благ человека (например, для применения лекарственных средств). Этот протокол описывает сочетание добычи генома (ГМ) и молекулярных сетей (MN), двух современных подходов, которые соответствуют генно-кодированным аннотациям во секвенировании всего генома с химическими сигнатурами структуры из грубых метаболических экстрактов. Это первый шаг на пути к открытию новых природных образований. Эти понятия, при применении вместе, определяются здесь как MS-управляемый геном добычи. В этом методе основные компоненты ранее обозначены (с использованием MN), а связанные структурно новые кандидаты связаны с аннотациями последовательности генома (с использованием ГМ). Объединение ГМ и MN является выгодной стратегией для целевой новой молекулы позвоночника или сбора метаболических профилей для того, чтобы определить аналоги из уже известных соединений.
Introduction
Исследования вторичного метаболизма часто состоят из скрининга сырых экстрактов для конкретных биологических видов деятельности с последующим очищением, идентификацией и характеристикой составляющих, принадлежащих к активным фракциям. Этот процесс оказался эффективным, способствуя изоляции нескольких химических организаций. Тем не менее, в настоящее время это рассматривается как неосуществимый, в основном из-за высоких темпов повторного открытия. Поскольку фармацевтическая промышленность произвела революцию, не зная о роли и функциях специализированных метаболитов, их идентификация проводилась в лабораторных условиях, которые не точно представляли природу1. Сегодня лучше понять естественные сигнальные влияния, секрецию и наличие большинства целей при неопределимо низких концентрациях. Кроме того, регулирование этого процесса поможет академическому сообществу и фармацевтической промышленности воспользоваться этими знаниями. Это также пойдет на пользу исследованиям, связанным с прямой изоляцией метаболитов, связанных с молчаливыми биосинтетическими генными кластерами (BGCs)2.
В этом контексте прогресс в области геномного секвенирования возродил интерес к скринингу метаболитов микроорганизмов. Это связано с тем, что анализ геномной информации нераскрытых биосинтетических кластеров может выявить гены, кодирующих новые соединения, не соблюдаемые и не производимые в лабораторных условиях. Многие микробные проекты всего генома или проекты доступны сегодня, и их число растет с каждым годом, обеспечивая огромные перспективы для раскрытия новых биологически активных молекул через геном добычи3,4.
Атлас биосинтетических генных кластеров является текущей крупнейшей коллекцией автоматически добытых генных кластеров в качестве компонента интегрированной платформы микробных геномов Объединенного института генома (JGI IMG-ABC)2. Совсем недавно, Минимальная информация для биосинтетических генных кластеров (MIBiG) Инициатива по стандартизации способствовала ручной реаннотации BGCs, обеспечивая высоко куратором набор справочных данных5. В настоящее время имеется множество инструментов, позволяющих осуществлять вычислительный анализ генетических данных и их связь с известными вторичными метаболитами. Различные стратегии были также разработаны для доступа к новым биологически активным натуральным продуктам (т.е. гетерологическое выражение, удаление целевых генов, восстановление в пробирке, геномная последовательность, изотоп-управляемый скрининг «геномисотемный подход», манипуляции с местными и глобальными регуляторами, добыча на основе сопротивления, культурная независимая добыча полезных ископаемых, и, в последнее время, MS-руководство/код подходы2,6,9,9,9, , 10,11,,12,,13,,14,,15).
Добыча генома как особая стратегия требует усилий по аннотатам одной или небольшой группы молекул; таким образом, сохраняются пробелы в этом процессе, в которых новые соединения отдают приоритет изоляции и разослать структуру. В принципе, эти подходы нацелены только на один биосинтетический путь в эксперимент, что приводит к медленному показателю открытия. В этом смысле использование Gm наряду с молекулярным подходом сетей представляет собой важный шаг для исследования природного продукта14,15.
Универсальность, точность и высокая чувствительность жидкой хроматографии-масс-спектрометрии (LC-MS) делают его хорошим методом для комплексной идентификации. В настоящее время несколько платформ инвестировали алгоритмы и пакеты программного обеспечения для нецелевых метаболомики16,,17,,18,,19,,20. Ядро этих программ включает в себя обнаружение функций (пиковый сбор)21 и пиковое выравнивание, что позволяет совпадать с идентичными функциями по пакету образцов и поиску шаблонов. MS шаблон на основе алгоритмов22,23 сравнить характерные модели фрагментации и матч MS2 сходства генерации молекулярных семейств обмена структурными особенностями. Эти функции могут быть выделены и сгруппированы, предоставляя возможность быстро обнаружить известные и неизвестные молекулы из сложного биологического экстракта тандемом MS2,24,25. Таким образом, тандем MS является универсальным методом получения структурной информации о нескольких хемотипов, содержащихся в большом объеме данных одновременно.
Алгоритм Глобальной естественной молекулярной сети (GNPS)26 использует нормализованную интенсивность ионов фрагментов для построения многомерных векторов, в которых сходства сравниваются с помощью косиновых функций. Взаимосвязь между различными родительскими ионами построена в представлении диаграммы, в котором каждая фрагментация визуализируется как узло (круги), а родственность каждого узла определяется краем (линиями). Глобальная визуализация молекул из одного источника определяется как молекулярная сеть. Структурно расходящиеся молекулы, которые фрагментируют однозначно, образуют свое собственное конкретное скопление или созвездие, в то время как связанные молекулы сгруппируются вместе. Кластеризация хемотипов позволяет гипотетическое соединение аналогичных структурных особенностей с их биосинтетическим происхождением.
Сочетание как хемотипа к генотипу, так и подходов к генотипу к хемотипу является мощным при создании биоинформатичных связей между BGCs и их небольшими молекулярными продуктами27. Таким образом, добыча генома с MS-наведением является быстрым методом и низкой потребляющей материал стратегией, и она помогает преодолеть родительские ионы и биосинтетические пути, выявленные WGS одного или нескольких штаммов в различных метаболических и экологических условиях.
Рабочий процесс этого протокола(рисунок 1) состоит из подачи данных WGS в биосинтетическую платформу аннотации генного кластера, такую как antiSMASH28,,29,30. Это помогает оценить разнообразие соединений и класс соединений, кодируется геномом. Стратегия для целевой биосинтетического генного кластера кодирования химической сущности, представляющие интерес, должны быть приняты, и культура экстракты из штамма дикого типа и / или гетерологовой штамм, содержащий BGC могут быть проанализированы для создания кластерных ионов на основе сходства с помощью GNPS26,31. Следовательно, можно определить новые молекулы, которые ассоциируются с целевым BGC и недоступны в базе данных (в основном неизвестные аналоги, иногда производимые в низких титрах). Уместно учитывать, что пользователи могут вносить свой вклад в эти платформы и что доступность биоинформатики и данных MS/MS быстро растет, что ведет к постоянному развитию и модернизации эффективных вычислительных инструментов и алгоритмов для руководства эффективными соединениями сложных экстрактов с молекулами.
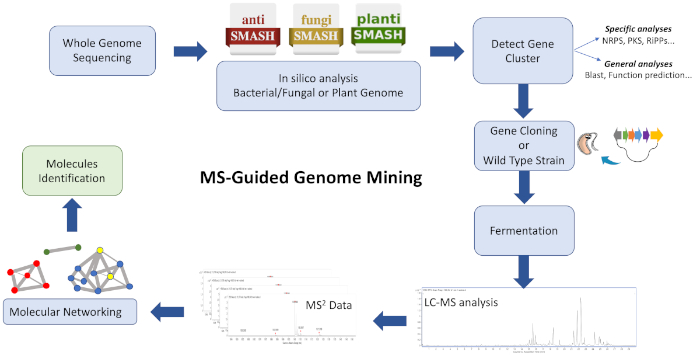
Рисунок 1: Обзор всего рабочего процесса. Показано иллюстрация биоинформатических, клонирующих и молекулярных сетей, участвующих в описанном подходе к добыче генома с гидом MS для выявления новых метаболитов. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Этот протокол описывает быстрый и эффективный рабочий процесс для объединения добычи генома и молекулярных сетей в качестве отправной точки для конвейера по открытию природных продуктов. Хотя многие приложения способны визуализировать состав и родственность MS-обнаруживаемых молекул в одной сети, некоторые из них приняты здесь, чтобы визуализировать структурно похожие кластерные молекулы. Используя эту стратегию, успешно идентифицируются новые циклодепсипептидные продукты, наблюдаемые в метаболических экстрактах streptomyces sp. CBMAI 2042. Руководствуясь добычей генома, весь биосинтетический ген кластер, кодируя для валиномицинов, распознается и клонируется в штамм производителя Streptomyces coelicolor M1146. Наконец, после MS шаблон на основе молекулярной сети, молекулы, обнаруженные MS коррелируют с BGCs ответственность за их биогенеза32.
Protocol
Representative Results
Discussion
Самым сильным преимуществом этого протокола является его способность быстро дереплицировать метаболические профили и мост геномной информации с MS данных для того, чтобы выяснить структуры новых молекул, особенно структурные аналоги2. На основе геномной информации можно …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Финансовая поддержка этого исследования была оказана Сан-Паулу исследовательский фонд – FAPESP (2019/10564-5, 2014/12727-5 и 2014/50249-8 до L.G.O; 2013/12598-8 и 2015/01013-4 до R.S.; и 2019/08853-9 до C.F.F.A). B.S.P., C.F.F.A. и L.G.O. получили стипендии от Национального совета по научно-техническому развитию – CNPq (205729/2018-5, 162191/2015-4 и 313492/2017-4). L.G.O. также благодарна за грантовую поддержку, предоставленную программой для женщин в науке (2008, Бразильское издание). Все авторы признают CAPES (Координация по улучшению высшего образования персонала) для поддержки после окончания программы в Бразилии.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
Riferimenti
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

