Mass pektrometri-guidad genombrytning som ett verktyg för att avslöja nya naturliga produkter
Summary
Här upprättas och beskrivs här ett masspektrometristyrt styrt genombrytningsprotokoll. Den bygger på genomsekvensinformation och LC-MS/MS-analys och syftar till att underlätta identifiering av molekyler från komplexa mikrobiella och växtextrakt.
Abstract
Det kemiska utrymme som täcks av naturliga produkter är enorm och allmänt okända. Därför, praktiska metoder för att utföra omfattande utvärdering av deras funktioner i naturen och potentiella mänskliga fördelar (t.ex. för drug discovery applikationer) önskas. Detta protokoll beskriver kombinationen av genombrytning (GM) och molekylära nätverk (MN), två samtida metoder som matchar genklusterkodade anteckningar i hela genomsekvensering med kemisk struktur signaturer från råa metaboliska extrakt. Detta är det första steget mot upptäckten av nya naturliga enheter. Dessa begrepp definieras här som MS-vägledd genombrytning. I den här metoden har huvudkomponenterna tidigare betecknats (med MN) och strukturellt relaterade nya kandidater associeras med genomsekvensanteckningar (med GM). Kombinera GM och MN är en lönsam strategi för att rikta nya molekyl ben eller skörda metaboliska profiler för att identifiera analoger från redan kända föreningar.
Introduction
Undersökningar av sekundär metabolism består ofta av screening råextrakt för specifika biologiska aktiviteter följt av rening, identifiering och karakterisering av de beståndsdelar som tillhör aktiva fraktioner. Denna process har visat sig vara effektiv och främjat isolering av flera kemiska enheter. Men numera är detta ses som omöjligt, främst på grund av den höga andelen återupptäckt. Eftersom läkemedelsindustrin revolutionerade utan kunskap om specialiserade metaboliters roller och funktioner utfördes deras identifiering under laboratorieförhållanden som inte korrekt representerade naturen1. Idag finns det en bättre förståelse för naturliga signalering influenser, sekretion, och förekomsten av de flesta mål på omätbart låga koncentrationer. Dessutom kommer reglering av processen att hjälpa den akademiska världen och läkemedelsindustrin att dra nytta av denna kunskap. Det kommer också att gynna forskning som omfattar direkt isolering av metaboliter relaterade till tysta biosyntetiska genkluster (BGCs)2.
I detta sammanhang har framsteg inom genomisk sekvensering förnyat intresset för screening av mikroorganismmetaboliter. Detta beror på att analysera genomisk information av avslöjade biosyntetiska kluster kan avslöja gener kodning nya föreningar som inte observerats eller produceras under laboratorieförhållanden. Många mikrobiella hela genom projekt eller utkast finns idag, och antalet växer varje år, vilket ger stora möjligheter att avslöja nya bioaktiva molekyler genom gruvdrift3,4.
Atlas of Biosynthetic Gene Clusters är den nuvarande största samlingen av automatiskt minerade genkluster som en del av den integrerade mikrobiella genomplattformen vid Joint Genome Institute (JGI IMG-ABC)2. Senast har standardiseringsinitiativet för biosyntetiska genkluster (MIBiG) främjat den manuella reannotationen av BGC, vilket ger en mycket kurerad referensdatauppsättning5. Numera finns det gott om verktyg för att möjliggöra beräkningsbrytning av genetiska data och deras anslutning till kända sekundära metaboliter. Olika strategier har också utvecklats för att få tillgång till nya bioaktiva naturprodukter (dvs. heterologuttryck, målgenborttagning, in vitro-reconstitution, genomisk sekvens, isotopstyrd screening [genomisotopmetoden], manipulering av lokala och globala tillsynsmyndigheter, motståndsbaserad gruvdrift, kulturoberoende gruvdrift och, på senare tid, MS-guidad/kod närmar sig2,6,,7,8,9, 10,11,12,13,14,15).
Genombrytning som en enastående strategi kräver insatser för att kommentera en enda eller liten grupp molekyler; Därmed kvarstår luckor i processen där nya föreningar prioriteras för isolering och strukturklarklarering. I princip, dessa metoder mål endast en biosyntetisk väg per experiment, vilket resulterar i en långsam upptäckt takt. I denna mening, med hjälp av GM tillsammans med en molekylär nätverksstrategi utgör ett viktigt framsteg för naturliga produktforskning14,15.
Mångsidigheten, noggrannheten och den höga känsligheten hos flytande kromatografi-masspektrometri (LC-MS) gör det till en bra metod för sammansatt identifiering. För närvarande har flera plattformar investerat algoritmer och programsviter för oriktade metabolomik16,17,18,19,20. Kärnan i dessa program innehåller funktionsdetektering (toppplockning)21 och toppjustering, vilket gör att matchning av identiska funktioner mellan en grupp prover och sökning efter mönster. MS mönsterbaserade algoritmer22,23 jämföra karakteristiska fragmentering mönster och matcha MS2 likheter som genererar molekylära familjer som delar strukturella funktioner. Dessa funktioner kan sedan markeras och grupperas, vilket ger möjlighet att snabbt upptäcka kända och okända molekyler från ett komplext biologiskt extrakt av tandem MS2,24,25. Tandem MS är därför en mångsidig metod för att få strukturell information av flera chemotypes som finns i en stor mängd data samtidigt.
Algoritmen Global Natural Products Social Molecular Networking (GNPS)26 använder den normaliserade fragmentjonintensiteten för att konstruera flerdimensionella vektorer, där likheter jämförs med hjälp av en cosinfunktion. Förhållandet mellan olika överordnade joner ritas i en diagramrepresentation, där varje fragmentering visualiseras som en nod (cirklar) och relateradheten för varje nod definieras av en kant (linjer). Den globala visualiseringen av molekyler från en enda källa definieras som ett molekylärt nätverk. Strukturellt divergerande molekyler som fragmenterar unikt kommer att bilda sin egen specifika kluster eller konstellation, medan relaterade molekyler kluster tillsammans. Klustring chemotypes möjliggör hypotetisk anslutning av liknande strukturella funktioner till deras biosyntetiska ursprung.
Kombinera både chemotype-to-genotyp och genotyp-till-chemotype metoder är kraftfull när du skapar bioinformatik länkar mellan BGCs och deras små molekylprodukter27. Därför är MS-guidad genombrytning en snabb metod och låg materialförbrukande strategi, och det hjälper bro moderjoner och biosyntetiska vägar avslöjas av WGS av en eller flera stammar under olika metaboliska och miljömässiga förhållanden.
Arbetsflödet för detta protokoll (figur 1) består av att mata WGS-data till en biosyntetisk genklusteranteckningsplattform som antiSMASH28,29,30. Det hjälper till att uppskatta olika föreningar och klass av föreningar kodade av genomet. En strategi för att rikta en biosyntetisk gen kluster kodning en kemisk enhet av intresse måste antas, och kultur extrakt från en vild typ stam och / eller heterolog stam som innehåller BGC kan analyseras för att generera klustrade joner baserat på likheter med GNPS26,31. Följaktligen är det möjligt att identifiera nya molekyler som associerar med den riktade BGC och som inte är tillgängliga i databasen (huvudsakligen okända analoger, ibland producerade i lågtiter). Det är relevant att anse att användarna kan bidra till dessa plattformar och att tillgången till bioinformatik och MS/MS-data ökar snabbt, vilket leder till en ständig utveckling och uppgradering av effektiva beräkningsverktyg och algoritmer för att styra effektiva anslutningar av komplexa extrakt med molekyler.
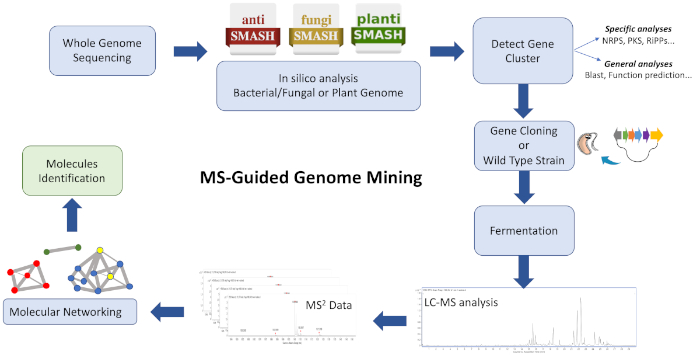
Bild 1: Översikt över hela arbetsflödet. Visas är en illustration av bioinformatik, kloning och molekylära nätverk steg som deltar i den beskrivna MS-guidad genomgruva strategi för att identifiera nya metaboliter. Klicka här för att se en större version av denna siffra.
Detta protokoll beskriver ett snabbt och effektivt arbetsflöde för att kombinera genombrytning och molekylära nätverk som utgångspunkt för den naturliga produktidentifieringspipelinen. Även om många applikationer kan visualisera sammansättningen och släktskapen hos MS-detekterbara molekyler i ett nätverk, antas flera här för att visualisera strukturellt liknande klustrade molekyler. Med hjälp av denna strategi, nya cyclodepsipeptide produkter som observerats i metaboliska extrakt av Streptomyces sp. CBMAI 2042 identifieras framgångsrikt. Guidad av genombrytning, hela biosyntetiska genkluster kodning för valinomycins är erkänd och klonas i producenten stam Streptomyces coelicolor M1146. Slutligen, efter en MS mönster-baserade molekylära nätverk, de molekyler som upptäckts av MS är korrelerade med BGCs ansvarar för deras biogenesis32.
Protocol
Representative Results
Discussion
Den starkaste fördelen med detta protokoll är dess förmåga att snabbt dereplikera metaboliska profiler och bro genomisk information med MS-data för att belysa strukturerna hos nya molekyler, särskilt strukturella analoger2. Baserat på genomisk information kan olika naturprodukter undersökas, såsom polyketider (PK), nonribosomala peptider (NRP) och glykoserade naturprodukter (BNP), samt kryptiska BGCs. Metabolomisk screening ger bevis på aktiverade BGC-profiler och kemisk mångfald som pr…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Det ekonomiska stödet till denna studie tillhandahölls av São Paulo Research Foundation – FAPESP (2019/10564-5, 2014/12727-5 och 2014/50249-8 till L.G.O; 2013/12598-8 och 2015/01013-4 till R.S.; och 2019/08853-9 till C.F.F.A). B.S.P, C.F.F.A., och L.G.O. fick stipendier från Nationella rådet för vetenskaplig och teknisk utveckling – CNPq (205729/2018-5, 162191/2015-4 och 313492/2017-4). L.G.O. är också tacksam för det bidragsstöd som programmet For Women in Science (2008, Brazilian Edition). Alla författare erkänner CAPES (Samordning för förbättring av högre utbildning personal) för att stödja efter examen program i Brasilien.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
Riferimenti
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

