Yeni Doğal Ürünleri Ortaya Çıkarmak İçin Bir Araç Olarak Kütle Spektrometresi Güdümlü Genom Madenciliği
Summary
Burada bir kütle spektrometresi güdümlü genom madenciliği protokolü oluşturulmuş ve tanımlanmıştır. Genom dizilimi bilgileri ve LC-MS/MS analizine dayanan bu analiz, kompleks mikrobiyal ve bitki özlerinden moleküllerin belirlenmesini kolaylaştırmayı amaçlamaktadır.
Abstract
Doğal ürünlerin kapsadığı kimyasal alan çok büyüktür ve yaygın olarak tanınmaz. Bu nedenle, doğadaki işlevlerinin ve potansiyel insani yararlarının (örneğin, uyuşturucu keşif uygulamaları için) geniş kapsamlı bir şekilde değerlendirilmesi için uygun metodolojiler isteilmektedir. Bu protokol, genom madenciliği (GM) ve moleküler ağ (MN) kombinasyonunu tanımlar, bu yaklaşım, tüm genom dizilimindeki gen küme kodlu ek açıklamaları ile metabolik ham ekstrelerin kimyasal yapı imzalarıyla eşleşen iki çağdaş yaklaşımdır. Bu, yeni doğal varlıkların keşfine giden ilk adımdır. Bu kavramlar, birlikte uygulandığında, burada MS güdümlü genom madenciliği olarak tanımlanır. Bu yöntemde, ana bileşenler daha önce belirlenmiş (MN kullanılarak) ve yapısal olarak ilişkili yeni adaylar genom dizisi ek açıklamaları (GM kullanılarak) ile ilişkilidir. GM ve MN’nin birleştirilmesi, zaten bilinen bileşiklerden analogları belirlemek için yeni molekül omurgalarını hedeflemek veya metabolik profilleri toplamak için karlı bir stratejidir.
Introduction
Sekonder metabolizmanın araştırılması genellikle belirli biyolojik faaliyetler için ham ekstrelerin taranması ve ardından aktif fraksiyonlara ait bileşenlerin saflaştırılması, tanımlanması ve karakterizasyonundan oluşur. Bu süreç, çeşitli kimyasal varlıkların izolasyon teşvik verimli olduğu kanıtlanmıştır. Ancak, günümüzde bu, özellikle yeniden keşif yüksek oranları nedeniyle mümkün olarak görülüyor. İlaç endüstrisi, özel metabolitlerin rol ve işlevleri hakkında bilgi sahibi olmadan devrim yarattıkça, bunların tanımlanması doğayı tam olarak temsil eden laboratuvar koşulları altında gerçekleştirilmiştir1. Bugün, doğal sinyal etkileri, salgılanması ve tespit edilemeyen düşük konsantrasyonlarda çoğu hedefin varlığı daha iyi bir anlayış vardır. Ayrıca, sürecin düzenlenmesi akademik topluluğun ve ilaç endüstrisinin bu bilgiden yararlanabilmelerine yardımcı olacaktır. Ayrıca sessiz biyosentetik gen kümeleri (BGCs)2ile ilgili metabolitlerin doğrudan izolasyon içeren araştırma yararlanacaktır.
Bu bağlamda genomik dizilimdeki gelişmeler mikroorganizma metabolitlerinin taranmasına olan ilgiyi yenilemidir. Bunun nedeni, ortaya çıkarılan biyosentetik kümelerin genomik bilgilerinin analiz edilmesi, laboratuvar koşullarında gözlemlenmeyen veya üretilmemiş yeni bileşikleri kodlayan genleri ortaya çıkarabilebilir. Birçok mikrobiyal bütün genom projeleri veya taslakları bugün mevcuttur, ve sayısı her yıl artıyor, genom madenciliği yoluyla yeni biyoaktif molekülleri ortaya çıkarmak için büyük umutları sağlayan3,4.
Biyosentetik Gen Kümeleri Atlası, Ortak Genom Enstitüsü (JGI IMG-ABC)2Entegre Mikrobiyal Genom Platformu’nun bir bileşeni olarak otomatik olarak çıkarılan gen kümelerinin mevcut en büyük koleksiyonudur. En son, Biyosentetik Gen Kümeleri için Minimum Bilgi (MIBiG) Standardizasyon Girişimi, son derece küratörlü referans dataset5sağlayarak, BGCs manuel reannotation teşvik etmiştir. Günümüzde, genetik verilerin hesaplamalı madenciliğini ve bunların bilinen ikincil metabolitlerle bağlantısını sağlamak için birçok araç mevcuttur. Farklı stratejiler de yeni biyoaktif doğal ürünlere erişmek için geliştirilmiştir (yani, heterolog ifade, hedef gen silme, in vitro reconstitution, genomik dizi, izotop güdümlü tarama [genomistopik yaklaşım], yerel ve küresel düzenleyicilerin manipülasyonu, direnç hedef tabanlı madencilik, kültür bağımsız madencilik, ve daha yakın zamanda, MS güdümlü / kodyaklaşımları 2,6,7,8,9, 10,11,12,13,14,15).
Tek bir strateji olarak genom madenciliği, tek veya küçük bir molekül grubuna açıklama yapmak için çaba gerektirir; böylece, izolasyon ve yapı açıklanma için yeni bileşiklerin öncelik lendirildiği süreçteki boşluklar devam etmektedir. Prensip olarak, bu yaklaşımlar deney başına sadece bir biyosentetik yolu hedefleyerek yavaş bir keşif hızına yol açacaktır. Bu anlamda, moleküler ağ yaklaşımı ile birlikte GM kullanarak doğal ürün araştırma için önemli bir ilerleme temsil eder14,15.
Sıvı kromatografi-kütle spektrometresinin (LC-MS) çok yönlülüğü, doğruluğu ve yüksek hassasiyeti onu bileşik tanımlama için iyi bir yöntem haline getirin. Şu anda, çeşitli platformlar hedeflenmemiş metabolomics16, 17,18,,1919,20için algoritmalar ve yazılım paketleri yatırım yaptık. Bu programların çekirdeği, bir dizi örnek arasında aynı özelliklerin eşleşmesine ve desenlerin aranmasına olanak tanıyan özellik algılama (tepe toplama)21 ve tepe hizalama içerir. MS desen tabanlı algoritmalar22,23 karakteristik parçalanma desenleri karşılaştırın ve yapısal özellikleri paylaşan moleküler aileler üreten MS2 benzerlikler maç. Bu özellikler daha sonra vurgulanmış ve kümelenmiş olabilir, hızla tandem MS2tarafından karmaşık bir biyolojik ekstresi bilinen ve bilinmeyen molekülleri keşfetmek için yeteneği conferring,24,25. Bu nedenle, tandem MS aynı anda büyük miktarda veri bulunan çeşitli kemotiplerin yapısal bilgi elde etmek için çok yönlü bir yöntemdir.
Global Natural Products Social Molecular Networking (GNPS)26 algoritması, benzerliklerin bir kosinüs fonksiyonu kullanılarak karşılaştırıldığı çok boyutlu vektörler oluşturmak için normalleştirilmiş parça iyonyoğunluğunu kullanır. Farklı üst iyonlar arasındaki ilişki, her parçalanmanın bir düğüm (daire) olarak görüntülendiği ve her düğümün ilişkililiğinin bir kenar (çizgiler) ile tanımlandığı bir diyagram gösteriminde çizilir. Moleküllerin tek bir kaynaktan küresel görselleştirilmesi moleküler ağ olarak tanımlanır. Yapısal olarak farklı moleküller bu parçayı benzersiz olarak oluştururken, ilgili moleküller birlikte kümelenirler. Kümeleme chemotypes biyosentetik kökenleri benzer yapısal özelliklerivarsayımsal bağlantı sağlar.
Hem kemotip-to-genotip ve genotip-to-chemotype yaklaşımları birleştirerek BGCs ve küçük molekül ürünleri arasında biyoinformatik bağlantılar oluştururken güçlüdür27. Bu nedenle, MS güdümlü genom madenciliği hızlı bir yöntem ve düşük malzeme tüketen bir stratejidir ve farklı metabolik ve çevresel koşullar altında bir veya daha fazla suşwgs tarafından ortaya ebeveyn iyonları ve biyosentetik yollar köprü yardımcı olur.
Bu protokolün iş akışı(Şekil 1)WGS verilerinin antiSMASH28,29,30gibi biyosentetik gen kümesi açıklama platformuna aktarılmasından oluşur. Genom tarafından kodlanmış bileşiklerin çeşitliliğinin ve sınıfının tahmin edilebilen bir şekilde tahmin edilebilen bir yapıya yardımcı olur. İlgi kimyasal bir varlık kodlama bir biyosentetik gen kümesi hedef bir strateji kabul edilmelidir ve BGC içeren vahşi bir tür gerginlik ve / veya heterolog suşu kültür özleri GNPS26kullanarak benzerliklere dayalı kümelenmiş iyonlar oluşturmak için analiz edilebilir,31. Sonuç olarak, hedeflenen BGC ile ilişkilendirmek ve veritabanında kullanılamaz yeni molekülleri belirlemek mümkündür (özellikle bilinmeyen analogları, bazen düşük titreleri üretilen). Kullanıcıların bu platformlara katkıda bulunabileceğini ve biyoinformatik ve MS/MS verilerinin kullanılabilirliğinin hızla arttığını ve karmaşık özlerin moleküllerle verimli bağlantılarını yönlendirmek için etkili hesaplama araçları nın ve algoritmalarının sürekli olarak geliştirilmesine ve yükseltilmesine yol açmaktadır.
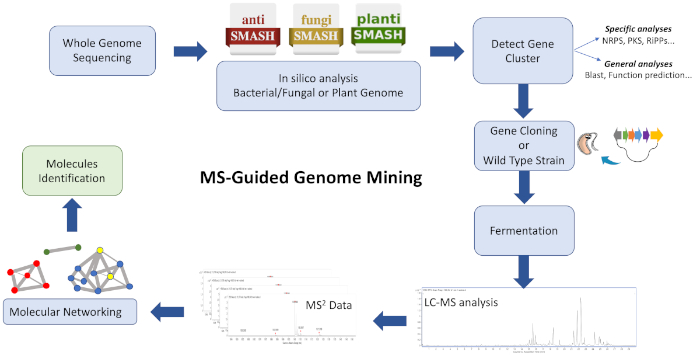
Şekil 1: Tüm iş akışına genel bakış. Gösterilen biyoinformatik bir örnektir, klonlama, ve moleküler ağ adımları tanımlanan MS güdümlü genom madenciliği yaklaşımında yeni metabolitleri tanımlamak için yer. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Bu protokol, genom madenciliği ve moleküler ağ lağımını doğal ürün keşif boru hattı için başlangıç noktası olarak birleştirmek için hızlı ve verimli bir iş akışını tanımlar. Birçok uygulama bir ağda MS-algılanabilir moleküllerin bileşimi ve ilgililiğini görselleştirmek mümkün olmasına rağmen, birkaç yapısal olarak benzer kümelenmiş molekülleri görselleştirmek için burada kabul edilmiştir. Bu strateji kullanılarak Streptomyces sp. CBMAI 2042 metabolik ekstrelerinde gözlenen yeni siklopsipeptid ürünleri başarıyla tanımlanmıştır. Genom madenciliği rehberliğinde, valinomycins için tüm biyosentetik gen küme kodlama tanınan ve üretici suşu Streptomyces coelicolor M1146 içine klonlanmış. Son olarak, MS desen tabanlı moleküler ağ lama sonrasında, MS tarafından saptanan moleküller biyogenez32’densorumlu BgC’lerle ilişkilidir.
Protocol
Representative Results
Discussion
Bu protokolün en güçlü avantajı, yeni moleküllerin, özellikle yapısal analogların yapılarını açıklığa kavuşturmak için metabolik profilleri hızla çoğaltma ve genomik bilgileri MS verileriyle birbirine bağlama yeteneğidir2. Genomik bilgilere dayanarak, polikeltidler (PK), nonribosomal peptidler (NRP) ve glikoziküllü doğal ürünler (GNP) gibi farklı doğal ürünler kemotipleri ve şifreli BGC’ler incelenebilir. Böylece, bir BGC moleküler ağ tarafından keşfedilen ben…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Bu çalışmanın mali desteği São Paulo Araştırma Vakfı – FAPESP (2019/10564-5, 2014/12727-5 ve 2014/50249-8′ den L.G.O’ya; 2013/12598-8 ve 2015/01013-4 r.s.; ve 2019/08853-9’dan C.F.F.A.’ya). B.S.P, C.F.F.A., ve L.G.O. Ulusal Bilimsel ve Teknolojik Gelişim Konseyi’nden burs aldı – CNPq (205729/2018-5, 162191/2015-4 ve 313492/2017-4). L.G.O. ayrıca Women in Science (2008, Brazilian Edition) programı tarafından sağlanan hibe desteği için minnettardır. Tüm yazarlar Capes (Yüksek Öğretim Personelinin İyileştirilmesi Için Koordinasyon) Brezilya’da mezuniyet sonrası programları desteklemek için kabul.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
Riferimenti
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

