ترجمة تنقية التقارب الريبوسم (TRAP) للتحقيق في تطوير جذر Arabidopsis thaliana على مقياس نوع الخلية الخاصة
Summary
ترجمة تنقية تقارب الريبوسوم (TRAP) يوفر إمكانية تشريح البرامج التنموية مع الحد الأدنى من معالجة الأعضاء والأنسجة. ينتج البروتوكول عن الحمض النووي الريبي عالي الجودة من الخلايا المستهدفة ببروتين فلورسنت أخضر (GFP) يسمى وحدة فرعية من الريبوزيوم. تكشف أدوات التحليل المصب، مثل qRT-PCR أو RNA-seq، عن ملفات تعريف التعبير الخاصة بنوع الأنسجة والخلايا.
Abstract
في هذه المقالة، نعطي تعليمات عملية للحصول على بيانات الترجمة من أنواع مختلفة من الخلايا الجذرية العربية dopsis thaliana عبر ترجمة التنقية تقارب الريبوسر (TRAP) ومتتالية الأمثل إعداد مكتبة منخفضة الإدخال.
كمادة البدء، ونحن توظيف خطوط النبات التي تعبر عن GFP الموسومة بروتين riبومRPL18 بطريقة محددة نوع الخلية عن طريق استخدام المروجين كافية. قبل المناعة واستخراج الحمض النووي الريبي ، يتم تجميد الأنسجة ، مما يحافظ على سلامة الأنسجة ويسمح في الوقت نفسه بتنفيذ دراسات السلسلة الزمنية بدقة زمنية عالية. وتجدر الإشارة إلى أن هياكل جدار الخلية لا تزال سليمة، وهو عيب رئيسي في الإجراءات البديلة مثل الأساليب القائمة على فرز الخلايا المنشطة للفلورسينس والتي تعتمد على الأنسجة المصممة لعزل مجموعات الخلايا المتميزة. بالإضافة إلى ذلك ، لا يلزم تثبيت الأنسجة كما هو الحال في تقنيات التقاط الميكروديزالقسمة بالليزر ، والتي تسمح بالحصول على الحمض النووي الريبي عالي الجودة.
ومع ذلك، فإن أخذ العينات من المجموعات الفرعية للخلايا وعزل الحمض النووي الريبي المرتبط بالبوليبعض فقط يحد بشدة من غلة الحمض النووي الريبي. ولذلك، من الضروري تطبيق أساليب إعداد المكتبة الحساسة بما فيه الكفاية من أجل الحصول الناجح على البيانات من قبل RNA-seq.
يقدم TRAP أداة مثالية لأبحاث النبات حيث أن العديد من العمليات التنموية تنطوي على مسارات إشارات ميكانيكية ذات صلة بجدار الخلية. استخدام المروجين لاستهداف مجموعات محددة من الخلايا هو سد الفجوة بين الجهاز ومستوى الخلية الواحدة التي تعاني بدورها من دقة قليلة أو تكاليف عالية جدا. هنا، نطبق TRAP لدراسة الاتصالات الخلية الخلية في تشكيل الجذر الجانبي.
Introduction
وبدافع من التطبيق المتزايد لتقنيات التسلسل من الجيل التالي، يمكن زيادة الاستبانة المكانية في البيولوجيا الإنمائية. تهدف الدراسات المعاصرة إلى تشريح الأنسجة وصولاً إلى أنواع الخلايا المتخصصة، إن لم يكن بخلية واحدة المستوى1،,2،,3،,4. ولتحقيق هذه الغاية، تم ابتكار عدد كبير من الطرق المختلفة على مدى السنوات الخمسين الماضية (انظر الشكل 1A)5،6،7،8،9،10،11،12،13،14،15.
وقد تم العديد من الأدوات في علم النبات التكيف من التقنيات التي كانت رائدة في مجال البحوث الحيوانية. وليس هذا هو الحال بالنسبة للطريقة التي نقدمها بالتفصيل هنا. في عام 2005، مجهزة خلفية قوية في ترجمة البروتين، مختبر بيلي سيريس المنصوص عليها لهندسة البروتينات riبوم لتنقية تقارب لاحقة16. وهكذا، فإنها يمكن أن تجنب التنميط متعدد السمات تستغرق وقتا طويلا وكثيفة العمالة، والذي يقوم على ultracentrifugation مع تدرج السكروز وكان يستخدم لتقييم ترجمة الريبوسوم منذ 1960s17،18. ومنذ ذلك الحين يشار إلى الأسلوب على أنه تنقية تقارب الريبوسرية الانتقالية (TRAP)16. بعد نجاح دراسات الترجمة في النباتات، Heiman وآخرون تكييفها TRAP للحيوانات19 وغيرها وسعت تطبيقها إلى الخميرة20، Drosophila21، زينوبوس22 وحمار وحشي23،24.
على الرغم من أن التعديل الجيني للنظام النموذجي هو شرط أساسي لـ TRAP ، الذي يحد من تطبيقه على الأنواع القابلة للتحول الوراثي ، يمكن للمرء في الوقت نفسه تسخير هذا الاعتراض لاستهداف مجموعات فرعية من الخلايا ذات أهمية خاصة ويصعب للغاية عزلها عن الأنسجة السليمة / الجهاز25 (على سبيل المثال ، الخلايا التشفية المتفرعة للغاية في دماغ فأر أو الهيفاي الفطري في الأنسجة النباتية المصابة). في النباتات ، يتم الاحتفاظ بجميع الخلايا في مكانها عبر جدران الخلايا التي تشكل أساس الهيكل العظمي الهيدروستاتيكي26. لتحرير خلية نباتية من هذه المصفوفة ، قام العلماء إما بقطع الخلية فعليًا من الأنسجة المحيطة بها من خلال إجراء عملية إزالة الميكروبات بالليزر (LCM)27 أو إجراء هضم أنزيمي لجدران الخلية28. من بين الخلايا الأخيرة، ما يسمى protoplasts، يتم تسمية السكان من الفائدة الفلورسنت ويمكن فصلها عن طريق فرز الخلايا المنشط ة الفلورية (FACS)7. LCM يتطلب عادة عينة لتكون ثابتة وجزءا لا يتجزأ من الشمع، مما يؤدي في نهاية المطاف إلى تدهور نوعية الحمض النووي الريبي29. الأساليب المستندة إلى FACS تنتج الحمض النووي الريبي عالي الجودة ، ولكن عملية protoplasting نفسها تدخل اختلافات في التعبير الجيني30 والأنسجة ذات جدران الخلايا الثانوية المعدلة والسميكة من الصعب علاجها. وعلاوة على ذلك، يفترض العديد من العمليات التنموية في النباتات أن تعتمد على إشارات تنتقل ميكانيكيا، وبالتالي سلامة جدار الخلية ذات أهمية قصوى31. طريقتان، تستخدمان اختصاراً للتحايل على عزل الخلايا من خلال العمل على مستوى النوى، هما الفرز النووي المنشط للفلورسينس (FANS) وعزل النوى الموسومة بأنواع خلايا محددة (INTACT). كما هو الحال في TRAP ، فإنها تستخدم المروجين نوع الخلية الخاصة لوضع علامة على النوى ، التي تحصل في وقت لاحق إثراء عن طريق الفرز أو سحب إلى أسفل ، على التوالي8،15. التحدي الرئيسي لجميع هذه النهج هو الحصول على ما يكفي من المواد الحمض النووي الريبي من مجموعات فرعية من الخلايا في الأنسجة. كما يلتقط TRAP سوى جزء صغير من الحمض النووي الريبي الخلوي، جمع عينة هو عنق الزجاجة كبيرة. ولذلك، هناك حاجة إلى بروتوكولات إعداد المكتبة الحساسة بشكل خاص لإنتاج بيانات عالية الجودة من كميات مدخلات منخفضة.
منذ إنشائها، وقد استخدمت TRAP إما في تركيبة مع microarrays الحمض النووي أو، كما انخفضت تكاليف التسلسل بشكل كبير في السنوات الأخيرة، RNA-seq10،32،33. وقد تم بالفعل توضيح العديد من الأسئلة البحثية كما تمت مراجعتها في Sablok et al.34. ونحن مقتنعون بأن المزيد من التقارير ستتبع في السنوات المقبلة كما تقنية متعددة جدا عند الجمع بين المروجين مختلفة لاستهداف أنواع محددة من الخلايا. في نهاية المطاف، سيتم ذلك حتى بطريقة لا يمكن اختزالها، ويمكن دمجها مع التحقيق في رد فعل النبات على العديد من عوامل الإجهاد الحيوي واللاأحيائي. بالإضافة إلى ذلك ، حيث لا تتوفر خطوط معدلة مستقرة ، تم استخدام أنظمة التعبير عن الجذر المشعر بنجاح أيضًا لتنفيذ TRAP في الطماطم وmedicago35،36.
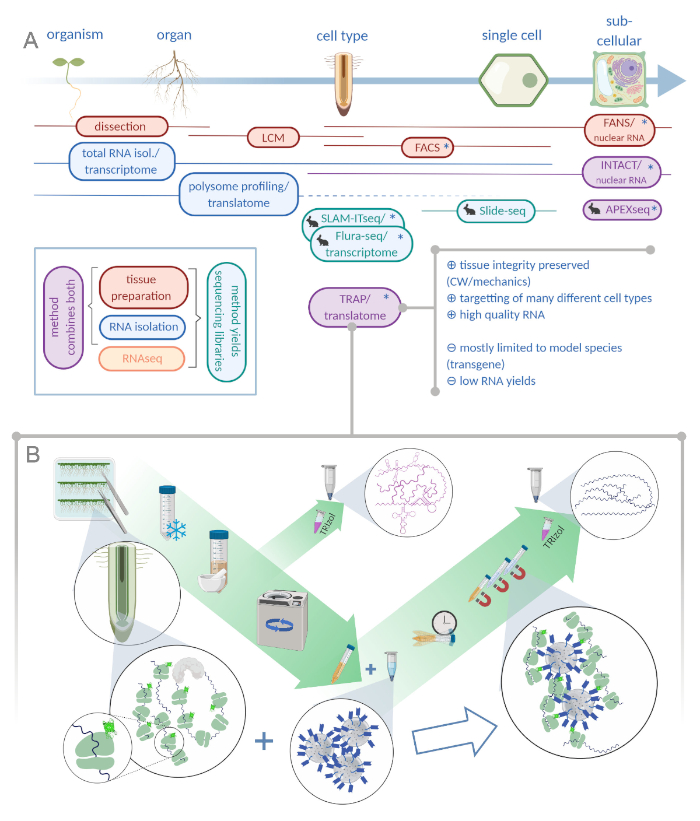
الشكل 1: ترجمة تنقية التقارب الريبوسومي (TRAP) تكمل محفظة تحليل “omics”. A. يمكن تحقيق زيادة مستويات الدقة التحليلية، وصولا إلى وحدة واحدة أو حتى دقة تحت الخلية من خلال عدد كبير من الأساليب أو تركيبات منها. ويقدم هذا المخطط لمحة عامة عن الأدوات المتاحة حاليا في المجال النباتي والحيواني. يمكن تحقيق جمع الأنسجة في دقة الخلوية من خلال بروتوكولات مثل LCM أو FACS ، والتي يتم اقترانها بعد ذلك بالنسخ القياسي أو تحليل التنميط / الترجمة المتعدد. TRAP وسليمة دمج كل من التقاط الأنسجة وعزل الحمض النووي الريبي لأنها تقوم على العلامات epitope. ومع ذلك ، عينات سليمة نواة الخلية فقط ويشكل ، وبالتالي ، حالة خاصة من تحليل النسخ. رمز أرنب صغير علامات الأساليب المطورة حديثا في مجال الحيوان: في حين أن SLAM-ITseq وFlura-seq تعتمد على التتار الأيضي من الحمض النووي الريبي الوليد مع قواعد uracil المعدلة في الخلايا التي تعبر عن الإنزيم المتساهل، الشريحة-seq يجعل استخدام شريحة الزجاج المغلفة مع الباركود الحمض النووي التي توفر المعلومات الموضعية في النطاق الخلوي. ويتبع نهج وضع علامات القرب في APEX-seq لأخذ عينات RNAs في مقصورات دون خلوية محددة. وتجدر الإشارة إلى أن زيادة الاستبانة كثيرا ما تتطلب توليد مواد معدلة وراثيا (علامات نجمية) ومن ثم تستخدم هذه الأساليب في الغالب للأنواع النموذجية. TRAP مناسب بشكل خاص لدراسات علوم النبات التي تنطوي على جدار الخلايا (CW) أو إشارات ميكانيكية وكذلك أنواع الخلايا التي يصعب إطلاقها من مصفوفة CW الخاصة بهم. ب. مفصلة الخطوات الرطب مختبر الإجراء TRAP: تزرع الشتلات التي تعبر عن بروتين riبوم الموسومة GFP في أنواع الخلايا المتميزة (مثل endodermis الجذر) على أطباق بيتري لمدة سبعة أيام والمواد الجذرية التي يتم حصادها عن طريق تجميد المفاجئة. يتم جمع عينة التحكم في الحمض النووي الريبي بالكامل من مستخلص النفط الخام المتجانس قبل أن يُسقط الحطام عن طريق الطرد المركزي. يتم إضافة الخرز المغناطيسي المضاد لـ GFP إلى المستخلص المنفّر لإجراء الترسيبات المناعي. بعد الحضانة وثلاث خطوات غسل ، يتم الحصول على الحمض النووي الريبي المرتبط بالبولي بعض (TRAP / POLYsome RNA) مباشرة عن طريق استخراج الفينول الكلوروفورم. LCM: الليزر التقاط microdissection، FACS / FANS: خلية تنشيط الفلورسينس / الفرز النووي، APEX-seq: طريقة تقوم على ascorbate peroxidase المهندسة، سليمة: عزل النوى الموسومة في أنواع خلايا محددة، SLAM-ITseq: thiol (SH) المرتبطة alkylation لتسلسل التمثيل الغذائي من الجيش الملكي النيبالي في الأنسجة، فلورا-seq: فلوروراسيل المسمى تسلسل الحمض النووي الريبي (خلق مع Biorender.com) يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الهدف من هذه المقالة هو توفير وصف مفصل لأسلوب TRAP، لتسليط الضوء على الخطوات الهامة وتوفير التوجيه لأسلوب إعداد مكتبة ممكن.
وستتألف تجربة TRAP العامة أساسا من الخطوات التالية (انظر أيضا الشكل 1باء):(1) إعداد المواد النباتية بما في ذلك استنساخ بناء العلامات الريبوسومية، وإنتاج الخطوط المحورة وراثيا واختيارها، وتزايد البذور والطلاء وستكثرها، وتطبيق/معالجة الإجهاد (اختياري) وحصاد الأنسجة؛ (2) إعداد المواد النباتية بما في ذلك استنساخ الريبوسوم يُحدّد البناء، وإنتاج الخطوط المحورة وراثيا واختيارها، وزراعة البذور والطلاء وستكثرها، وتطبيق/معالجة الإجهاد (اختياري) وحصاد الأنسجة؛ (2) إعداد المواد النباتية بما في ذلك استنساخ الريبوسوم لوضع العلامات على الخلايا. (2) التنقية المناعية بما في ذلك تجانس الأنسجة وتطهير مستخلص النفط الخام، وغسل النحل وتنقية المناعة، وخطوات الغسيل؛ (3) استخراج الحمض النووي الريبي وتقييم الجودة؛ و(4) إعداد المكتبة.
وقد جذر Arabidopsis نظام نموذجي لدراسة تطوير النبات منذ عرضه كمصنع نموذجي37,38. هنا ، يتم عرض تطبيق TRAP في سياق تطوير الجذر الجانبي النباتي. في النباتات ، يعتمد تراكم نظام الجذر بأكمله على تنفيذ هذا البرنامج وبالتالي فهو مهم جدًا لبقاء الكائن الحي39. في Arabidopsis، تنشأ الجذور الجانبية من أنسجة الدراجة المحيطة التي تتواجد بجوار أوعية xylem وبالتالي يطلق عليها اسم xylem pole pericycle (XPP ؛ انظر الشكل 2C)40. بعض خلايا XPP ، والتي تقع في عمق الجذر ، والحصول على هوية الخلية مؤسس ، وعلى الزناد الهرمونية المحلية ، والبدء في الانتشار عن طريق تورم وتقسيم anticlinally41. ومع ذلك ، بسبب وجود مصفوفة جدار الخلية جامدة ، وهذه العملية تمارس الإجهاد الميكانيكي على الأنسجة المحيطة بها. على وجه الخصوص ، يتأثر endodermis التجاوزي ، كما هو الحال في طريق محور نمو الجذر الجانبي42،43،44. في الواقع ، فإن تشكيل بريمورديوم حديثا يجب أن تنمو من خلال الخلية endodermis المبالغ فيها(الشكل 2C2)في حين يتم دفع الخلايا القشرة والبشرة جانبا لprimordium لتظهر أخيرا45،46. وقد أظهرت الأعمال الأخيرة في مختبرنا أن endodermis يساهم بنشاط لاستيعاب انتشار في pericycle. حظر المستهدفة من الإشارات الهرمونية endodermal كافية لمنع حتى الانقسام الأول جدا في خلايا XPP47. وبالتالي، الاتصالات pericycle-endodermis يشكل نقطة تفتيش مبكرة جدا لتطوير الجذر الجانبي في Arabidopsis. ومع ذلك، لا يُعرف كيف يتم تنفيذ هذا الكلام المتقاطع. لكشف هذا اللغز، اخترنا نهج TRAP-seq لاستهداف XPP والخلايا endodermal. لإثراء للخلايا في برنامج الجذر الجانبي، ونحن تحاكي الزناد الهرموني عن طريق تطبيق خارجي تناظرية الأوكسين (1-حمض النفتاليناستيك، NAA)48،والتي سمحت في الوقت نفسه لحل مؤقتا المرحلة الأولية من تشكيل الجذر الجانبي.
Protocol
Representative Results
Discussion
التحقق من نمط تعريب RPL18
حاسمة لتجنب سوء تفسير البيانات من أي تجربة TRAP هو نمط التعبير السليم للوحدة الفرعية riبومال الموسومة. لذلك ، فإن دمج GFP كعلامة epitope إلى RPL18 يسمح بأناقة كبيرة بالتحقق من نمط التعبير المطلوب وعلى التوالي ، والسحب من الكسر المتعدد الأجزاء من نفس الأنسجة. يتبع جي?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
ونود أن نشكر جان كلود والسر من مركز التنوع الوراثي في زيوريخ على مشورة الخبراء الحاسمة في المرحلة المبكرة من هذا المشروع. تم دعم العمل في مختبر فيرمير من خلال منحة الأستاذية SNF (PP00P3_157524) ومنحة معدات R’EQUIP (316030_164086) من المؤسسة الوطنية السويسرية للعلوم (SNSF) الممنوحة لـ JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
Riferimenti
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

