Het vertalen van Ribosome Affinity Purification (TRAP) om Arabidopsis thaliana Root Development te onderzoeken op een cell type-specifieke schaal
Summary
Het vertalen van ribosoomaffiniteitszuivering (TRAP) biedt de mogelijkheid om ontwikkelingsprogramma’s te ontleden met minimale verwerking van organen en weefsels. Het protocol levert hoogwaardig RNA op uit cellen die zijn gericht op een groene fluorescerende eiwit (GFP)-gelabelde ribosomalsubunit. Downstream analysetools, zoals qRT-PCR of RNA-seq, onthullen weefsel- en celtypespecifieke expressieprofielen.
Abstract
In dit artikel geven we hands-on instructies om translationoom gegevens te verkrijgen van verschillende Arabidopsis thaliana wortelceltypes via de vertaalribomasoom affinity purification (TRAP) methode en opeenvolgende geoptimaliseerde low-input bibliotheek voorbereiding.
Als uitgangsmateriaal maken we gebruik van plantlijnen die GFP-gelabelde ribosomal eïne RPL18 op een celtype-specifieke manier uitdrukken door gebruik te maken van adequate promotors. Voorafgaand aan immunozuivering en RNA-extractie wordt het weefsel vastgemaakt, wat de weefselintegriteit behoudt en tegelijkertijd de uitvoering van tijdreeksstudies met een hoge temporele resolutie mogelijk maakt. Met name celwandstructuren blijven intact, wat een groot nadeel is in alternatieve procedures zoals op fluorescentie geactiveerde celsorteermethoden die afhankelijk zijn van weefselprotoplasting om verschillende celpopulaties te isoleren. Bovendien is er geen weefselfixatie nodig zoals in lasercapture op microdissectie gebaseerde technieken, waardoor rna van hoge kwaliteit kan worden verkregen.
Echter, bemonstering van subpopulaties van cellen en alleen isoleren polysome-geassocieerdRNA ernstig beperkt RNA opbrengsten. Het is daarom noodzakelijk om voldoende gevoelige bibliotheekbereidingsmethoden toe te passen voor succesvolle gegevensverwerving door RNA-seq.
TRAP biedt een ideaal hulpmiddel voor plantenonderzoek, omdat veel ontwikkelingsprocessen celwandgerelateerde en mechanische signaleringstrajecten omvatten. Het gebruik van promotors om specifieke celpopulaties te richten overbrugt de kloof tussen orgaan- en eencellige niveau dat op zijn beurt te lijden heeft onder weinig resolutie of zeer hoge kosten. Hier passen we TRAP toe om celcelcommunicatie te bestuderen in laterale wortelvorming.
Introduction
Gedreven door de toenemende toepassing van next-generation sequencing technieken, ruimtelijke resolutie in de ontwikkelingsbiologie zou kunnen worden uitgebreid. Hedendaagse studies zijn gericht op het ontleden van weefsels tot gespecialiseerde celtypes, zo niet eencellige niveau1,2,3,4. Daartoe is in de afgelopen vijftig jaar een overvloed aan verschillende methoden bedacht (zie figuur 1A)5,6,7,8,9,11,11,12,13,14,15.
Veel instrumenten in de plantenwetenschap zijn aanpassingen van technieken die werden pionier in dierlijk onderzoek. Dit is niet het geval voor de methode die we hier in detail introduceren. In 2005, uitgerust met een sterke achtergrond in eiwitvertaling, ging het Bailey-Serres Lab op zoek naar ribosomale eiwitten voor latere affiniteitszuivering16. Zo konden ze tijdrovende en arbeidsintensieve polysome profilering vermijden, die is gebaseerd op ultracentrifugatie met een sacharosegradiënt en werd gebruikt om het vertalen van ribosomen te beoordelen sinds de jaren 196017,18. De methode is sindsdien aangeduid als translationele ribosoom affiniteit zuivering (TRAP)16. Na succesvolle translationoomstudies in planten, hebben Heiman et al. TRAP voor dieren19 en anderen de toepassing ervan uitgebreid tot gist20, Drosophila21, Xenopus22 en zebrafish23,24.
Hoewel genetische modificatie van het modelsysteem een voorwaarde is voor TRAP, dat de toepassing ervan beperkt tot soorten die vatbaar zijn voor genetische transformatie, kan men dit bezwaar tegelijkertijd benutten om subgroepen van cellen aan te pakken die van bijzonder belang zijn en anderszins uiterst moeilijk te isoleren van het intacte weefsel/orgaan25 (bijvoorbeeld sterk vertakte dendritische cellen in een muishersenof schimmelhyphae in besmet plantaardig weefsel). In planten worden alle cellen op hun plaats gehouden via celwanden die de basis vormen van het hydrostatische skelet26. Om een plantencel uit deze matrix te bevrijden, hebben wetenschappers de cel fysiek uit zijn omringende weefsel gesneden door middel van lasercapture microdissectie (LCM)27 of enzymatische spijsvertering van de celwandenuitgevoerd 28. Onder de laatste cellen, de zogenaamde protoplasten, is de populatie van belang fluorescerend gelabeld en kan worden gescheiden via fluorescentie-geactiveerde celsortering (FACS)7. LCM vereist meestal een monster te worden vastgesteld en ingebed in was, die uiteindelijk verslechtert de kwaliteit van haar RNA29. FACS-gebaseerde methoden leveren rna van hoge kwaliteit op, maar het proces van protoplasting zelf introduceert verschillen in genexpressie30 en weefsels met gemodificeerde en dikke secundaire celwanden zijn notoir moeilijk te behandelen. Bovendien wordt aangenomen dat veel ontwikkelingsprocessen in installaties afhankelijk zijn van mechanisch overgebrachte signalen en daarom is de integriteit van de celwand van het grootste belang31. Twee methoden, die een snelkoppeling gebruiken om celisolatie te omzeilen door te werken op het niveau van nucleii, zijn fluorescentie-geactiveerde nucleaire sortering (FANS) en isolatie van kernen die zijn getagd in specifieke celtypen (INTACT). Net als in TRAP gebruiken ze celtypespecifieke promotors om kernen te markeren, die vervolgens verrijkt worden via sorteren of naar beneden trekken, respectievelijk8,15. Een grote uitdaging voor al deze benaderingen is om voldoende RNA-materiaal te krijgen uit subsets van cellen in een weefsel. Aangezien TRAP slechts een fractie van de cellulaire RNA’s vangt, is monsterverzameling een aanzienlijk knelpunt. Daarom zijn er vooral gevoelige protocollen voor de voorbereiding van bibliotheken nodig om gegevens van hoge kwaliteit te produceren van lage invoerhoeveelheden.
Sinds de oprichting is TRAP ofwel gebruikt in combinatie met DNA-microarrays of, aangezien de sequencingkosten de afgelopen jaren aanzienlijk daalden, RNA-seq10,32,33. Een veelheid van onderzoeksvragen is al opgehelderd zoals beoordeeld in Sablok et al.34. We zijn ervan overtuigd dat er de komende jaren meer rapporten zullen volgen, omdat de techniek zeer veelzijdig is bij het combineren van verschillende promotors om specifieke celtypen te targeten. Uiteindelijk zal dit zelfs op een ondoordringbare manier gebeuren, en kan worden gecombineerd met het indringende van de reactie van de plant op vele biotische en abiotische stressfactoren. Bovendien, waar stabiele transgene lijnen niet beschikbaar zijn, zijn harige wortelexpressiesystemen ook met succes gebruikt om TRAP uit te voeren in tomaat en medicago35,36.
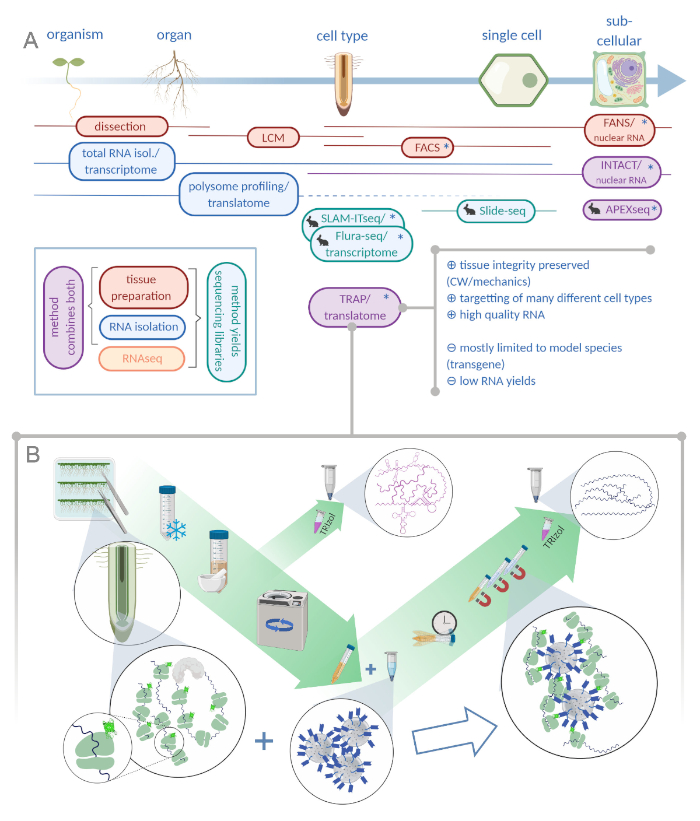
Figuur 1: Het vertalen van ribosoomaffiniteitszuivering (TRAP) vormt een aanvulling op het “omics” analyseportfolio. A. Het verhogen van de mate van analytische precisie, tot eencellige of zelfs subcellulaire resolutie kan worden bereikt door een overvloed aan methoden of combinaties daarvan. De regeling geeft een overzicht van de momenteel beschikbare gereedschappen op het gebied van planten en dieren. Weefselverzameling bij cellulaire resolutie kan worden bereikt door protocollen zoals LCM of FACS, die vervolgens worden gekoppeld aan standaard transcriptome of polysome profiling /translatome analyse. TRAP en INTACT integreren zowel weefselopname als RNA-isolatie omdat ze gebaseerd zijn op epitoop-tagging. INTACT monstert echter alleen celkernen en vormt daarom een speciaal geval van transcriptoomanalyse. Een klein konijn pictogram markeert nieuw ontwikkelde methoden in het gebied van dieren: Terwijl SLAM-ITseq en Flura-seq vertrouwen op metabole targetting van ontluikende RNAs met gemodificeerde uracil bases in cellen uitdrukken van de tolerante enzym, Slide-seq maakt gebruik van een gecoate glazen dia met DNA barcodes die positionele informatie in het cellulaire bereik. Een proximity labeling benadering wordt gevolgd in APEX-seq om RNA’s te proeven in specifieke subcellulaire compartimenten. Met name een verhoogde resolutie vereist vaak de productie van transgeen materiaal (sterretjes) en deze methoden worden dus voornamelijk gebruikt voor modelsoorten. TRAP is vooral geschikt voor plantenwetenschappelijke studies waarbij celwand (CW) of mechanische signalering en celsoorten die moeilijk te bevrijden zijn uit hun CW-matrix. B. Gedetailleerde wet-lab stappen van de TRAP procedure: Zaailingen uitdrukken GFP-gelabeld ribosomal eiwit in verschillende celtypes (bijvoorbeeld wortel endodermis) worden geteeld op petrischaaltjes gedurende zeven dagen en wortelmateriaal geoogst door snap bevriezing. Een totaal RNA controlemonster wordt verzameld uit het gehomogeniseerde ruwe extract voordat pelleting het puin via centrifugeren. Magnetische anti-GFP kralen worden toegevoegd aan het gewiste extract om immunoneerslag uit te voeren. Na incubatie en drie wasstappen wordt het polysome-geassocieerde RNA (TRAP/polysome RNA) rechtstreeks verkregen via fenol-chloroform extractie. LCM: lasercapture microdissectie, FACS/FANS: fluorescentie-geactiveerde cel/nucleaire sortering, APEX-seq: methode gebaseerd op ontworpen ascorbate peroxidase, INTACT: isolatie van kernen die zijn getagd in specifieke celtypen, SLAM-ITseq: thiol(SH)-gekoppelde alkylation voor de metabole sequencing van RNA in weefsel, Flura-seq: fluorourcila-label RNA-sequencing (Gemaakt met Biorender.com) Klik hier om een grotere versie van dit cijfer te bekijken.
Het doel van dit artikel is om een gedetailleerde beschrijving van de TRAP-methode te geven, om kritieke stappen te markeren en om richtlijnen te bieden voor een mogelijke methode voor het voorbereiden van bibliotheken.
Een generiek TRAP-experiment zal hoofdzakelijk bestaan uit de volgende stappen (zie ook figuur 1B):(1) Bereiding van plantaardig materiaal, met inbegrip van het klonen van ribosoom-tagging constructie, transgene lijnproductie en -selectie, het kweken en ophopen van zaden, sterilisatie en beplating, en stresstoepassing/behandeling (optioneel) en weefseloogsten; (2) immunozuivering, met inbegrip van weefselhomogenisatie en clearing van het ruwe extract, de wassen van de kraal en de immunozuivering, en de wasstappen; (3) RNA-extractie en kwaliteitsbeoordeling; en (4) bibliotheekvoorbereiding.
De Arabidopsis wortel is een modelsysteem om plantontwikkeling te bestuderen sinds de introductie als een model plant37,38. Hier wordt de toepassing van TRAP getoond in de context van de ontwikkeling van de wortel van planten. Bij planten is de opbouw van het gehele wortelstelsel afhankelijk van de uitvoering van dit programma en is daarom zeer belangrijk voor de overleving van het organisme39. In Arabidopsiszijn laterale wortels afkomstig van pericycleweefsel dat naast xylemvaten ligt en daarom xylempaalpericycle (XPP; zie figuur 2C)40. Sommige XPP-cellen, die zich diep in de wortel bevinden, verwerven een identiteit van de stichterscel en beginnen zich, na een lokale hormonale trigger, te vermenigvuldigen door anticlinally te zwellen en te delen41. Echter, als gevolg van de aanwezigheid van een stijve celwand matrix, dit proces oefent mechanische stress op de omliggende weefsels. Met name de bovenliggende endodermis wordt beïnvloed, omdat deze in de weg staat van de laterale wortelgroeias42,43,44. Inderdaad, de nieuw gevormde primordium zal moeten groeien door de bovenliggende endodermis cel (Figuur 2C2), terwijl cortex en opperhuid cellen zijn gewoon opzij geschoven voor de primordium om eindelijk te voorschijn45,46. Recent werk in ons lab heeft aangetoond dat de endodermis actief bijdraagt aan de proliferatie in de pericycle. Gerichte blokkering van endodermale hormonale signalering is voldoende om zelfs de allereerste divisie in de XPP-cellen47te remmen. Vandaar, pericycle-endodermis communicatie vormt een zeer vroege checkpoint voor laterale wortel ontwikkeling in Arabidopsis. Het is echter niet bekend hoe deze crosstalk wordt uitgevoerd. Om dit mysterie te ontrafelen, kozen we voor de TRAP-seq-benadering om XPP- en dodermale cellen te targeten. Om te verrijken voor cellen in het laterale wortelprogramma, bootsten we de hormonale trigger na door een auxine analoog (1-naphthaleneacetisch zuur, NAA)48exogenly toe te passen, waardoor tegelijkertijd de eerste fase van laterale wortelvorming tijdelijk kon worden opgelost.
Protocol
Representative Results
Discussion
Verificatie van rpl18-lokalisatiepatroon
Cruciaal om verkeerde interpretatie van gegevens uit een TRAP-experiment te voorkomen, is het juiste expressiepatroon van de gelabelde ribosomale subeenheid. Daarom maakt de integratie van GFP als epitoop tag aan RPL18 zeer elegant verificatie van het gewenste expressiepatroon en achtereenvolgens, pulldown van de polysome fractie uit hetzelfde weefsel. Meer invasieve benaderingen om de juiste promotorpatronen te verzekeren worden gevolgd door Jiao en Mayerowitz…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
We willen Jean-Claude Walser van het Genetic Diversity Center Zürich bedanken voor cruciaal deskundig advies in de beginfase van dit project. Het werk in het Vermeer-lab werd ondersteund door een SNF Professorship grant (PP00P3_157524) en een R’EQUIP equipment grant (316030_164086) van de Swiss National Science Foundation (SNSF) toegekend aan JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
Riferimenti
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

