Funktionell bedömning av BRCA1-varianter med CRISPR-medierade basredigerare
Summary
Personer med BRCA1-mutationer har en högre risk att utveckla cancer, vilket motiverar noggrann utvärdering av funktionen hos BRCA1-varianter. Häri beskrev vi ett protokoll för funktionell bedömning av BRCA1-varianter med CRISPR-medierade cytosinbasredigerare som möjliggör riktad C:G till T:A-konvertering i levande celler.
Abstract
Nyligen genomförda studier har undersökt riskerna med BRCA1 genmutationer med hjälp av olika funktionella bedömningsmetoder såsom fluorescerande reporter analyser, embryonala stamcellens livskraft analyser och terapeutiska läkemedelsbaserade känslighetsanalyser. Även om de har förtydligat många BRCA1-varianter, är dessa analyser som inbegriper användning av exogent uttryckta BRCA1-varianter förknippade med överuttrycksproblem och kan inte tillämpas på post-transkriptionell reglering. För att lösa dessa begränsningar har vi tidigare rapporterat en metod för funktionell analys av BRCA1-varianter via CRISPR-medierad cytosinbasredigerare som inducerar riktad nukleotidersättning i levande celler. Med den här metoden identifierade vi varianter vars funktioner förblir tvetydiga, inklusive c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T och c.4986+5G>A, och bekräftade att CRISPR-medierade basredigerare är användbara verktyg för att omklassificera varianterna av osäker betydelse i BRCA1. Här beskriver vi ett protokoll för funktionell analys av BRCA1-varianter med CRISPR-baserad cytosinbasredigerare. Detta protokoll innehåller riktlinjer för val av målplatser, funktionell analys och utvärdering av BRCA1-varianter.
Introduction
Bröstcancer typ 1 mottaglighet genen (BRCA1) är en allmänt känd tumör suppressor gen. Eftersom BRCA1-genen är relaterad till reparation av DNA-skador, skulle mutationer i denna gen leda till en större risk för cancerutveckling hos en individ1. Bröst-, äggstocks-, prostata- och bukspottskörtelcancer är kopplade till ärvda funktionsförlustmutationer (LOF) av BRCA1-genen 2. Funktionell bedömning och identifiering av BRCA1 varianter kan bidra till att förebygga och diagnostisera de olika sjukdomarna. För att ta itu med funktionen hos BRCA1-varianter har flera metoder utvecklats och använts i stor utsträckning för att undersöka patogeniciteten hos BRCA1-varianter som embryonala stamcellens livsduglighetsanalyser, fluorescerande reporteranalyser och terapeutiska läkemedelsbaserade känslighetsanalyser3,4,5,6. Även om dessa metoder har bedömt funktionen hos många BRCA1-varianter, utgör metoderna som involverar exogent uttryckta BRCA1-varianter begränsningar när det gäller överuttryck som kan påverka nedströmsreglering, gendosering och proteinvikning7. Dessutom kan dessa analyser inte utnyttjas till den posttranscriptional förordningen såsom mRNA splitsning, transkription stabilitet och effekt av oöversatta region8,9.
CRISPR-Cas9-systemet möjliggör riktad genomredigering i levande celler och organismer10. Genom en enkelriktad RNA kan Cas9 inducera dubbelsträngsbrytningar (DSB) i kromosomalt DNA vid specifika genomiska lokus för att aktivera två DNA-reparationsvägar: felbenägen nonhomologous end-joining (NHEJ) väg och felfri homologistyrd reparation (HDR) väg11. HDR är en exakt reparationsmekanism; DSB framkallas dock av Cas9 nuclease för HDR resulterar ofta i oönskade införing och borttagning (indel) mutation. Dessutom behöver den homologa donator-DNA-mallar för att reparera DNA-skador och har relativt låg effektivitet. Nyligen har Cas9 nickase (nCas9) smälts med cytidindeaminasdomäner för att rikta in sig på C:G till T:A-substitutioner, utan behov av homologa DNA-mallar och DNA-dubbelsträngsbrott12,13,14,15. Med hjälp av cytosin basredigeraren utvecklade vi en ny metod för funktionell analys av BRCA1 varianter16.
I denna studie använde vi CRISPR-medierad cytosinbasredigerare, BE314, som inducerar effektiva C:G till T:A-punktmutationer, för att genomföra den funktionella bedömningen av BRCA1-varianter och framgångsrikt identifierat funktionerna hos flera BRCA1-varianter (Figur 1).
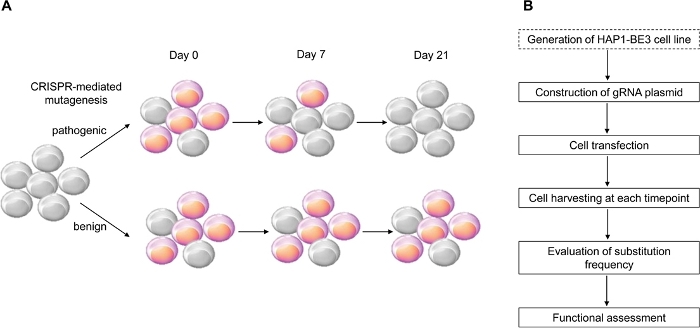
Figur 1: En översikt över arbetsflödet för funktionell utvärdering. a)Schematiskt som visar den funktionella bedömningen av BRCA1. Eftersom BRCA1:s LOF påverkar cellens livskraft, när BRCA1-mutationen är patogen, dör cellerna när passagenumret ökar. b)Stadier i den funktionella bedömningen av BRCA1. Prickad låda är valfri. Det kan ersättas av co-transfection av gRNA uttrycker och BE3 uttrycker plasmider DNA. Klicka här om du vill visa en större version av den här figuren.
Protocol
Representative Results
Discussion
Detta protokoll beskriver en enkel metod för funktionella bedömningar av BRCA1 varianter med CRISPR-meditated cytosin bas editor. Protokollet beskriver metoder för utformning av gRNAs vid målloka och konstruktion av de plasmid DNAs från vilka de uttrycks. Cytosinbasredigerare inducerar nukleotidkonvertering i ett aktivt fönster (vid BE3, nukleotider 4-8 i PAM-distala änden av gRNA-målsekvenserna). Forskaren bör noggrant välja målsekvenser eftersom alla cytosiner i aktivt fönster kan ersättas med tym…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Detta arbete stöddes av Koreas nationella forskningsstiftelse (bidrag 2017M3A9B4062419, 2019R1F1A1057637 och 2018R1A5A2020732 till Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
Riferimenti
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Ricerca sul cancro. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

